ICLR 2025 | PepTune: 通过多目标引导的离散扩散实现治疗性肽段的从头设计
PepTune 是来自 Chatterjee 实验室的一项创新成果,提出了基于多目标离散扩散模型结合蒙特卡洛树搜索(MCTS)的治疗肽段 SMILES 生成与优化方法。该模型支持非天然氨基酸和环状肽段设计,利用基于肽键的掩码调度与全局无效损失,有效提高肽段结构有效性,避免生成无效序列。通过 MCTS 引导实现多目标(结合亲和力、溶解度、膜通透性、溶血性、抗污染性)优化,无需梯度估计,克服了多目标冲突和数据稀疏问题。
PepTune 成功设计出短链 GLP-1R 结合肽段,其结合能力优于经典激动剂赛美特肽和利拉鲁肽,降低了合成难度和免疫风险。同时,实现了双靶点 TfR 和 GLAST 结合肽段的设计,为神经疾病药物跨越血脑屏障提供新思路。该方法无需靶点三维结构,适用于构象多样且动态的蛋白靶点。

获取详情及资源:
- 论文:https://doi.org/10.48550/arXiv.2412.17780
- 代码: https://huggingface.co/spaces/ChatterjeeLab/SMILES2PEPTIDE (肽段过滤、分析和可视化工具) | https://huggingface.co/ChatterjeeLab/PepTune (PepTune 代码库)
- 查一下 这个通讯 介绍一下课题组 这个组以后会经常碰到!!!简单介绍一两句 然后放一个课题组链接
0 摘要
PepTune 是一种基于多目标离散扩散模型的系统,能够同时生成并优化治疗性肽段的 SMILES 序列。该方法基于掩码离散语言模型(Masked Discrete Language Model,MDLM)框架,利用依赖于键合关系的掩码策略和基于惩罚的目标函数,确保生成的肽段结构有效且合理。为了引导扩散过程,提出了基于蒙特卡洛树搜索(Monte Carlo Tree Search,MCTS)的策略,通过平衡探索与利用,逐步优化得到帕累托最优的序列。MCTS 结合了基于分类器的奖励机制与搜索树的扩展,成功克服了梯度估计的困难和数据稀疏性问题。
通过 PepTune,可生成多样化的化学修饰肽段,且同时针对多个治疗相关的性质进行优化,包括靶点结合亲和力、膜通透性、溶解度、溶血性和抗污染能力,适用于多种疾病相关靶点。整体结果表明,MCTS 引导的掩码离散扩散方法在离散状态空间的多目标序列设计中展现了强大且模块化的潜力。
1 引言
肽段作为一种治疗手段具有独特优势,其低细胞毒性和结构灵活性使得肽段能够结合多样的结合位点,而无需依赖稳定的结合口袋,因此特别适合靶向结构多样的蛋白表面。然而,仅由20种天然氨基酸组成的肽段存在一定局限性,如易受酶降解和膜通透性较低。为克服这些问题,非天然氨基酸(nAAs)通过对肽骨架和侧链进行多样化的化学修饰被引入肽段,以增强其治疗性能。尽管肽类药物开发取得了进展,但对庞大化学修饰肽段空间的探索依然是主要瓶颈,这也推动了生成式深度学习模型的发展,这些模型能够有效学习临床相关肽段的空间,并根据多种治疗特性进行全新肽段的采样。
生成扩散模型被视为全新结合体设计的先进技术,但它们通常依赖于靶蛋白稳定的三级结构,限制了对无序及动态靶点肽段设计,而这些靶点在众多疾病中占有重要地位。基于肽段序列的生成语言模型在无序及结构多样的靶点上取得了显著成功,但因仅使用20种天然氨基酸,限制了其对化学修饰或环状肽段空间的采样能力。此外,离散生成模型在多目标引导的生成与优化方面仍面临显著挑战。已有的基于分类器或无分类器的引导策略,虽被尝试用于引导离散扩散模型实现特定性质的优化,但常因目标冲突、梯度估计难题及高质量数据稀缺而效果有限。
本研究提出了 PepTune,这是首个面向肽段 SMILES 全新生成的多目标引导离散扩散模型。其主要贡献包括:
首先,设计了用于肽段 SMILES 生成的掩码扩散语言模型,支持非天然氨基酸和环状修饰的肽段生成。其次,提出了基于键合关系的掩码调度策略,并推导了其负对数证据下界(NELBO)及反向后验,有效提升生成肽段结构的合理性。第三,引入了基于肽段 SMILES 过滤器的全局序列无效损失,能将离散序列中的惩罚高效传递到连续概率分布中。第四,构建了基于蒙特卡洛树搜索(MCTS)的多目标分类器引导框架,无需梯度估计或重新训练即可迭代优化治疗相关性质上的帕累托最优肽段序列。最后,训练了一套用于预测结合亲和力、膜通透性、溶解度、溶血性及抗污染性的分类器和回归器,形成全面的性质预测工具包。
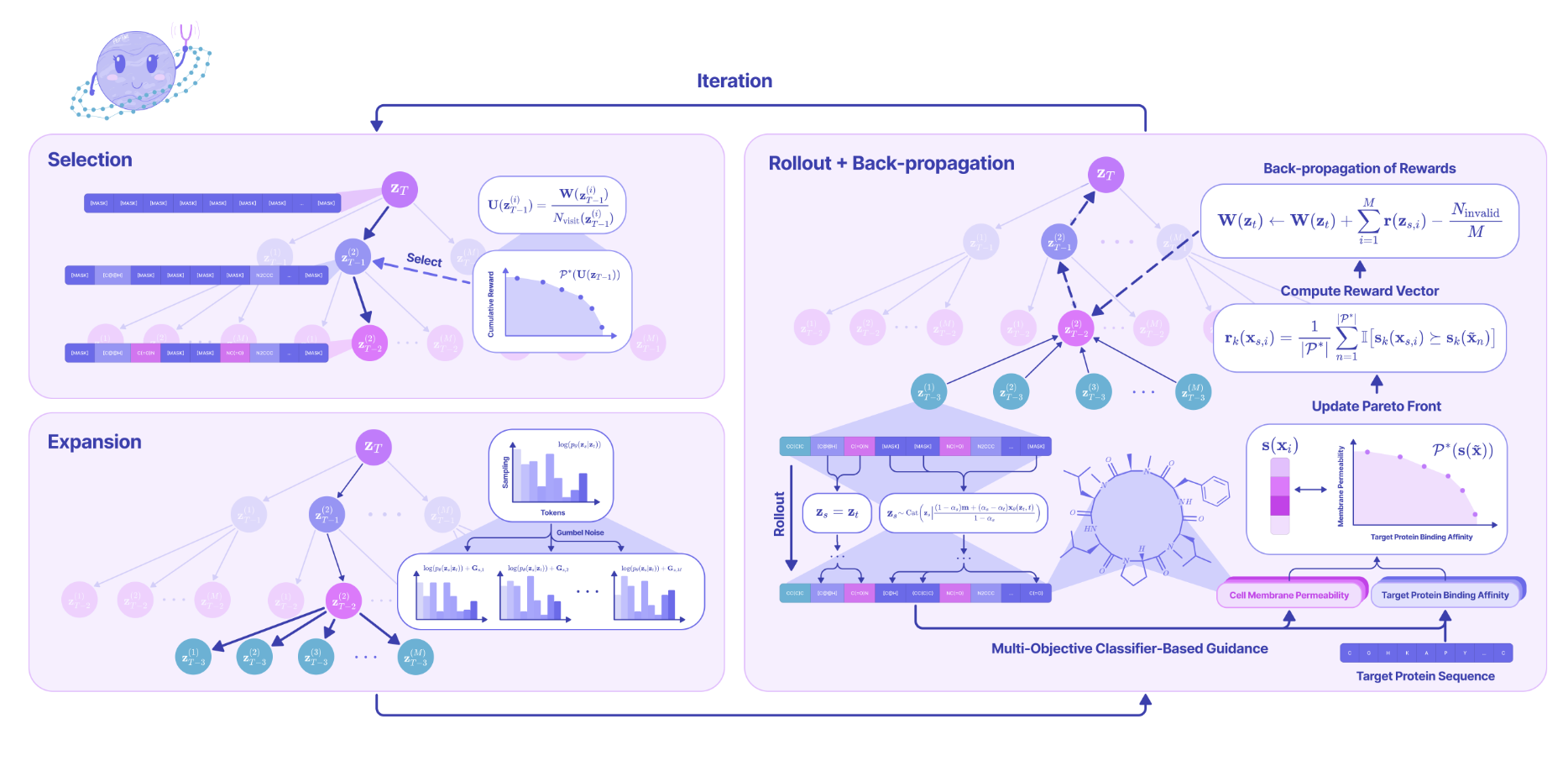
2 结果
2.1 用于肽段 SMILES 生成的掩码离散扩散模型
该方法基于掩码扩散语言模型(MDLM)框架,无条件地学习从含有 [MASK] 标记的序列中重建完整序列。在此基础上,扩展了以往的状态依赖掩码策略,设计了基于键合关系的掩码调度 αt(x0),使得肽键的掩码速率低于非肽键,从而在前向扩散过程中更缓慢地掩盖肽键。通过推导带参数的反向后验分布
公式如下:
除此之外,发现采样过程中的细微语法错误会导致完全无效的 SMILES 序列,因此引入了基于肽段 SMILES 有效性过滤器的无效损失项
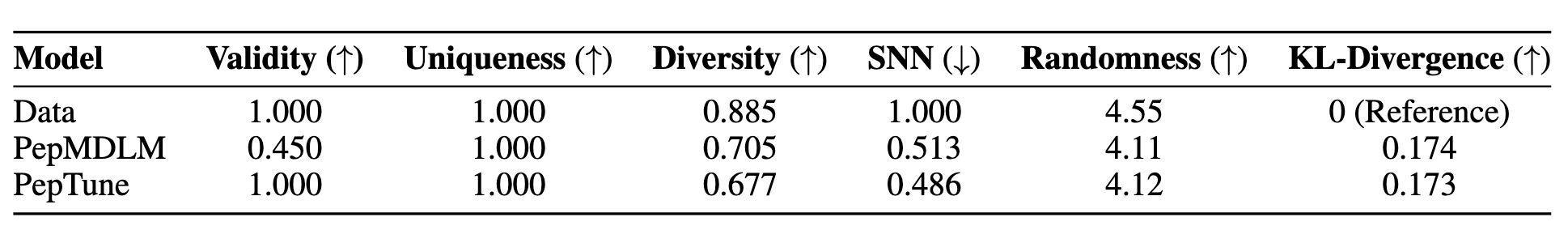
优化后的无条件 MDLM(PepMDLM)相较于宏环肽段的自回归生成器 HELM-GPT,展现出更高的独特性和多样性,同时保持较低的 SNN(序列相似性)分数,显示了在有效肽段 SMILES 子空间内的全面搜索能力。此外,PepMDLM 生成的肽段在非天然氨基酸(nAA)频率上高于实验验证的肽段,特别是在膜通透性和结合亲和力方面,表现出独特的设计能力,支持含环状和化学修饰肽段的全新设计,极大扩展了治疗肽段的搜索空间,超越了基于天然氨基酸的传统生成模型。
2.2 离散扩散中针对治疗性质的多目标引导
提出了基于分类器的多目标引导策略,结合蒙特卡洛树搜索(MCTS)生成一组非支配肽段 SMILES 序列
PepTune 的多目标引导框架流程如下:每轮迭代从全掩码序列
每个子节点通过贪婪 Gumbel-max 采样去掩码得到完整序列
在性质预测方面,训练了结合亲和力和膜通透性的回归模型,以及针对溶解度、溶血性和抗污染性的二分类模型,均专门针对肽段 SMILES 数据进行训练。尽管选择过程倾向于高奖励路径,生成的肽段集合依旧保持与无条件 MDLM 生成序列及训练集相似的独特性和多样性。此外,在仅经过20次 MCTS 迭代后,生成肽段的有效率持续达到100%,证明了分类器奖励与无效惩罚的反向传播在提升生成质量上的有效性。
2.3 肽段结合剂多目标生成案例研究
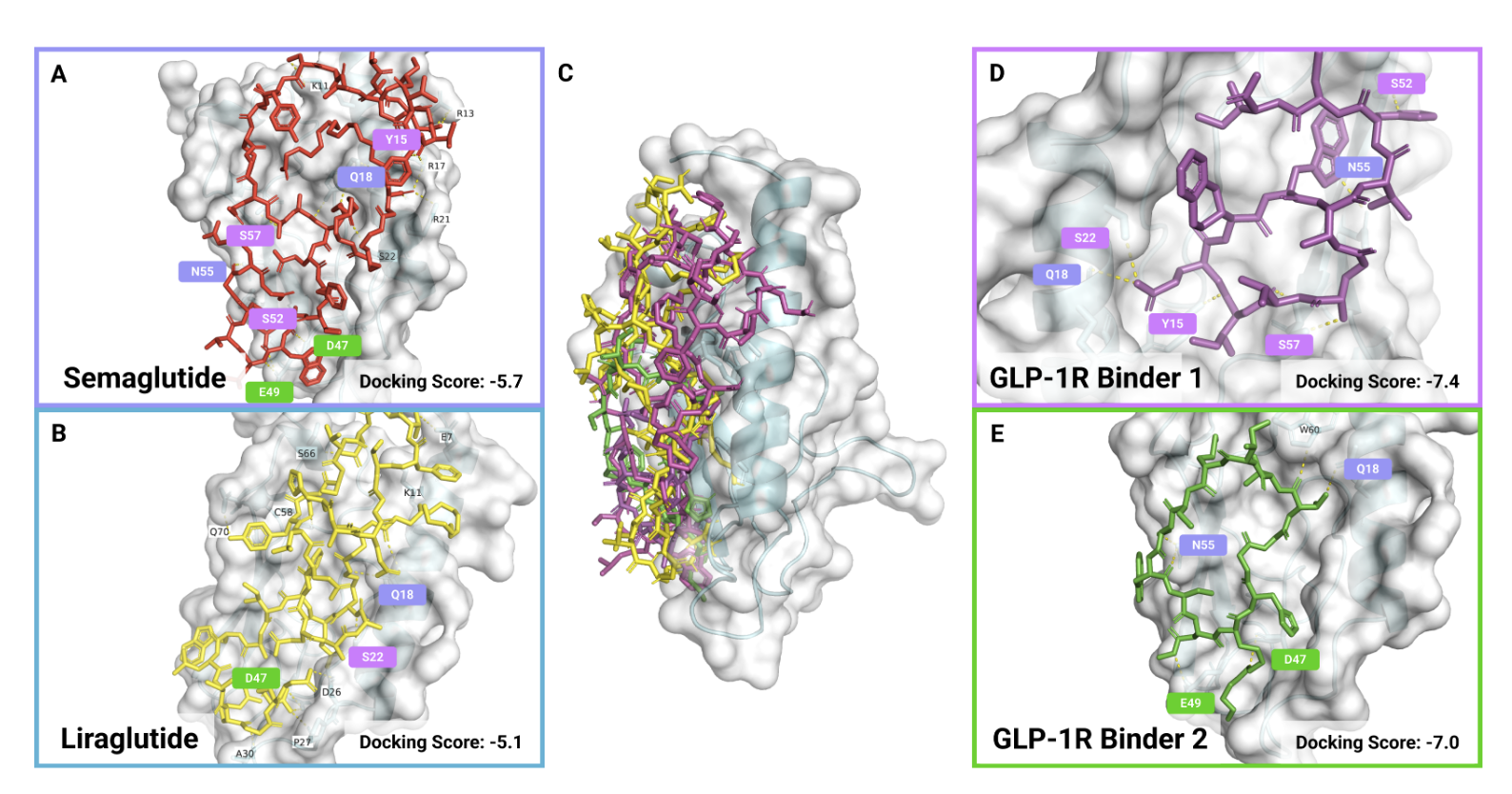
鉴于胰高血糖素样肽-1 受体(GLP-1R)肽类激动剂在治疗2型糖尿病和肥胖症方面的显著进展,本研究将基于 PepTune 生成的针对 GLP-1R 结合亲和力条件的肽段与现有的明星药物赛美特肽(semaglutide)和利拉鲁肽(liraglutide)进行了比较。赛美特肽和利拉鲁肽均由超过30个氨基酸组成,通过高精度模拟天然 GLP-1 激素与 GLP-1R 激活口袋的结合发挥作用。相比之下,较短链的激动剂或拮抗剂在治疗胰岛素相关疾病方面具有多重优势,包括合成成本和复杂度降低、免疫原性减弱以及更快的组织穿透速度。
因此,研究旨在设计出能够以较高结合亲和力结合 GLP-1R 的短链肽段。首先生成了一组基于结合亲和力、溶解度、溶血性和抗污染性等条件的肽段结合剂。从帕累托非支配集筛选出结合亲和力最高的肽段,随后进行了分子对接实验。两个最佳候选肽段的对接得分分别为 -7.4 kcal/mol 和 -7.0 kcal/mol,优于现有药物,并且结合位点与赛美特肽和利拉鲁肽在天然激素 GLP-1 的结合位点有重叠。这表明,PepTune 生成的肽段有潜力作为有效的 GLP-1R 激动剂或拮抗剂。
2.4 双靶点结合肽段案例研究
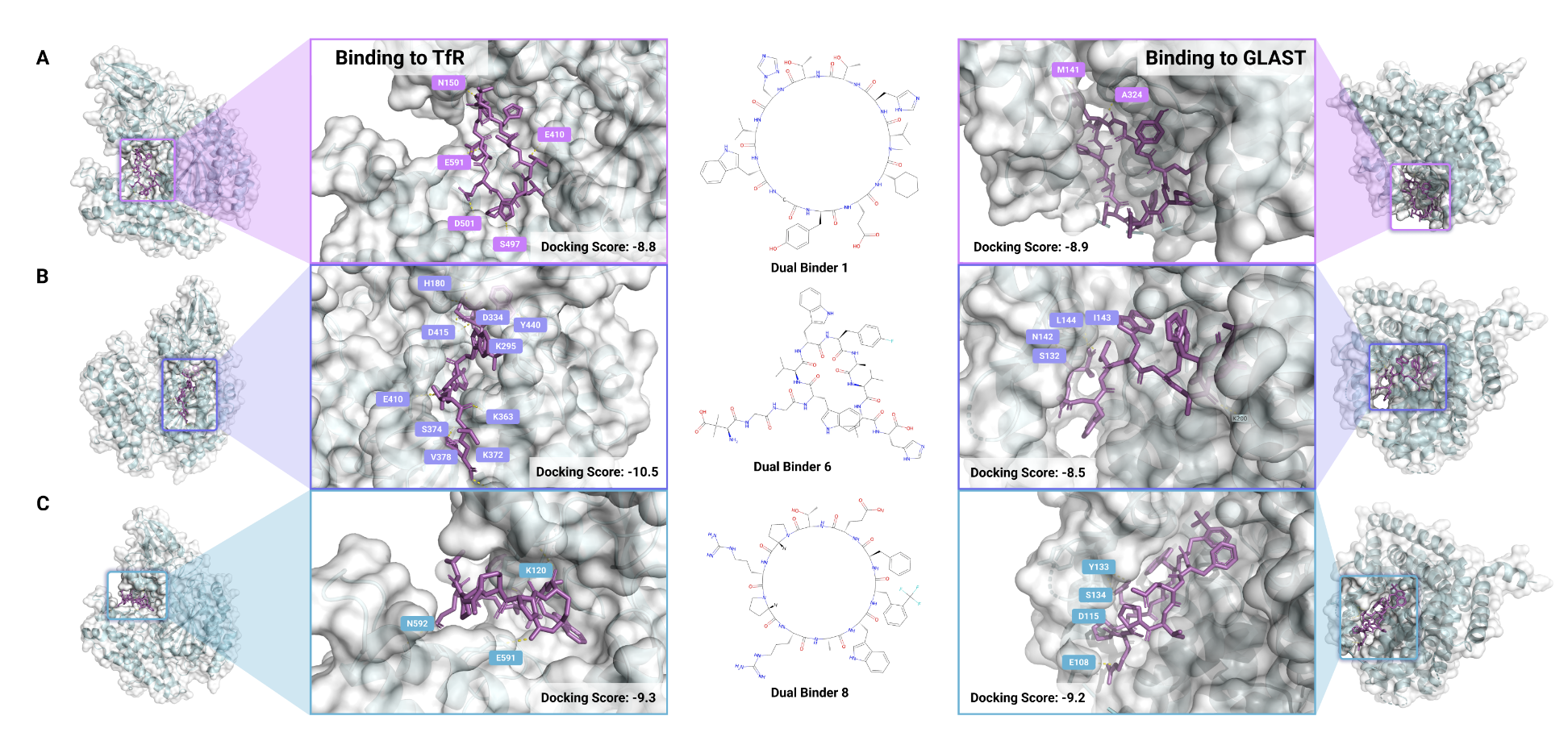
多靶点药物开发在癌症治疗和神经疾病药物输送等领域备受关注,因其能够执行多种功能,如跨越血脑屏障(BBB)、穿透目标细胞及抑制蛋白-蛋白相互作用。为了验证 PepTune 在多靶点引导上的能力,生成了针对血清转铁蛋白受体(TfR)和谷氨酸-天冬氨酸转运蛋白(GLAST)的双特异性肽段结合剂。GLAST 富集于脑内星形胶质细胞表面,这种设计有望实现通过 TfR 介导的 BBB 跨越及 GLAST 介导的星形胶质细胞摄取,从而为包括亚历山大病、阿尔茨海默病、帕金森病、亨廷顿病、多发性硬化及多种精神疾病等神经系统疾病的静脉给药提供新策略。
生成了100个基于五项性质(TfR结合亲和力、GLAST结合亲和力、溶解度、溶血性、抗污染性)条件的肽段结合剂。所有性质指标在迭代过程中均得到提升,最终溶解度、溶血性和抗污染性评分均优于仅以 TfR 结合亲和力为条件的肽段,表明多靶点引导策略有效避免了重要性质间的权衡。选取了7个结合剂进行分子对接,所有对接得分均 ≤ -7.5 kcal/mol,其中最佳结合剂在 TfR 和 GLAST 上分别获得了 -10.5 kcal/mol 和 -9.2 kcal/mol 的优异得分。这些肽段展示了多样的二级结构,表现出良好的溶解性和低溶血概率。结合位点和极性相互作用在不同肽段间存在差异,显示出 PepTune 能够独立于特定结合基序,发现多样且最优的双靶点结合肽段。
3 结论
本研究提出了 PepTune,这是一种在离散序列空间中直接进行多目标优化的生成框架。PepTune 创新性地将蒙特卡洛树搜索(MCTS)应用于扩散模型,生成针对结合亲和力、溶解度、膜通透性和溶血性等治疗性质优化的帕累托最优肽段 SMILES 序列。该方法在离散状态空间内实现引导,克服了梯度估计和连续-离散空间映射的限制。最关键的是,PepTune 无需依赖靶点蛋白的三维结构,区别于近期的结合剂设计方法,使其能够为构象多样的蛋白设计肽段,并针对局部几何交互以外的性质进行优化。