NMI 2024 | ActFound: 利用成对元学习的生物活性基础模型
今天介绍发表在 《Nature Machine Intelligence》 上的ActFound研究。这项工作不仅提出了一个新的生物活性基础模型,更在方法论层面回应了药物研发长期面临的核心困境:实验数据稀缺、不同assay间难以对齐,以及如何在计算与实验之间建立有效桥梁。研究者通过成对学习(pairwise learning)避免了不同实验间数值不兼容的问题,使模型能够学习到跨实验的相对活性差异;再结合元学习(meta-learning),赋予模型在小样本场景下快速适应新实验的能力。这种思路的高明之处在于,它不再依赖于某个单一实验的绝对预测,而是从跨任务的一致性中提炼出普适模式,从而更接近“生物活性知识图谱”的抽象层面。
更值得强调的是,ActFound的意义超越了技术细节。它为机器学习在药物发现中的角色提出了新的可能:不只是辅助预测,而是作为底层通用模型,像语言模型在自然语言处理中的地位一样,成为生物活性预测的“基础设施”。其在FEP替代、跨域预测以及癌症药物反应零样本预测中的表现,说明这种范式不仅能带来更高的计算效率,还能触及实验设计和候选筛选的上游环节。换句话说,ActFound不仅是一个模型,更是推动药物研发从数据孤岛走向统一表示、从个案方法走向通用平台的重要一步。

获取详情及资源:
0 摘要
化合物的生物活性在药物研发与发现中具有重要作用。然而,现有机器学习方法在生物活性预测中的泛化能力较差,主要原因在于每个实验(assay)中化合物数量有限,以及不同实验间测量方式不兼容。作者提出了ActFound,一个生物活性基础模型,基于ChEMBL数据库中160万条实验测得的生物活性数据和35,644个实验进行训练。
ActFound的核心思想是利用成对学习(pairwise learning),学习同一实验中两个化合物之间的相对生物活性差异,从而规避实验间的不兼容问题。同时,模型通过元学习(meta-learning)在所有实验上联合优化。在六个真实的生物活性数据集上,ActFound不仅展现了高精度的域内预测,还表现出在不同实验类型和分子骨架间的强泛化能力。
此外,研究表明ActFound可作为领先的物理计算工具FEP+(OPLS4)的有效替代方案,仅需少量数据进行微调,即能获得可比的性能。结果显示,ActFound有望成为用于化合物生物活性预测的基础模型,为基于机器学习的药物研发与发现开辟新的途径。
1 引言
评估化合物的生物活性是药物发现与开发的核心环节。生物活性涵盖了化合物的多种属性,如与靶点的相互作用、对生物系统的影响以及治疗效果。它已被广泛应用于药物研发的不同阶段,包括先导化合物发现、先导优化以及药物再利用。生物活性预测的目标是在特定实验(assay)中预测化合物的活性,从而帮助研究人员在大量候选分子中快速筛选出理想化合物,并减少对昂贵且耗时的湿实验的依赖。
传统的基于物理学的计算方法依赖难以获取的三维蛋白结构,并在精度与计算成本之间存在天然权衡。例如,主流的 自由能微扰(FEP) 方法可以提供高精度预测,并已成功应用于多个真实药物发现项目,但其计算资源消耗极大,难以适用于大规模应用。过去十年,深度学习在生物活性预测中展现出巨大潜力。然而,现有方法存在两大局限:首先,由于实验成本高,每个实验中仅有少量化合物(ChEMBL中平均12.7个),不足以训练实验特定模型;其次,即便利用迁移学习、多任务学习或元学习等方法联合训练,不同实验在单位、数值范围和测量方式上常常不兼容,导致迁移失效,削弱预测性能。
为克服这些限制,研究者引入基础模型。这类模型在大规模数据上预训练,可作为多种下游任务的通用起点,尤其适合标注数据昂贵的生物医学领域。该研究提出的ActFound正是一种生物活性基础模型,结合了元学习(meta-learning)与成对学习(pairwise learning)。其中,元学习用于在多样化的大量实验上联合训练,并为仅有少量数据的新实验提供良好的初始化;成对学习则通过学习同一实验中两个化合物的相对活性差异,绕过实验间的不兼容问题,从而得到可泛化于所有实验类型的统一模型。值得一提的是,这是首次在生物活性预测中结合元学习与成对学习。
实验结果表明,ActFound在多个任务中表现优异:在六个真实数据集上的域内预测中,显著优于九种对比方法,展现出几乎覆盖所有实验类型的能力;在跨域预测中,同样超过最新方法,体现出良好的泛化性。此外,在先导优化场景下,ActFound表现出可替代FEP的潜力,仅需少量数据微调即可达到相当性能。进一步地,利用ActFound预训练的癌症药物反应预测模型在零样本药物敏感性预测任务中也展现了可观的效果。
综上,ActFound通过成对元学习构建,作为一个生物活性基础模型,不仅能够解决实验数据稀缺与不兼容的问题,还可在药物研发与发现的多种应用中发挥作用。
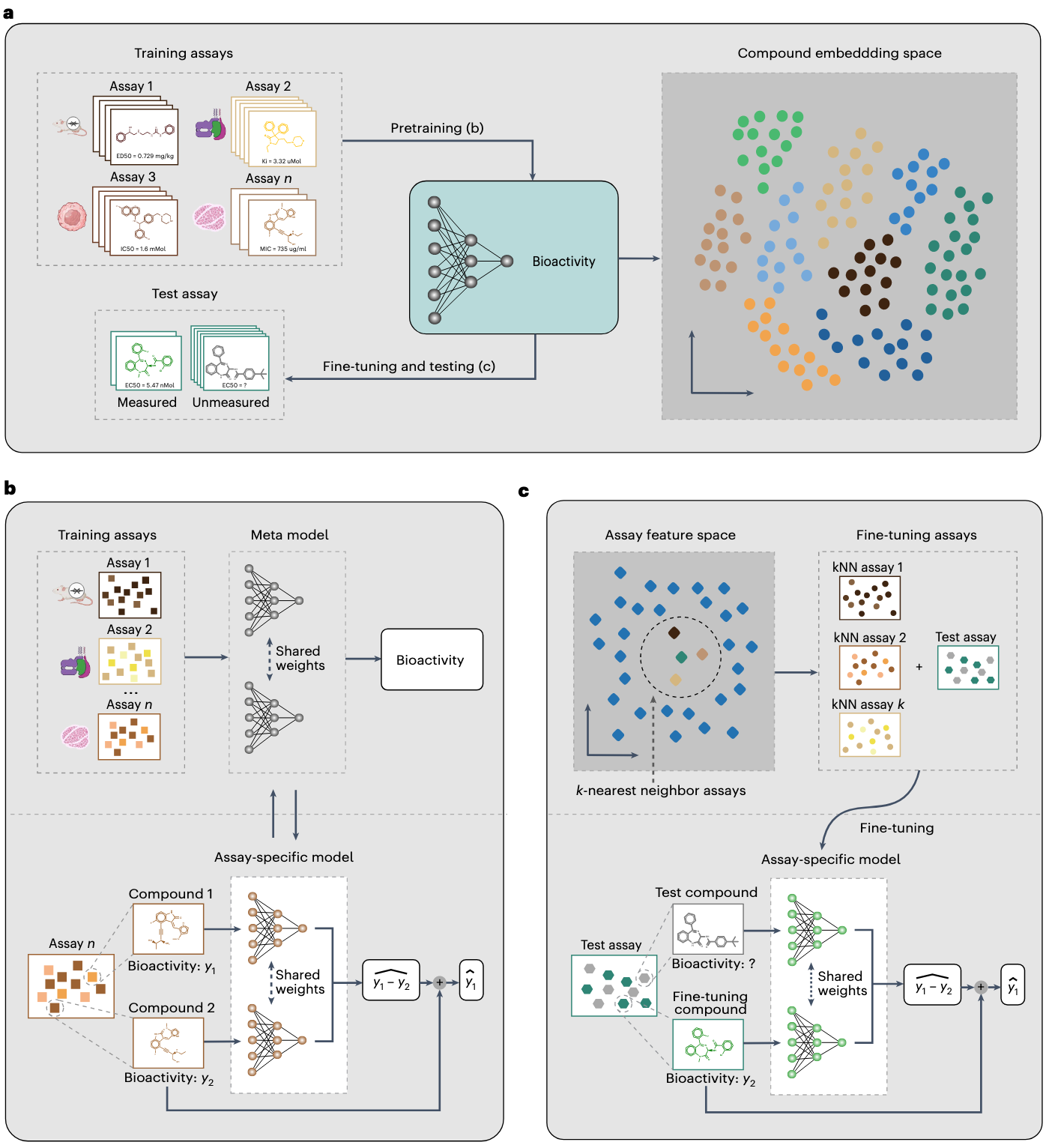
图1 | ActFound 概览 a, 总体架构。ActFound是一个基于大规模实验预训练的生物活性基础模型。在预训练后,ActFound只需少量已测化合物即可在目标实验上进行微调,并预测该实验中其他未测化合物的生物活性值。预训练阶段使ActFound能够将来自不同实验的化合物投射到统一的嵌入空间。b, 预训练流程。ActFound采用成对元学习作为预训练策略。通过孪生网络学习同一实验中两种化合物的相对生物活性差异,从而解决实验间的不兼容问题。最终的活性值可由预测的相对差异计算得到。c, 微调流程。ActFound应用基于kNN的微调策略,选择k个最邻近实验与目标实验联合微调,使预训练模型能够快速适应目标实验,从而提升泛化能力。(图使用BioRender.com绘制)
2 结果
2.1 ActFound 概览
ActFound是一个生物活性基础模型,旨在解决生物活性预测中的两大挑战:单个实验中标注数据有限以及不同实验之间测量方式不兼容。该模型能够高效利用大量实验与化合物数据,并通过元学习与成对学习提供预训练的生物活性模型(图1a)。
在预训练阶段,ActFound的参数通过双层优化过程在多样化实验上交互更新:内循环用于特定实验的微调,外循环则根据微调后模型的表现更新全局参数(方法见文中)。这一机制使ActFound对来自不同实验的更新更为敏感,并能在后续快速适应新的实验。
模型骨干为孪生网络,用于计算两种化合物之间的相对生物活性差异。通过关注相对活性,而非绝对值,模型能够规避实验间数值不兼容的问题。最终的生物活性值可由预测的相对差异重建(图1b)。在实际应用中,ActFound只需利用少量已测化合物进行微调,即可预测该实验中未测化合物的生物活性(图1c)。
实验表明,ActFound在域内生物活性预测上具有高准确性,并在跨域预测中展现出强大的泛化能力。
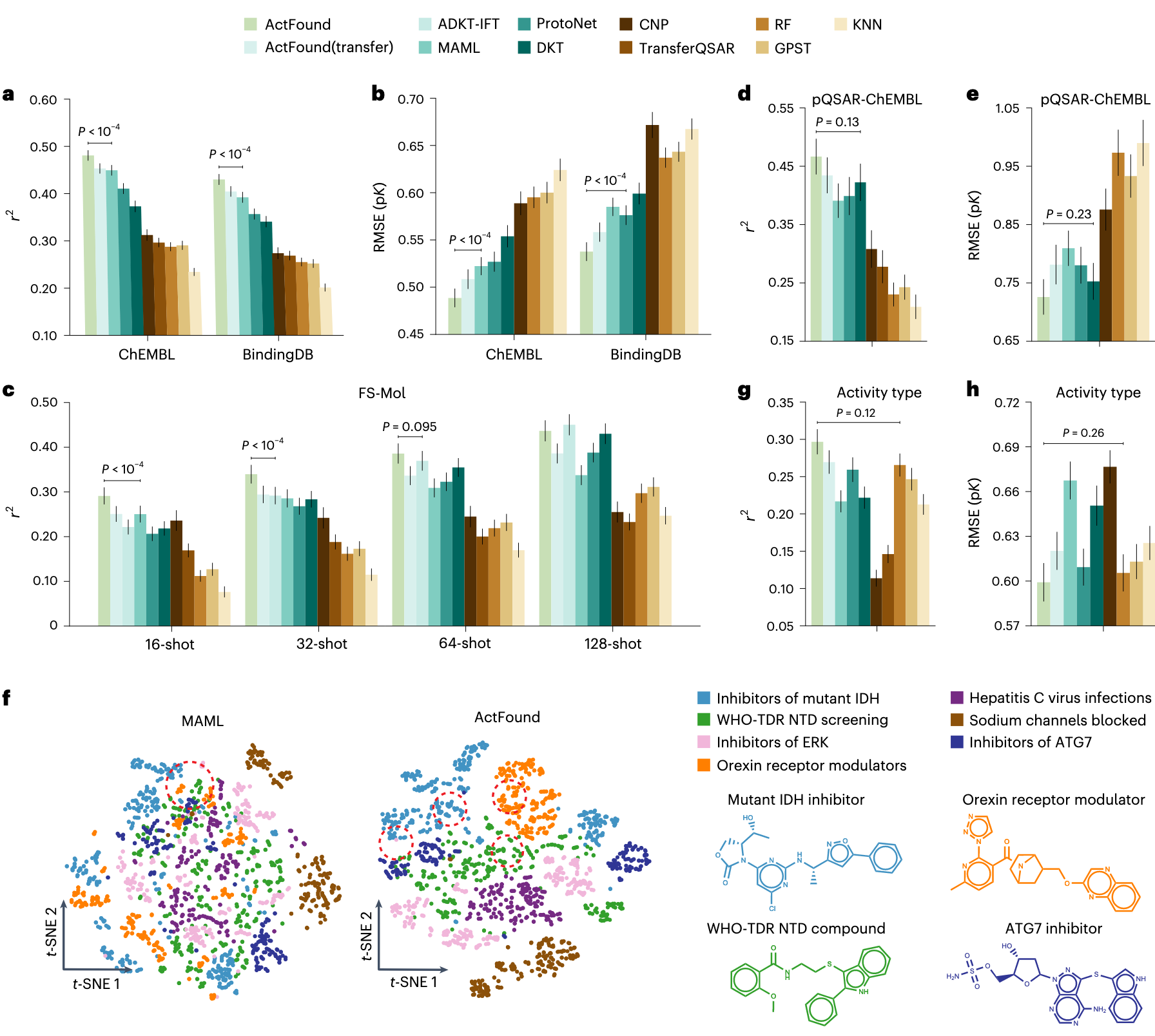
图2 | 域内生物活性预测的评估 a,b, 条形图比较了ActFound与对比方法在ChEMBL和BindingDB上的16-shot预测性能,以
2.2 精确的域内生物活性预测
研究首先在ChEMBL与BindingDB的多类实验上评估了ActFound的域内预测性能。这些实验涵盖了药物发现中的多种环节,涉及化合物的结合、功能、ADMET等性质。按照既往研究的设定,采用16-shot方案:在每个实验中用16个化合物进行微调,其余化合物用于测试。结果显示,ActFound在两个数据集上均显著优于所有对比方法,不论是
进一步分析表明:
- ActFound大幅超越传统元学习方法MAML与ProtoNet,验证了成对学习捕捉相对活性差异的有效性。
- 与替换为迁移学习的变体相比,ActFound表现更佳,证明了元学习在多实验联合训练中构建基础模型的优势。尤其在ChEMBL数据集上提升更明显,这与其包含35,644个实验(远超BindingDB的15,225个)有关,能够更好发挥元学习在快速适应新实验上的优势。
- 消融实验发现,去掉kNN-MAML模块会显著降低性能,说明利用邻近实验辅助微调是有效的。
在FS-Mol数据集(ChEMBL子集,仅保留浓度单位实验)上,ActFound在低资源条件(16-shot和32-shot)下优势最为显著,在128-shot时表现与对比方法相当,这归因于元学习在数据有限时更具优势。进一步,在更具挑战性的pQSAR-ChEMBL划分中(训练与测试化合物结构差异显著),ActFound依然表现最佳(图2d,e),说明其能够泛化至未知分子骨架。
对化合物嵌入的可视化分析(图2f)表明,ActFound相比MAML产生了更清晰的聚类模式,符合同一实验分子应结构相似的预期,显示了其更高质量的表征能力。值得注意的是,当实验未包含百分比单位时,ActFound的性能相对较低(图2g,h),但仍优于其他迁移学习与元学习方法,表明其在不同单位间仍具有较强的跨域泛化性。
综上,ActFound在域内任务中的优异表现证明了成对元学习构建生物活性基础模型的有效性,并为进一步探索跨域场景下的性能奠定了基础。
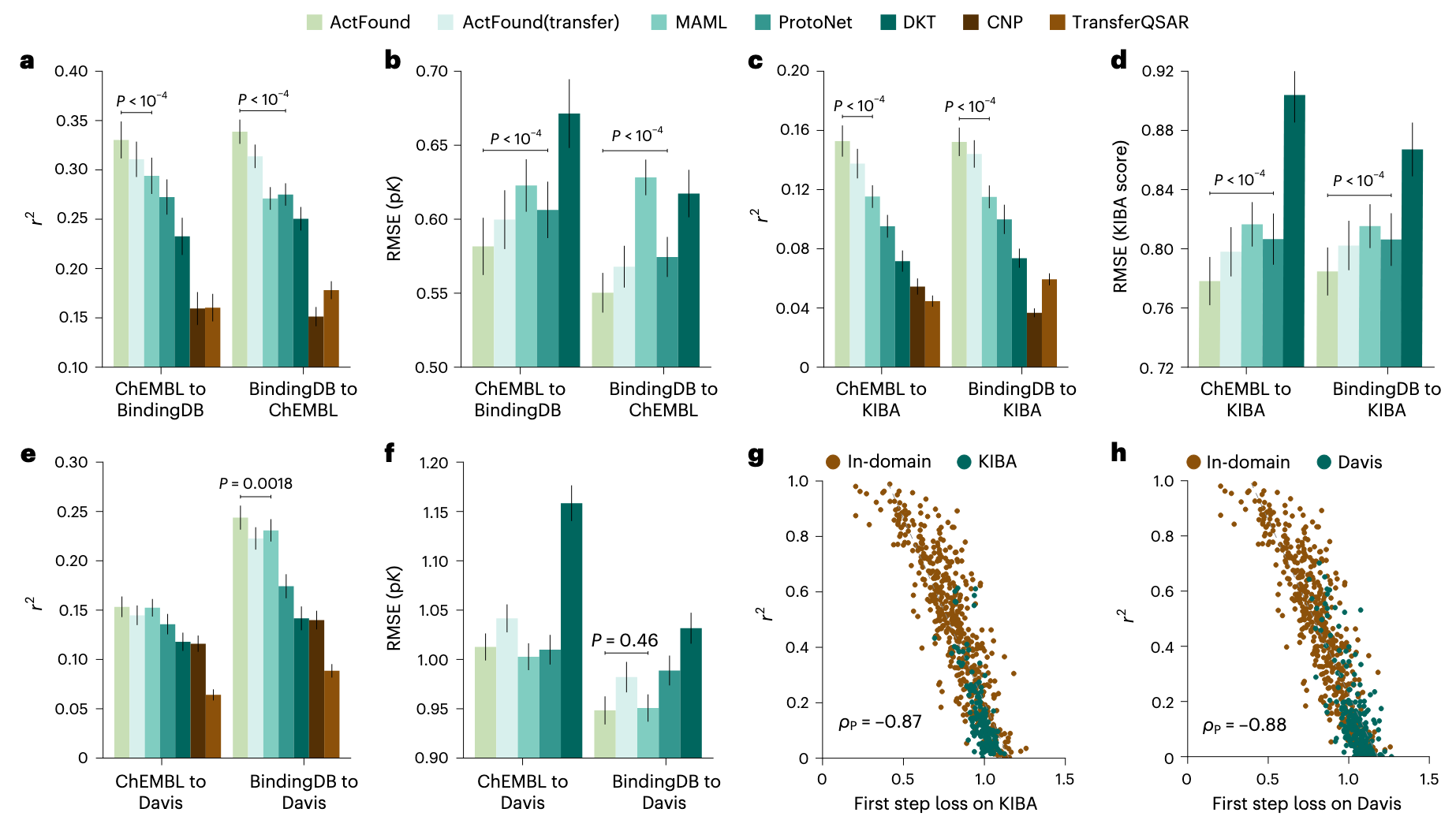
图3 | 跨域生物活性预测的评估 a,b, ChEMBL与BindingDB间跨域预测的比较,以
2.3 跨域泛化性能的提升
基础模型的一大优势在于其能够在不同领域和数据集间实现预测泛化。基于ActFound在未知分子骨架和单位上的良好表现,研究进一步系统评估了其跨域预测能力:即在一个领域的实验上训练模型,再在另一个领域的实验上进行微调(图3)。结果表明,与域内预测相比,跨域预测性能略有下降,说明该设定更具挑战性。然而,ActFound在所有数据集与指标上均优于对比方法。具体而言,ChEMBL→BindingDB时,
考虑到BindingDB与ChEMBL来源多样,可能存在重叠实验带来的数据泄漏,研究还引入了两个独立的激酶抑制剂数据集KIBA与Davis进行验证。结果显示,这一设定下性能显著下降,进一步体现其挑战性。但ActFound在两者上依然获得最佳表现(图3c–f),证明了成对学习与元学习的优势。此外,ActFound相较MAML在KIBA上的提升更大,而KIBA的测量单位在预训练实验中未出现,说明ActFound在应对全新测量单位时仍具较强泛化能力。
在与最新的药物-靶点亲和力(DTA)方法对比时,ActFound在BindingDB(IC50)→KIBA和BindingDB(IC50)→Davis实验中优于SSM-DTA、GraphDTA以及DeepPurpose中的四种方法,且未使用靶蛋白序列作为输入,表明其有潜力作为DTA预测的基础模型。进一步在 活性悬崖(activity cliff) 数据集中,尽管测试化合物与微调化合物结构相似但活性差异极大,ActFound的平均RMSE为1.447 pK,依然领先于对比方法,为药物发现提供了更具价值的预测线索。
值得注意的是,在跨域设定下,所有方法性能均低于域内设定,因此让终端用户了解模型在哪些测试实验中表现更好显得重要。研究发现,测试实验的性能与首次优化步骤的loss值高度相关(ρP = −0.88),小的loss意味着模型更容易适应新实验(图3g,h)。这一发现表明,用户可据此识别哪些实验能够通过ActFound获得更大提升,从而在实际应用中更高效地利用该模型。
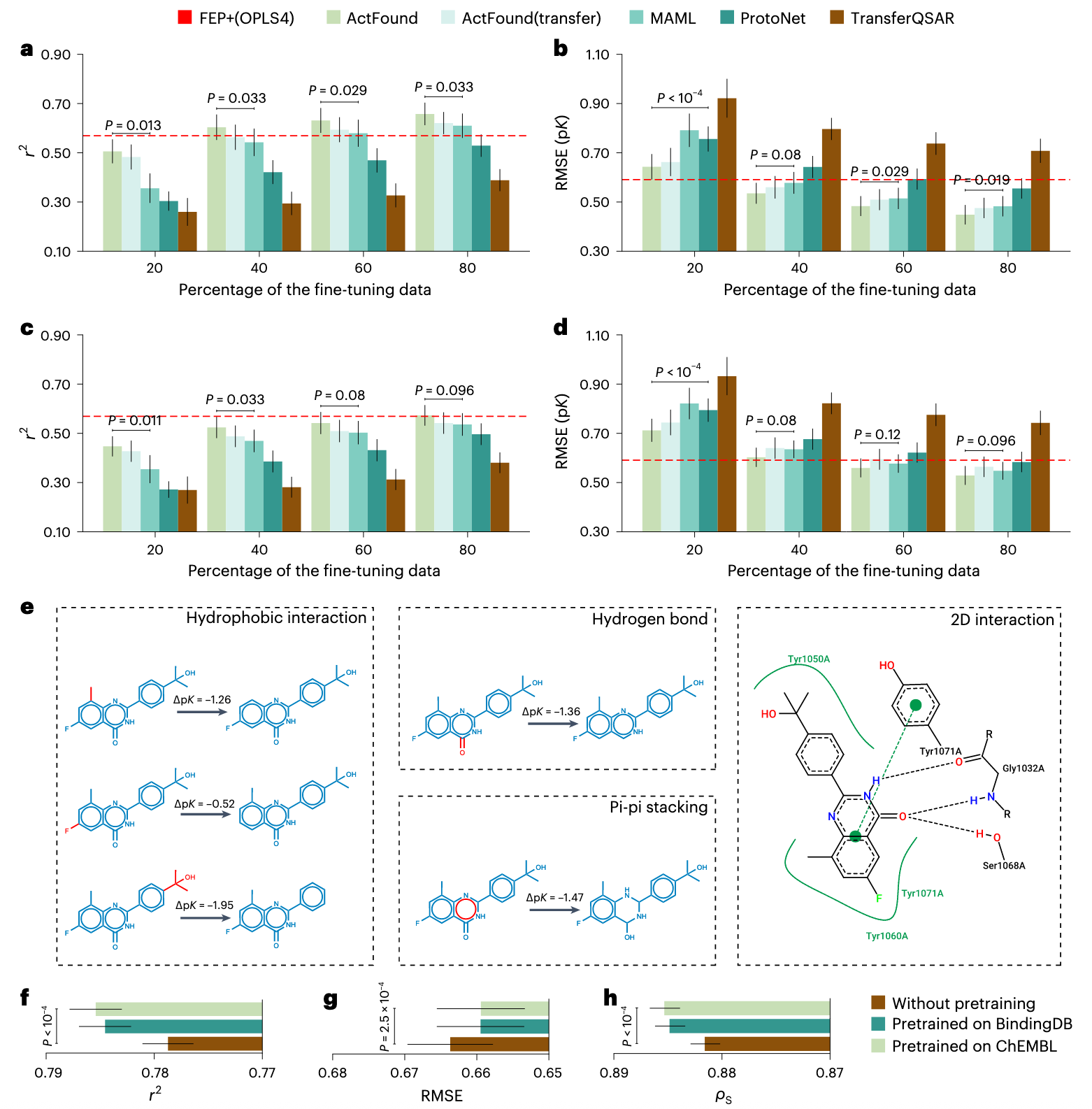
图4 | FEP基准与癌症药物反应预测的评估 a,b, 在FEP基准上比较不同方法,当20%、40%、60%或80%的实验活性数据用于微调时,以
2.4 FEP的机器学习替代方案
为进一步展示ActFound在药物设计中的实用性,研究将其应用于两个FEP基准测试。FEP是重要的基于物理的计算方法,已在多个药物发现项目中取得成功,被广泛用于指导药物结构优化以提升生物活性。然而,FEP计算资源消耗极大,计算两个化合物之间相对结合自由能差异往往需要24至48 GPU小时。研究者假设,在ActFound框架中通过成对学习建模的生物活性差异,能够近似结合自由能的相对变化。
在两个FEP基准(各包含16个实验,平均每个实验29个化合物)上,ActFound在不同比例微调数据(20%–80%)下均优于对比方法(图4a,b),且在低数据条件下优势更为显著,说明其能更高效利用有限标注数据。更重要的是,仅用40%数据(平均每个实验12个化合物)微调,ActFound就能超越商业FEP工具FEP+(OPLS4)的性能。为避免数据泄漏,研究还将所有FEP基准化合物从预训练集中剔除,结果ActFound依旧表现良好。与最新的结构建模方法PBCNet及多种DTA方法对比时,ActFound同样优于这些方法,且无需靶蛋白信息。
进一步,研究还测试了利用FEP+(OPLS4)计算数据代替实验数据进行微调(图4c,d)。结果显示这是一个更具挑战性的设定,所有方法性能均下降,但ActFound依然优于对比方法。在使用80%化合物进行微调时,其
最后,研究通过TNKS2靶点实验案例进一步解释了ActFound的优势。研究首先利用ProteinsPlus识别潜在相互作用,然后在最佳活性化合物上进行结构修饰,并预测由此带来的活性变化(图4e)。结果显示,疏水片段的删除、骨架改变以及关键相互作用(如氢键与π–π堆叠)的破坏,均引发了预期方向的活性下降;而将羟基(–OH)替换为甲基(–CH₃)则预测得到活性提升(0.567 pK),与体外实验吻合。
综上,ActFound利用成对学习有效捕捉官能团与骨架的结构差异,为药物优化提供了低成本且高准确度的替代方案,展现了其作为FEP替代工具的潜力。
2.5 癌症药物反应预测
ActFound在小样本生物活性预测中的优异表现进一步激发了其跨领域应用的探索。研究考察了ActFound是否能够用于癌症药物反应预测,目标是对新的细胞系预测药物敏感性。由于细胞系可用基因表达值作为特征,因此在实验中采用了零样本设定:对新的细胞系不使用任何实验数据进行微调。研究使用了 癌症药物敏感性基因组学(GDSC) 数据集,训练监督模型预测细胞系对药物的敏感性。
结果显示,以ChEMBL或BindingDB预训练的ActFound初始化药物特征的模型,相较于随机初始化(Kaiming uniform)的模型,在所有指标上表现更优(P < 5 × 10⁻⁴),证明ActFound能提供更好的化合物表征(图4f–h)。这一结果表明,ActFound在未见过的癌症细胞系药物敏感性预测中具有广泛适用性。为了进一步评估ActFound学习的化合物表征质量,研究还将其与多种分子表征学习方法对比,包括三维的Uni-Mol、二维的Mole-BERT以及一维的SMILES-BERT。在GDSC数据集上进行16–128 shot实验(964个细胞系任务),结果显示ActFound学习到的分子表征全面优于上述方法(补充图39、40),表明其预训练策略是一种有效的分子表征学习方法。
3 讨论
该研究提出了ActFound,一个几乎可覆盖所有实验的生物活性基础模型。该模型结合元学习与成对学习,充分利用多样化实验中的信息,在小样本生物活性预测中显著提升性能。研究验证了其在域内与跨域预测中的突出表现,并展示了其在FEP基准与新细胞系药物敏感性预测中的实用价值,说明ActFound能够成为促进药物发现的重要基础模型。
与现有的无靶点小样本生物活性预测方法相比,ActFound具有两个关键差异:
- 现有方法主要针对摩尔浓度单位的实验,而ActFound对测量单位无依赖,可同时在不同单位的实验上训练并实现泛化;
- 现有方法预测的是生物活性的绝对值,而ActFound通过成对学习预测相对生物活性差异,更有效利用了不同实验间的活性排序相关性。尽管成对学习曾用于QSAR任务的不确定性量化,但在小样本生物活性预测中尚属首次应用。
此外,ActFound与药物-靶点亲和力(DTA)预测相关。现有DTA方法分为结构基与非结构基两类,而ActFound与其不同:
- ActFound利用元学习提升小样本性能,而DTA方法多依赖迁移学习;
- ActFound是一个通用的生物活性基础模型,可利用ChEMBL与BindingDB等大规模公共数据库,适用于无特定蛋白靶点的实验,因而具备更广泛的适用性。
研究也指出了ActFound的两点局限:
- 目前的预测尚未整合实验的元数据,如靶蛋白序列或实验文本描述,未来将利用这些信息提升实验表征,并拓展至其他任务;
- ActFound目前依赖于简单的分子指纹特征,而未结合预训练分子结构模型。基于其在各项实验中的优异表现,研究推测若引入其他预训练方法,ActFound仍有进一步提升的潜力。