JMC 2025 | CYC_BUILDER: 利用强化学习从头设计靶点特异性环肽结合剂
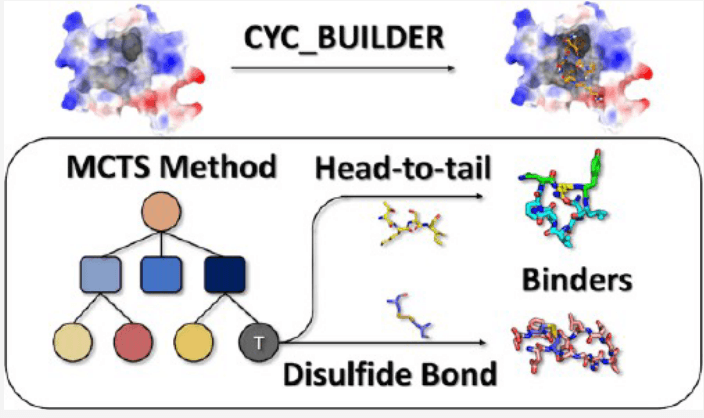
因其结构稳定性和靶向蛋白–蛋白相互作用(PPI)的能力,成为治疗“难成药”靶点的重要分子类型。然而,从头设计靶点特异性的环肽结合剂在计算上仍极具挑战。本文介绍的研究发表于 Journal of Medicinal Chemistry,提出了全新设计框架 CYC_BUILDER,结合片段拼接、蒙特卡洛树搜索(MCTS)与强化学习策略,可高效生成头尾酰胺或二硫键环化的肽段。相比现有方法,CYC_BUILDER 在结合能、结构多样性和效率方面均表现更优。研究进一步将其应用于 TNFα 靶点,成功设计出多种活性环肽,其中4 个在实验中显示出良好的结合能力和细胞抑制活性。CYC_BUILDER 为环肽药物设计提供了强大而高效的工具,在药物开发与合成生物学中具有广泛应用潜力。
获取详情及资源:

0 | 摘要
环肽是一类对难以成药靶点,特别是蛋白–蛋白相互作用,具有巨大潜力的治疗候选分子。然而,在计算上设计环肽结合剂依然面临重大挑战。本文提出了 CYC_BUILDER —— 一个基于强化学习的设计框架,能够通过片段组装和高效的头尾酰胺键或二硫键环化,实现环肽的自动生成。该方法采用 蒙特卡洛树搜索(Monte Carlo Tree Search, MCTS) 引导片段选择、肽链增长以及结构优化。
实验结果显示,CYC_BUILDER 能够成功重建已知环肽-蛋白复合物的天然结合序列与构象。研究团队还将其应用于 肿瘤坏死因子 α(TNFα) 的环肽结合剂设计,并与 AfCycDesign 与 Anchor Extension 方法进行了比较,在结合能、结构多样性与设计效率方面均表现出色。进一步地,研究人员对九种设计肽进行了活性测试,其中有四种显示出强效的结合能力与细胞活性。
CYC_BUILDER 为环肽的发现与优化提供了一种强有力的计算工具,在治疗药物开发与合成生物学中具有广泛应用前景。
1 | 引言
环肽作为一类具有生物活性的分子,近年来在多种细胞过程的调控中展现出重要作用。许多疾病涉及复杂的蛋白–蛋白相互作用(PPI)网络,难以用传统小分子进行靶向干预。相比之下,环肽能够结合体积较大的蛋白表面,尤其是那些被视为“难成药”的区域,因此在治疗复杂疾病方面具有巨大潜力,且同时具备良好的生物降解性与合成可及性。
环肽在多数情况下展现出比线性肽更优异的稳定性、亲和力和生物活性,已有多种环肽药物获批用于癌症、T 细胞淋巴瘤、狼疮肺炎和止血等疾病的治疗。此外,环肽还被视为“分子胶”或可用于构建 PROTACs(蛋白降解靶向嵌合体),因而能够靶向传统难以作用的蛋白,提供全新的治疗策略。
近年来,机器学习的应用极大推动了小分子药物和蛋白设计的发展,包括对环肽的设计探索。相关数据库(如 CREMP)促进了高效的环肽构象采样,而模型如 StrEAMM、RINGER 等加深了对环肽结构的理解。同时,AlphaFold 在环肽主链及其与蛋白复合物的结构优化中也展现出潜力。
然而,基于蛋白结构的环肽配体从头设计仍属探索阶段,其主要受限于已知环肽–蛋白复合物晶体结构数量稀少。在此背景下,本文提出了一种结构驱动的环肽结合剂设计方法 CYC_BUILDER,其核心为 蒙特卡洛树搜索(Monte Carlo Tree Search, MCTS)与强化学习算法。该方法通过片段构建策略在蛋白结合位点内生长肽链,实现天然类稳定构象的环肽构建。结合同时考虑结合能与结构合理性的奖励函数,CYC_BUILDER 能够针对多种靶点高效生成环肽结合剂,具有良好的跨靶点泛化能力。
与现有方法(如 Rosetta 框架下的 Anchor Extension、主链匹配策略、AfCycDesign、RFpeptide 等)相比,CYC_BUILDER 展示出更强的结构多样性探索能力与环化路径灵活性(可处理头尾闭环或二硫键环化),其所生成的序列与主链也展现出较强的多样性。本文进一步将该方法应用于 TNFα(肿瘤坏死因子α)环肽结合剂的设计与实验验证,并成功获得了高效的肽类抑制剂。
2 | CYC_BUILDER 方法概述
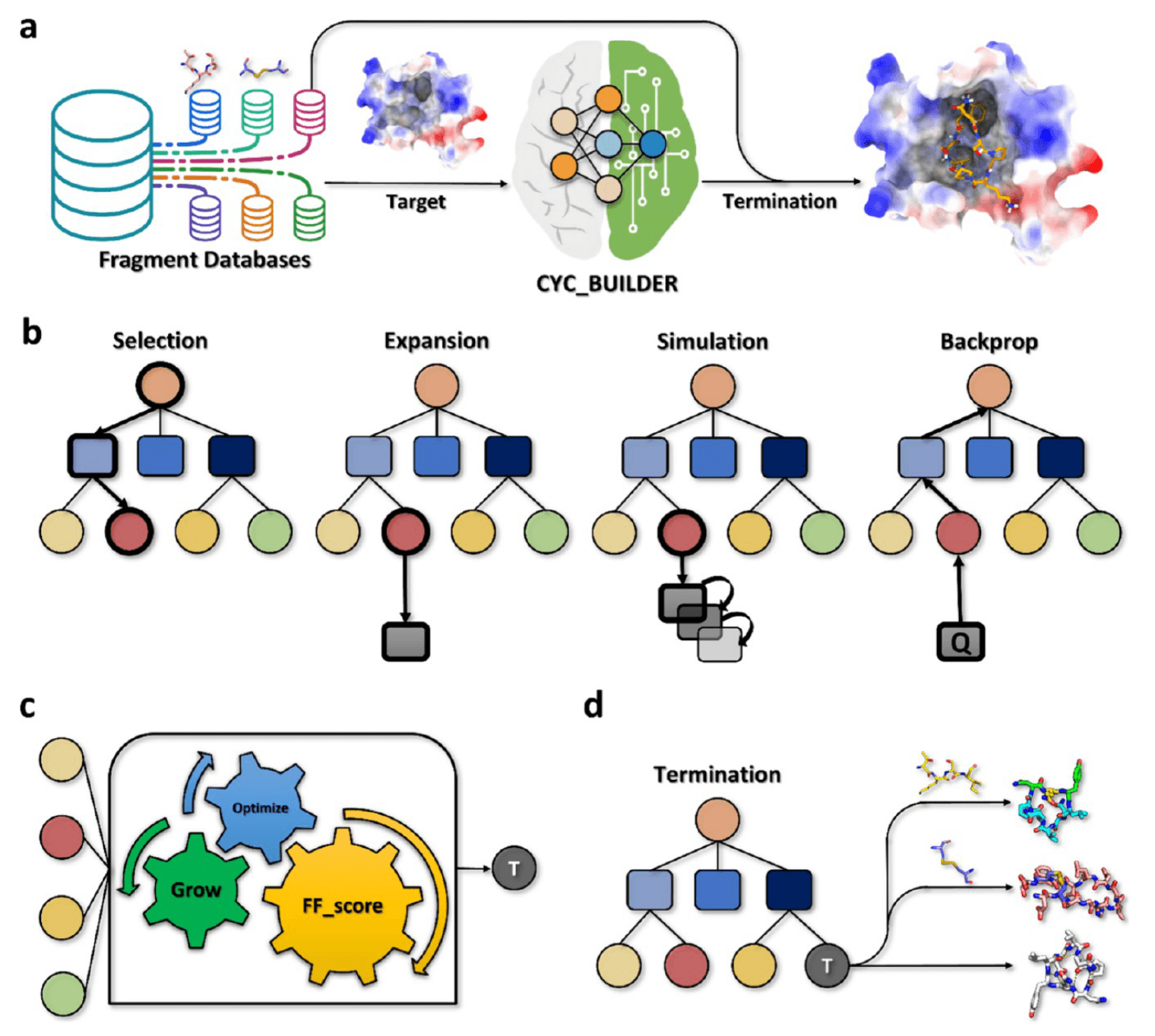
CYC_BUILDER 是一个通用的片段生长式结构驱动环肽设计框架(如图 1a),基于从蛋白–蛋白界面提取的片段数据库,可高效构建头尾酰胺闭环或二硫键环化的肽段,适用于表面或口袋区域等多种蛋白界面类型。
系统以 蒙特卡洛树搜索算法 驱动片段生长与环化过程,在不依赖复杂训练数据的情况下,逐步优化片段选择以获得优质构象。评分模块则综合评估构象稳定性、结合亲和力及环化倾向。整体框架由四个模块组成:片段采样、片段生长、结构优化、主链闭环(图 1b、1c)。
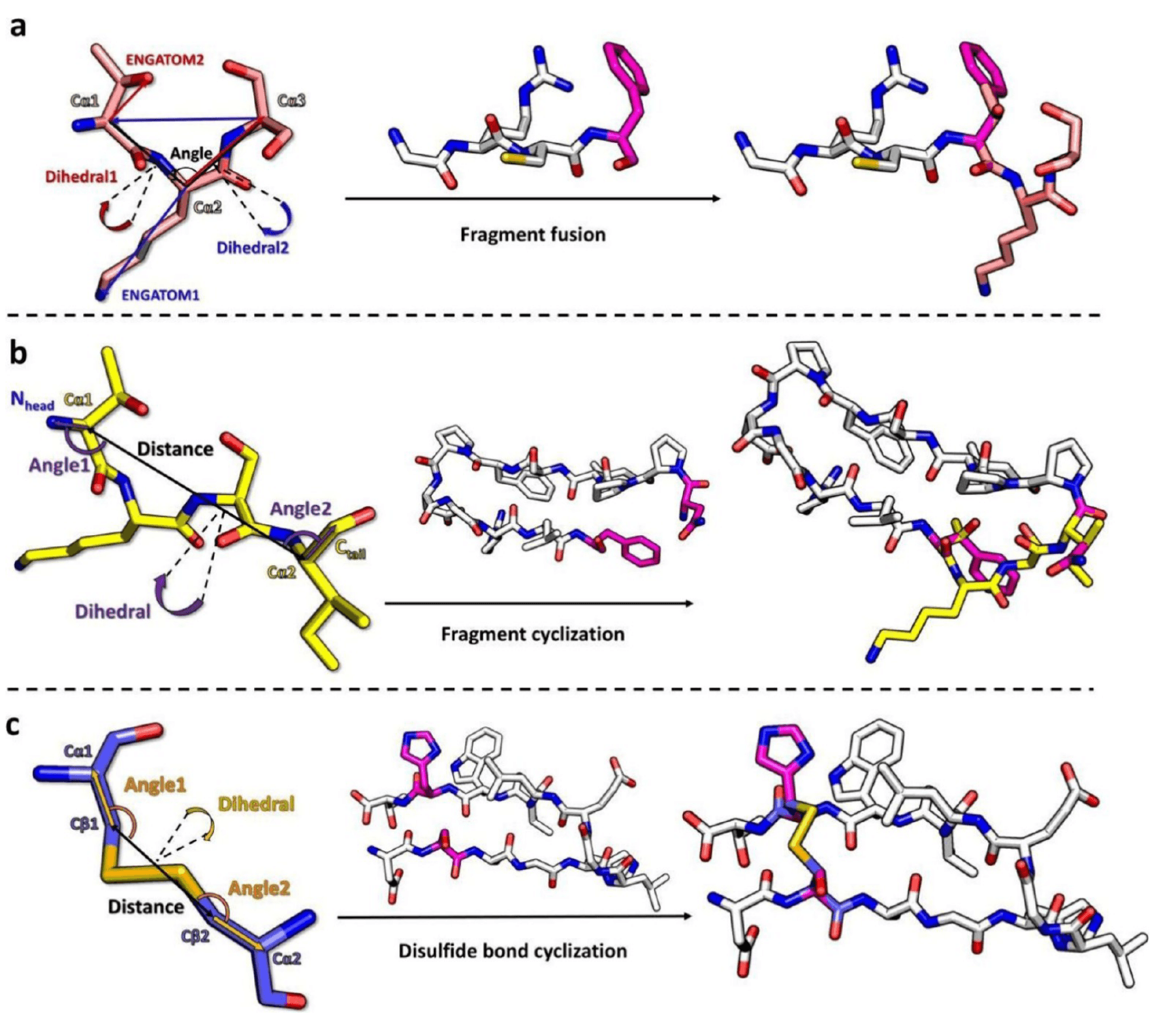
生长过程由种子片段(seed)开始,作为 MCTS 树的根节点。该种子可从已有蛋白或肽结合位点的热点区域提取,也可通过“寻找种子片段算法”自动生成(见 5.2 节)。每一个生长步骤都涉及从三肽片段库中采样新片段,并拼接至当前肽段(图 2a)。
为提升采样效率,三肽片段根据骨架构象、中间残基取向以及极性与体积特征进行分类(20 个天然氨基酸分为 7 类,见 5.1 节与表 S1)。采样概率矩阵根据片段类别初始化,所采样片段则通过柔性拼接算法组装至现有结构。每一次片段拼接后都会生成新的叶节点,该结构将通过评分函数进行打分,评分包含四项指标:结合能、界面性质、结构稳定性、环化倾向。
评分结果将反向传播,用于更新当前状态的采样概率矩阵与状态值;同时判断当前叶节点是否满足终止条件——若满足,则执行环化操作,否则继续生长(见图 1d 与图 2b、2c)。
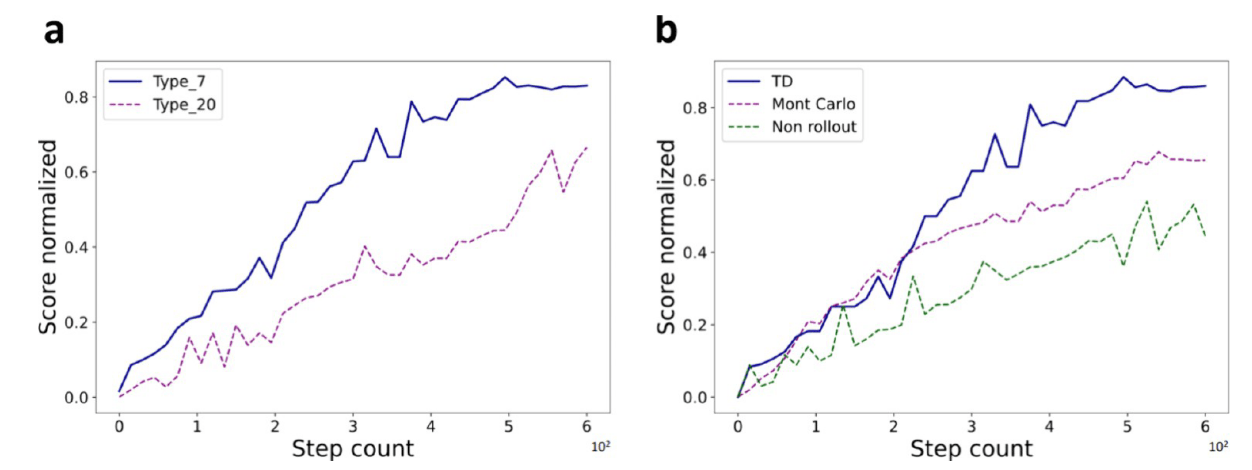
不同于传统 MCTS 使用的“随机延伸直至终止”的 rollout 策略,CYC_BUILDER 采用基于时间差分(TD)算法的 rollout 策略,避免因 episode 不能终止(即未成功环化)而陷入死循环。此外,模型还使用策略梯度优化采样矩阵与评分权重,最大采样深度与子节点数可由用户自定义(见 5.5 节)。
在结构优化模块中,生长过程中对主链进行扰动,随后对侧链进行重排。开发了一套高效的打分函数评估肽–蛋白结合能力、主链构象稳定性以及环化潜力(详见附录 S2)。最终输出的环肽–蛋白复合结构将通过 Rosetta 工具集 进一步优化与筛选。
CYC_BUILDER 在一台普通服务器上即可高效运行(仅需 10 个 Xeon Gold 6132 CPU,每个 28 核,主频 2.60 GHz),充分展现了其高效率与低资源需求的优势。
3 | 结果
3.1 再生测试(Regeneration Test)
为验证 CYC_BUILDER 是否能够生成具有天然相互作用模式与相似结合亲和力的环肽结合剂,作者从 ADCP 环肽测试集中进行了数据裁剪。该测试集包含蛋白与环肽复合物的 X 射线晶体结构。研究选取了其中包含 6–20 个天然氨基酸残基 的环肽,并移除了序列冗余样本(肽段序列相似度 ≥70% 或受体蛋白序列相似度 ≥80%,由 Biopython 计算)。
对于包含二硫键闭环的复合物,仅保留了 仅含一个二硫键 且两半胱氨酸之间的残基数量占总残基数至少 50% 的肽段。最终得到裁剪数据集 CYCB19C,包含 7 个头尾酰胺闭环肽 和 12 个二硫键环肽,共 19 个条目,用于后续再生测试(详细见附表 S4)。
每个案例中,种子片段由种子生成算法产生(见 5.2 节,结合位点定义为原始配体几何中心 10 Å 内的残基),并输入生成算法进行设计。生成时所采用的环化方式(头尾闭环或二硫键闭环)与原始数据一致。每个目标蛋白仅运行一次生成流程,设置最大采样深度为 5,最大子节点数为 1000。
首先,使用 Rosetta 评分系统 对生成的静态结构进行分析,计算其结合能:
每个设计的环肽结合剂分别计算出其 最佳(最低)Rosetta 结合能 ΔG 与 前十个结构的平均 ΔG(结果见表 1)。值得注意的是:
- 42.1% 的设计结合剂的最佳 ΔG 低于原始天然配体;
- Top10 的平均 ΔG 范围为 −31.71 到 −49.97 kcal/mol,显示出优异的预测亲和力。
同时,研究还计算了 native contacts 恢复率(FNC) 以评估生成肽是否能够还原天然相互作用,结果显示所有案例中 FNC 均保持在 50% 以上。
为进一步验证生成结构的结合稳定性,研究者使用 ADCP 工具对 CYCB19C 数据集进行再对接测试。每个案例中采样 250 万个 docking 构象,执行 50 次并行搜索。结果如下(详见附表 S2):
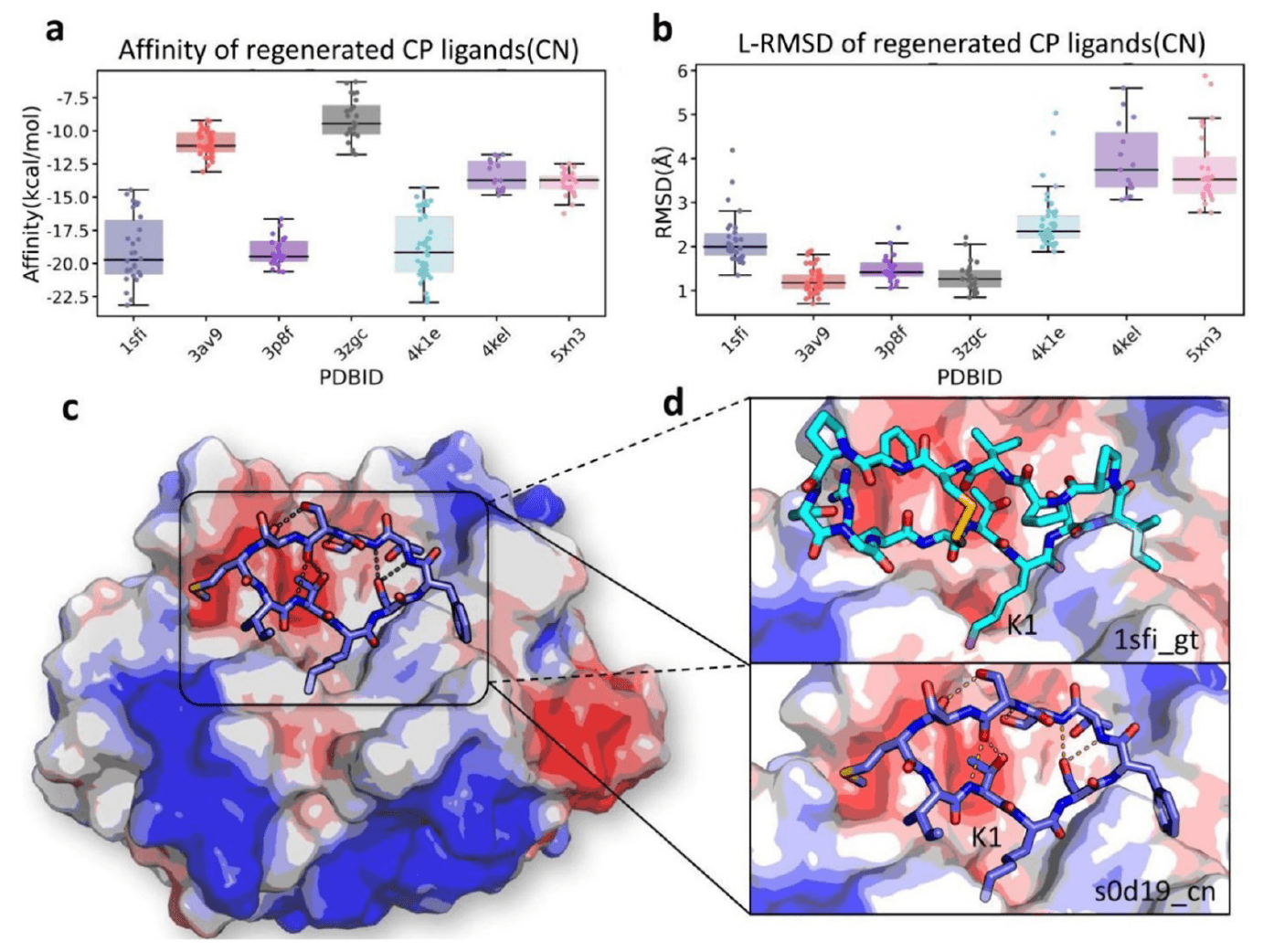
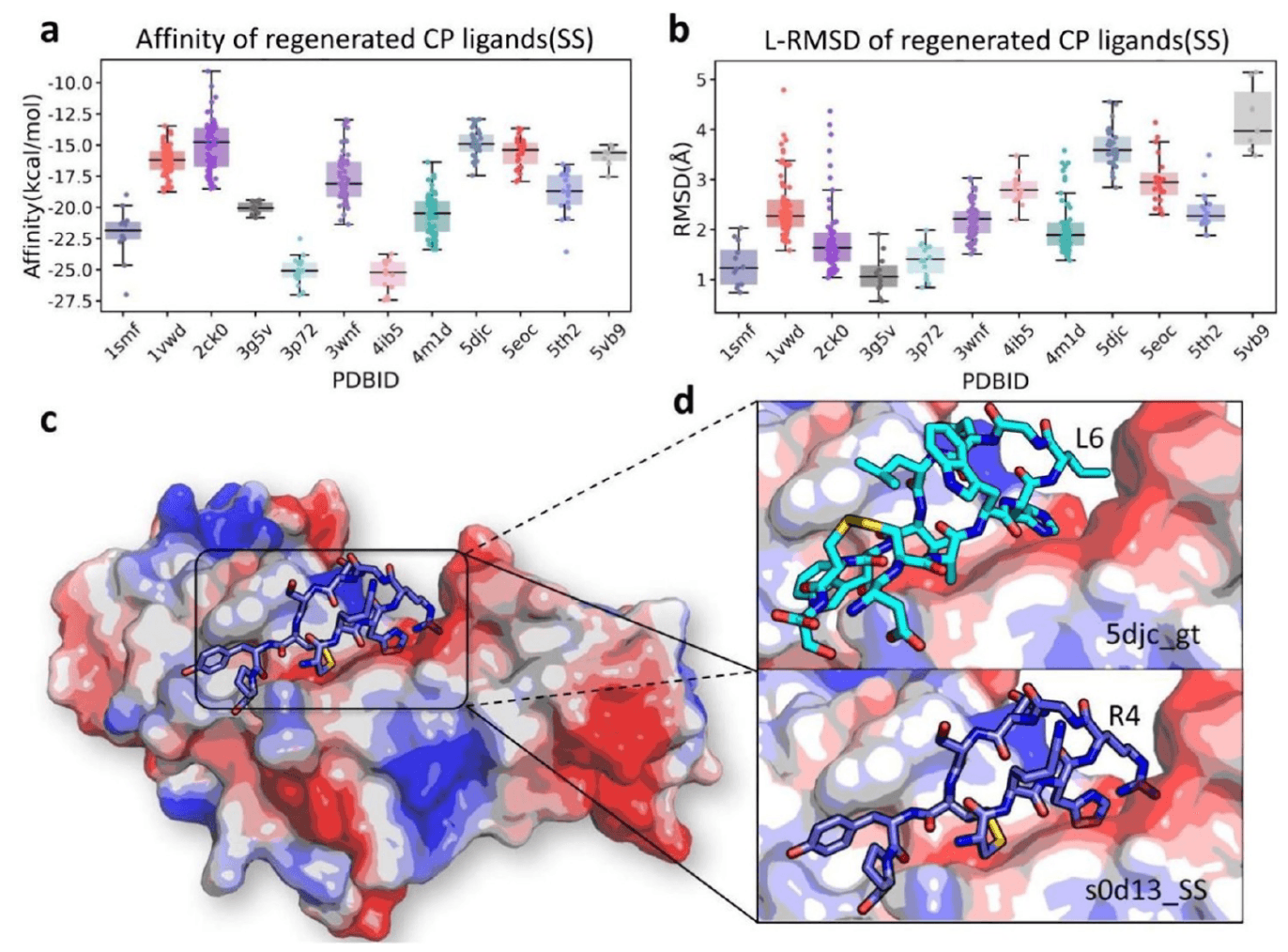
- ADCP 对天然环肽的结合位点预测效果良好,L-RMSD(配体 Cα)通常 < 2 Å,整体成功率为 78.9%;
- 在 63.2% 的案例中,设计肽对接亲和力优于天然肽;
- 所有最佳对接结果的 L-RMSD 均低于 3.5 Å,超过半数设计肽的 L-RMSD < 2.0 Å;
- L-RMSD 与亲和力的分布情况如图 3a,b 与图 4a,b 所示,大部分结果都表明生成肽具备 结构保守性与稳定性。
此外,相较于天然肽,CYC_BUILDER 所生成的结合剂往往能覆盖更大结合界面,并形成更多 疏水与静电相互作用。例如:
- 在牛 β-胰蛋白酶中,所生成的头尾闭环肽 s0d19_cn 能重建 K1 位点的关键氢键和疏水作用,且在口袋区域形成额外疏水作用(图 3c,d);
- Ig γ链 C 区中,设计肽 s0d13_ss 引入了一个正电荷的 Arg(R4),与带负电的靶蛋白表面形成互作,而天然肽在该位点是疏水残基 Leu(L6)(图 4c,d);
- 更多再生结果见图 S6。
此外,研究还对打分函数的不同组成进行了 消融实验(见表 S5),使用优化后的平均 ΔG 与环肽总能量(CYCttE)评估结合亲和力与结构合理性:
- 移除分子间作用力评分项,ΔG 上升 16.61 kcal/mol;
- 移除分子内评分项,CYCttE 上升 13.24 kcal/mol,同时结合力下降;
- 去除环化势能项,增加了搜索步数与运行时间,且整体输出质量下降(ΔG 上升 3.91 kcal/mol,CYCttE 上升 11.35 kcal/mol)。
总体而言,完整打分函数表现最佳,证明其在多指标下兼顾了亲和力与结构稳定性,是当前设计体系中不可或缺的组成部分。
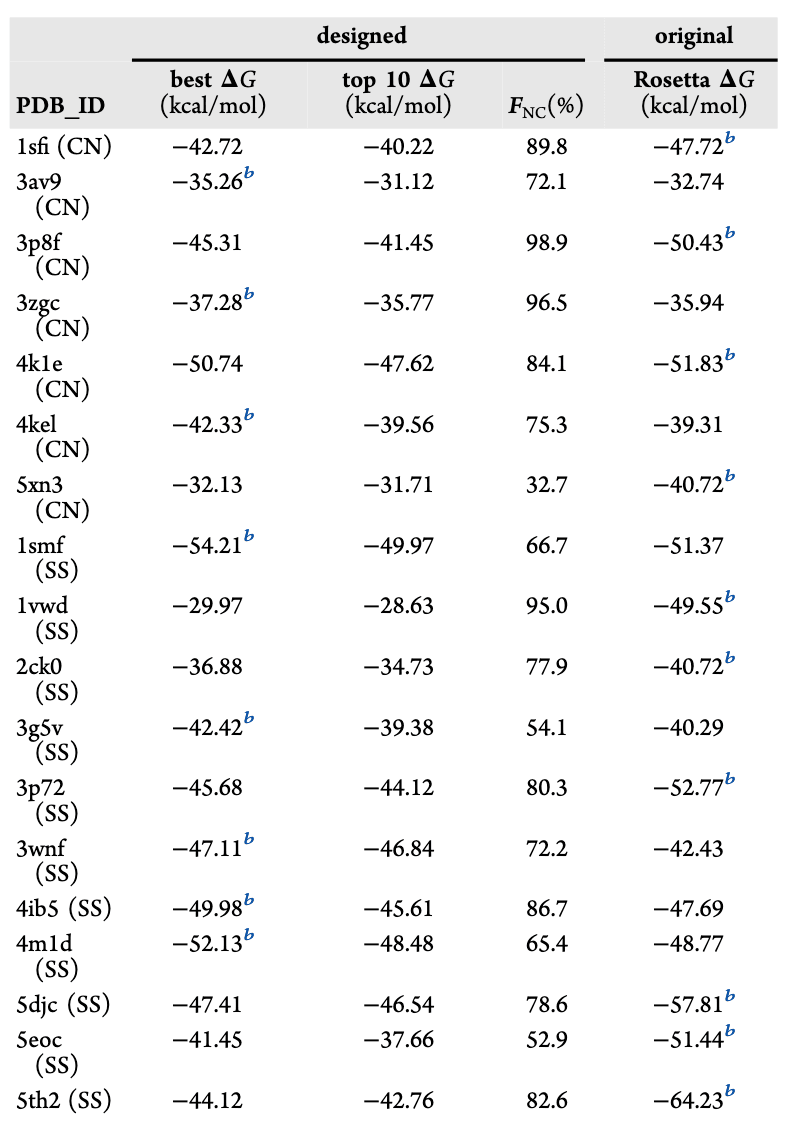
3.2 靶向 TNFα 的环肽从头生成
肿瘤坏死因子 α(TNFα) 是一种参与全身性炎症反应的细胞因子,在急性期免疫反应中发挥关键作用。本文使用 人源 TNFα 与纳米抗体 VHH2 的复合物(PDB ID: 5m2j)界面 作为种子片段搜索模块的输入。
首先,对 TNFα–VHH2 界面进行 丙氨酸扫描(alanine scan),计算相互作用残基的结合自由能(ΔG)。随后,采用滑动窗口策略选取 ΔG ≤ −2.0 kcal/mol 的连续三肽片段,加入种子库(见图 S7a)。接着,调用 CYC_BUILDER 的 种子搜索算法,生成候选片段,并基于 Rosetta 接口分析器评分与 Scoreseed 值,筛选出 ΔG 最低的 Top10 片段加入种子库。
在此基础上,启动环肽生成程序,从每个种子出发,设置最大采样深度为 5,子节点数上限为 500,共生成 10 万条头尾环化的候选肽段,肽长为 7–15 个残基。
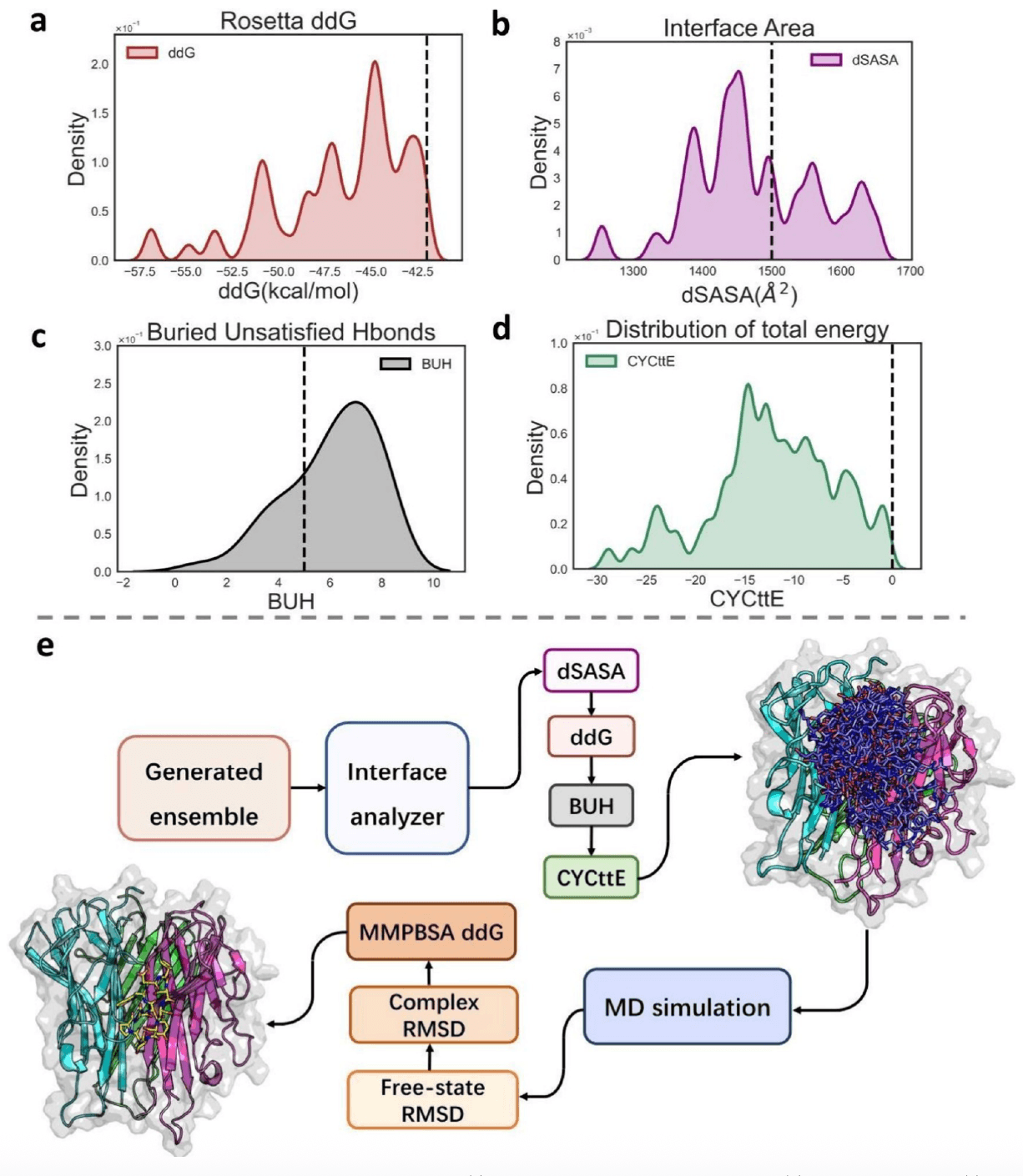
随后对候选肽进行初步筛选,筛选标准如下(详见图 5):
- 结合界面面积 > 1500 Ų
- 结合自由能 ΔG < −40 kcal/mol
- 接口未满足氢键数 < 5
- 总肽段能量 < 0 kcal/mol
通过筛选后,对每条环肽执行 100 ns 分子动力学(MD)模拟。为了评估肽段游离状态下的构象变化,计算其末 20 ns 模拟构象与设计构象之间的 主链 Cα RMSD。结果显示,42.5% 的环肽 RMSD < 1.8 Å(图 S8),从中选出 37 条 RMSD ≤ 1.5 Å 的肽段用于后续复合结构模拟。
这些设计肽–靶标复合物进一步进行 100 ns MD 模拟。将最后 20 ns 的构象与原始设计结构进行比对,对以下两项进行评估:
- 主链 L-RMSD
- 接口残基 RMSD(iRMSD)
其中,接口残基定义为:在设计构象中埋藏溶剂可及表面积(SASA)大于 30 Ų,或其占总 SASA 比例超过 30% 的残基。
若 iRMSD ≤ 2.5 Å,表明结合方式保持稳定,则不进入实验测试(即排除构象变化太小者,以集中测试可能形成新互作模式的设计)。
最后,使用 MMPBSA(分子力学泊松-玻尔兹曼表面积)方法 计算复合物的结合自由能:
筛选标准为:
- 总 ΔG ≤ −35 kcal/mol,或
- 单残基 ΔG(ΔG/n)≤ −3 kcal/mol
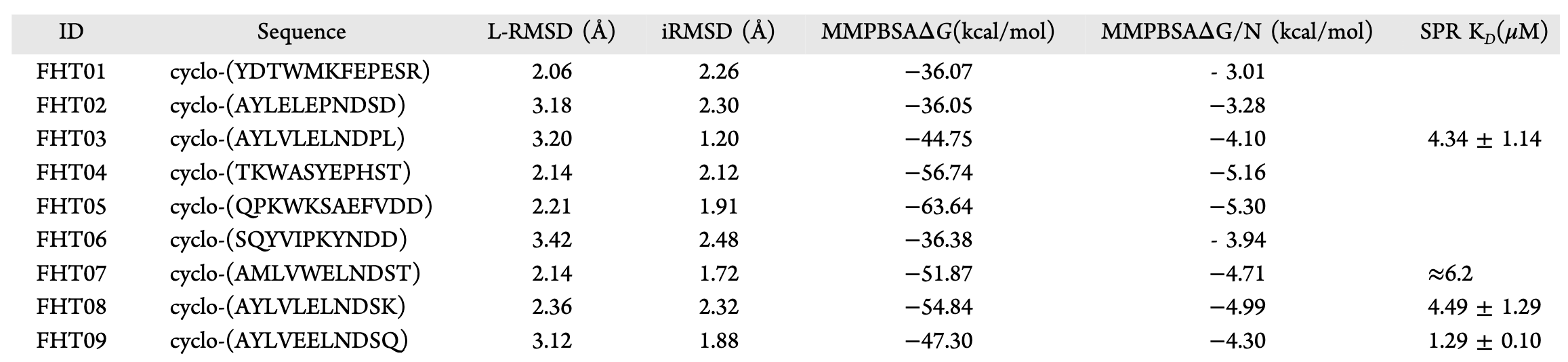
满足上述任一条件的设计肽(FHT01–09)被选中用于化学合成与实验结合测试(相关 MD 模拟的 L-RMSD 轨迹见图 S9)。
3.3 候选结合剂的实验验证
首先,研究通过 表面等离子共振(SPR)实验 测试了所设计环肽与 TNFα 的结合能力。将纯化后的 TNFα 固定在 CM5 芯片上,设计的环肽作为分析物注入进行检测。在六个候选环肽中,FHT03 展现出与 TNFα 的结合活性,拟合的解离常数为 KD = 4.34 μM(基于 1:1 Langmuir 结合模型,见图 6a)。
进一步在 基于 luciferase 的细胞活性实验 中,将不同浓度的 FHT03 与 TNFα 共孵育,观察其对 TNFα–TNFR 相互作用的抑制效应。实验发现:FHT03 在剂量依赖下显著抑制 TNFα 活性(图 6b)。
随后通过 实时荧光定量 PCR(qPCR) 检测四个 TNFα 下游基因的时间依赖性表达,包括三个早期响应基因(A20、IL6、CXCL2)和一个晚期响应基因(NK4)。以 4 ng/mL TNFα 刺激细胞作为阳性对照,大部分细胞能激活 NF-κB 信号通路。抑制实验中,加入 25 μM FHT03 并与 TNFα 共同孵育 12 小时,在不同时间点(1、2、3、6、10 h)收集细胞,检测基因表达(图 S11)。结果表明,FHT03 能显著降低上述基因的表达峰值,进一步验证其在细胞水平对 TNFα–NF-κB 通路的抑制作用。
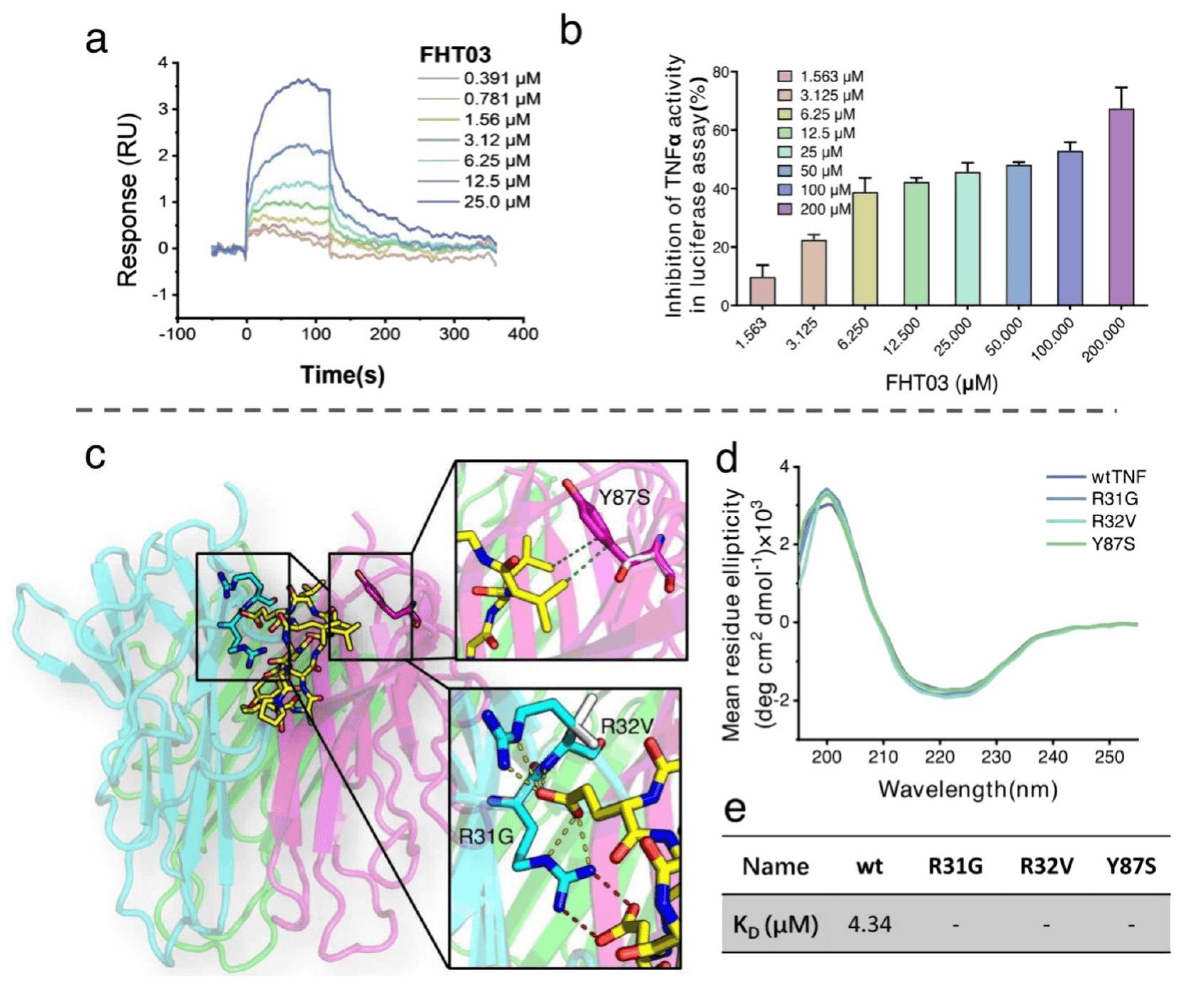
换句话说,FHT03 不仅展现出 中等水平的结合亲和力,也具备 在细胞中抑制 TNFα 功能的能力。
MMPBSA 的能量分解结果揭示了与 TNFα 结合的关键残基:
- 链 B 的 R31 和 R32,对结合能的贡献分别为 −20.92 kcal/mol 和 −9.94 kcal/mol;
- 链 C 的 Y87,贡献为 −4.68 kcal/mol(见表 S6)。
为进一步验证 FHT03 与 TNFα 的结合模式,研究构建了三个 TNFα 突变体:R31G、R32V、Y87S(图 6c),旨在破坏其与 FHT03 之间的关键氢键、盐桥或疏水互作。CD 光谱表明突变体保留了与野生型蛋白类似的二级结构(图 6d)。SPR 实验显示,FHT03 无法与这三种突变体结合(图 6e),验证了其结合构象的正确性与特异性。
由于 FHT03 显示出较好活性与明确结合位点,研究团队将自由态肽段的 MD 筛选 RMSD 阈值放宽至 1.8 Å,在其对应的种子片段(Seedfht03,图 S7b)下进一步筛选,使用相同的 MD 流程最终得到三个新环肽结合剂:FHT07、FHT08 和 FHT09。
在 SPR 验证中,三者均表现出与 TNFα 的结合活性:
- FHT07:KD = 6.2 μM
- FHT08:KD = 4.49 μM
- FHT09:KD = 1.29 μM(图 7a)
在 luciferase 实验中,FHT08 和 FHT09 在更低浓度下表现出比 FHT03 更强的抑制效果(图 7b),并且能显著抑制 TNFα 诱导的四个 NF-κB 靶基因表达(图 7c,d)。
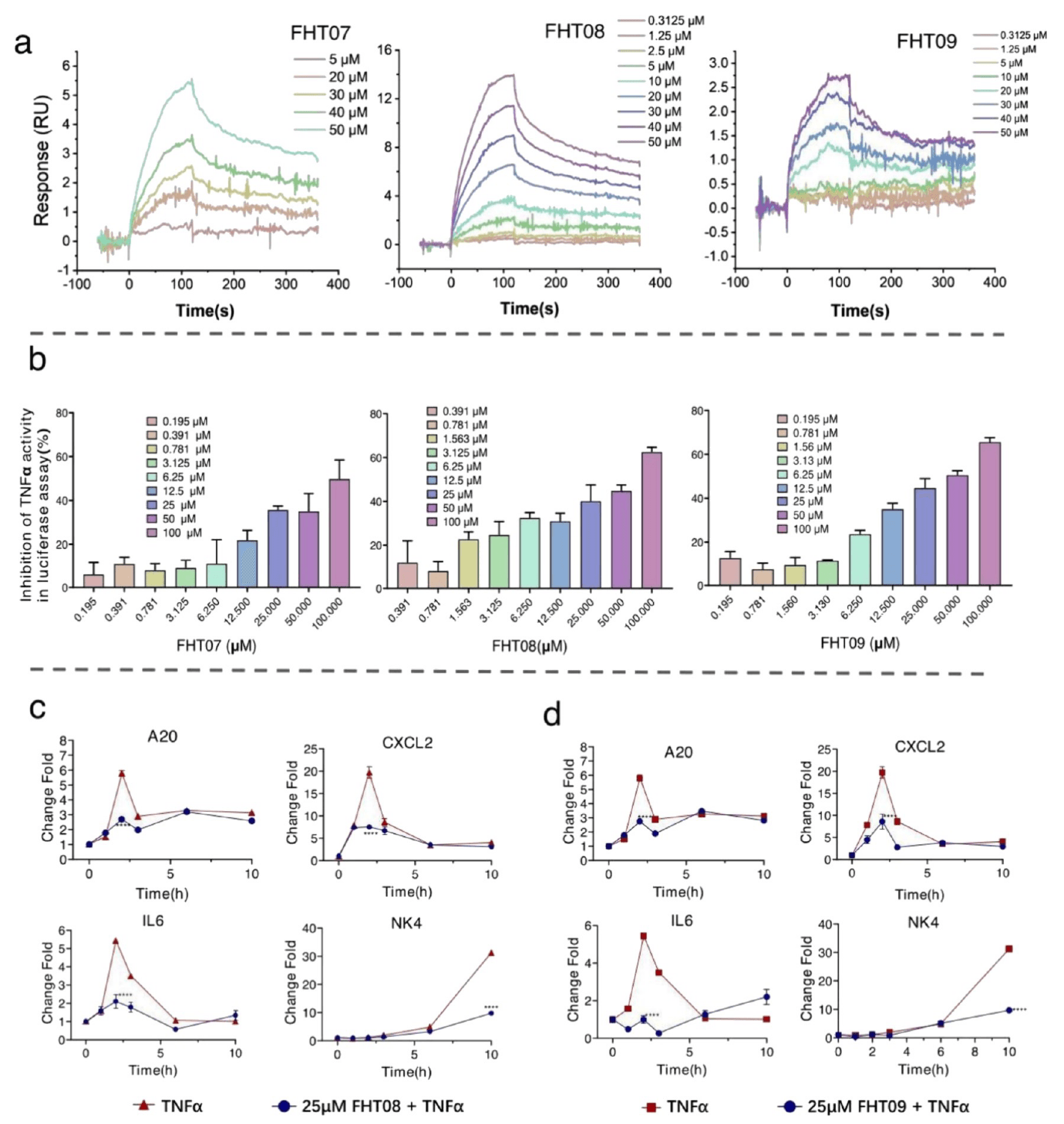
综上,这些结果表明:
FHT08 与 FHT09 是成功实现 TNFα 抑制的设计环肽代表。
本研究所开发的环肽配体生成模型,不仅成功生成了具有活性的 TNFα 抑制剂,并在 SPR 结合实验与细胞水平功能验证中取得积极结果,进一步证明了 CYC_BUILDER 所采用设计策略的有效性与鲁棒性。
4. 结论与讨论
本文提出了 CYC_BUILDER,一种结合片段组装与强化学习采样的环肽结合剂生成方法,能够高效生成针对特定靶点的 头尾酰胺闭环或二硫键闭环 的肽段,并已成功应用于 TNFα 抑制剂的从头设计与实验验证。
在 TNFα 的从头设计任务中,CYC_BUILDER 在计算结合能、结构多样性与生成效率上均优于现有方法(AfCycDesign 与 Anchor Extension)。例如,以往报道中通过高通量实验筛选获得了与 TNFα 结合的双环肽(KD = 5.3–28.0 nM),但其作用靶点为 TNFα 的三聚界面,并不干扰其与 TNFR 的相互作用。相比之下,团队此前开发的 CycDockAssem 能生成直接作用于 TNFα–TNFR 接口的抑制肽,机制更加功能相关。然而,CycDockAssem 计算成本高昂,而 CYC_BUILDER 提供了一种更高效的替代方案,在设计周期短、实验活性验证成功的基础上实现了更具实用价值的环肽筛选。
由于目前蛋白–环肽复合物结构数据较少,CYC_BUILDER 在以模拟为主的评价体系下依然展现出出色性能,其优势来自三个关键模块:
- 面向蛋白–蛋白接口的三肽片段库:支持生成高亲和力且结构多样的肽段;
- 蒙特卡洛树搜索(MCTS)框架:高效引导片段采样与装配过程;
- 多目标评分函数:综合考虑构象稳定性、结合亲和力与环化倾向。
该模型无需预训练,可进行实时决策,并提供可解释性结构结果。团队在 CYCB19C 数据集上进行 600 步的性能测试,发现 rollout 策略显著影响收敛速度与最优得分,其中 time-dependent 方法表现最佳。此外,将氨基酸分类为 7 类(见表 S1)也有助于提高搜索效率。
在 TNFα 案例研究中,研究分析了生成肽段的多样性:
- 相同长度的肽段主链平均两两 L-RMSD 为 8.66 Å;
- 序列平均相似性为 0.33。
这表明 CYC_BUILDER 能够在序列与结构两个维度上生成丰富多样的结合模式(见图 S10 与图 5e)。
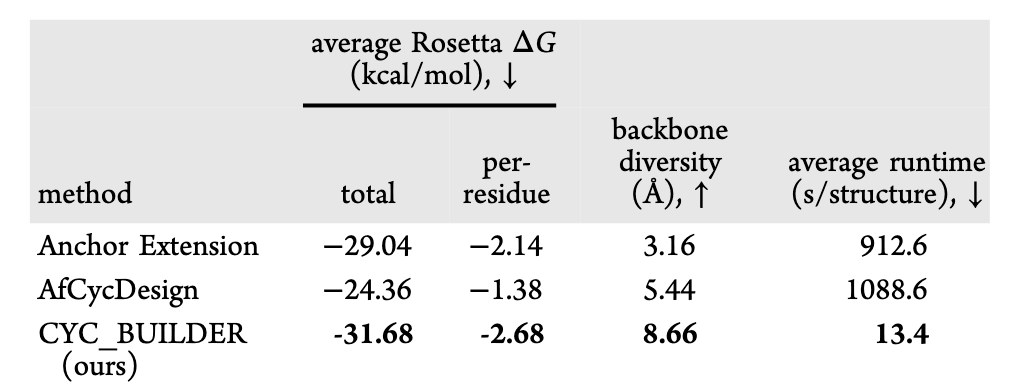
在与 AfCycDesign 和 Anchor Extension 方法的对比实验中,使用相同的结合热点作为参考,生成长度为 7–15 的肽段。CYC_BUILDER 实现了:
- 最优的平均 Rosetta 结合能 ΔG = −31.68 kcal/mol;
- 最优的单位残基结合能 ΔG/n = −2.68 kcal/mol;
- 最高的主链结构多样性(以平均主链 RMSD 计);
- 更短的运行时间,显示出明显的计算效率优势(见表 3)。
在从头设计的 9 个候选环肽中,4 个在实验中验证具有可测结合活性。不过当前的结构筛选流程仍存在不足。例如:用于筛选的 100 ns MD 模拟可能无法捕捉所有关键结合构象,导致假阴性或假阳性。这一点在表 2 中可见:FHT03 与 FHT07–09 均在实验中表现活性,但未在筛选中排名靠前,说明目前的构象采样时间尺度仍存在限制。
CYC_BUILDER 在同一片段组装框架内同时支持 头尾环化与二硫键环化,根据几何可兼容性与能量有利性进行肽段扩展与闭环。虽然模型本身未对两类环化模式设置偏好,但由于二硫键形成的空间要求更高,在单次采样中头尾环化的出现频率约为二硫键环化的 11 倍。这体现出在结构可行性上的自然偏向,尽管在打分体系中两者被等同处理。
当前版本仅支持 20 种天然氨基酸,但其 模块化架构为未来扩展提供可能,包括:
- 支持非天然氨基酸(UAA);
- 引入多环拓扑结构;
- 使用量子化学或 MD 方法构建复杂环化单元的构象库;
- 使用第一性原理或基于 MD 的力场构建能量评估模块。
这些扩展均可进一步集成入 CYC_BUILDER 的原子类型定义与打分函数中,相关工作正在推进。
展望未来,模型的可靠性还可通过引入更严谨的增强采样方法进一步提升,例如:
- 复制交换分子动力学(REMD)
- metadynamics 等偏置采样方法
- 改进的力场参数
这些策略尤其适用于发现低占比但功能关键的结合构象,能提高筛选的准确性、降低假阴性风险。由于环肽的构象稳定性直接决定其结合模式与生物活性状态,增强采样将有望显著提升 in silico 筛选的可信度。