Nature 2025 | BindCraft: 一次性设计功能性蛋白质结合子
在生命过程中,蛋白–蛋白相互作用(PPIs)起着核心调控作用,但功能性结合子的精准设计长期受限于结合位点复杂性与结构信息缺失。近日发表于《Nature》的研究 《BindCraft: 一次性设计功能性蛋白质结合子》 提出了一种全新的 de novo 设计流程 —— BindCraft,利用 AlphaFold2 反向传播机制,无需筛选即可生成纳摩尔亲和力结合子。
该方法引入靶点柔性、界面优化与深度学习筛选策略,能够在多种结构与功能背景下生成具有实际活性的结合子。研究验证了其在 PD-1、PD-L1、Bet v1、Cas9 等 12 个挑战性靶点上的高成功率,并在中和过敏原、调控基因编辑、AAV 靶向递送等应用中展现出功能效果。BindCraft 不仅提高了设计精度与实验转化效率,也推动蛋白质设计从专业化走向普适化,为治疗与生物工程提供了强有力的新工具。

获取详情及资源:
0 摘要
蛋白质–蛋白质相互作用是所有关键生物过程的核心。然而,这类相互作用所依赖的结构特征极为复杂,因而相关设计任务一直具有高度挑战性。该研究介绍了一种开源且自动化的去新(de novo)蛋白结合子设计流程 —— BindCraft,该方法在实验中的成功率达到了 10% 至 100%。BindCraft 利用 AlphaFold2 的权重,无需高通量筛选或实验优化,即便在缺乏已知结合位点信息的情况下,也能生成具有纳摩尔亲和力的蛋白结合子。研究中成功针对一系列具挑战性的靶点设计了结合子,包括细胞表面受体、常见过敏原、去新设计的蛋白质以及多结构域核酸酶(如 CRISPR–Cas9)。所设计的结合子展示了突出的功能性与治疗潜力,例如:
- 在患者来源样本中有效降低白桦树过敏原的 IgE 结合能力
- 调节 Cas9 基因编辑活性
- 降低食源性细菌肠毒素的细胞毒性
此外,研究还利用特异性细胞受体结合子实现了腺相关病毒(AAV)衣壳的重定向,以实现靶向基因递送。
这一成果代表了向“一设计一结合子”策略的重要进展,在治疗、诊断及生物技术等领域具备巨大应用潜力。BindCraft 的设计策略展现了功能蛋白工程的高效性与广泛适应性。
1 引言
蛋白质在生物体内很少以孤立方式执行功能,其生物活性通常依赖于**蛋白–蛋白相互作用(PPIs)**来完成复杂的生物过程。因此,设计能够特异性靶向并调控 PPI 的蛋白结合子,在治疗和生物技术应用中具有巨大潜力。
然而,传统的蛋白结合子生成方法,如免疫接种、抗体文库筛选或定向进化,通常过程繁琐、耗时,并且在结合位点选择上缺乏精细控制。相比之下,计算蛋白质设计为这一问题提供了强有力的替代方案,使得研究者能够将结合子定制至特定靶点与结合位点。
早期的基于物理的设计方法,例如 Rosetta,通过支架构建与侧链优化,实现了初代结合子的设计尝试。然而,此类方法的实验成功率极低(通常低于 0.1%),且需要对大量设计进行采样。此外,这些方法通常要求将预定义的支架对接至固定靶标结构,容易导致表面不匹配,从而造成结合效果不佳,甚至无法靶向某些特定位点。
随着深度学习的突破,生物大分子建模领域迎来了变革。以 AlphaFold2(AF2)为代表的模型在准确预测蛋白质结构及复杂 PPI 方面展现了惊人能力。已有研究表明,利用 AF2 进行筛选评估能提升结合子设计的成功率,因为它可以判断预测复合物的合理性。此外,深度学习也已被广泛用于蛋白质和结合子的 de novo 设计。
目前最前沿的设计策略通常结合 RFdiffusion(用于主链构建)与 ProteinMPNN(用于序列生成)。在结合子设计任务中,这一组合方案相比以往方法显著提高了成功率。然而,RFdiffusion 仍依赖于将无侧链的主链结构定位于刚性的目标界面,并最终通过 AF2 对复合物进行预测以筛选出可行结合子。这一流程表明,从主链生成到功能性结合界面之间仍存在差距,而 AF2 筛选在一定程度上弥合了这一空缺。
鉴于 AF2 在提高筛选成功率方面的重要作用,研究者进一步提出:是否可以直接利用 AF2 本身来设计蛋白结合子?基于此构想,BindCraft 应运而生 —— 一个用户友好、几乎无需人工干预或高级计算知识的 de novo 蛋白结合子设计流程。
BindCraft 通过 反向传播算法直接作用于 AF2 网络,高效地生成新型结合子与结合界面(见 Fig. 1a)。研究在 12 个结构多样、挑战性强且具治疗意义的蛋白靶点上对其效率进行了验证(见 Fig. 1b),并在无需实验高通量筛选的前提下,为每个靶点均识别出多个高亲和力结合子。
这一成果标志着蛋白结合子按需设计迈出了关键一步,使非专业计算设计团队或缺乏高通量实验设备的研究者也能便捷地开展功能性结合子设计工作。BindCraft 显著降低了设计门槛,拓宽了精准生物分子工程的适用范围。
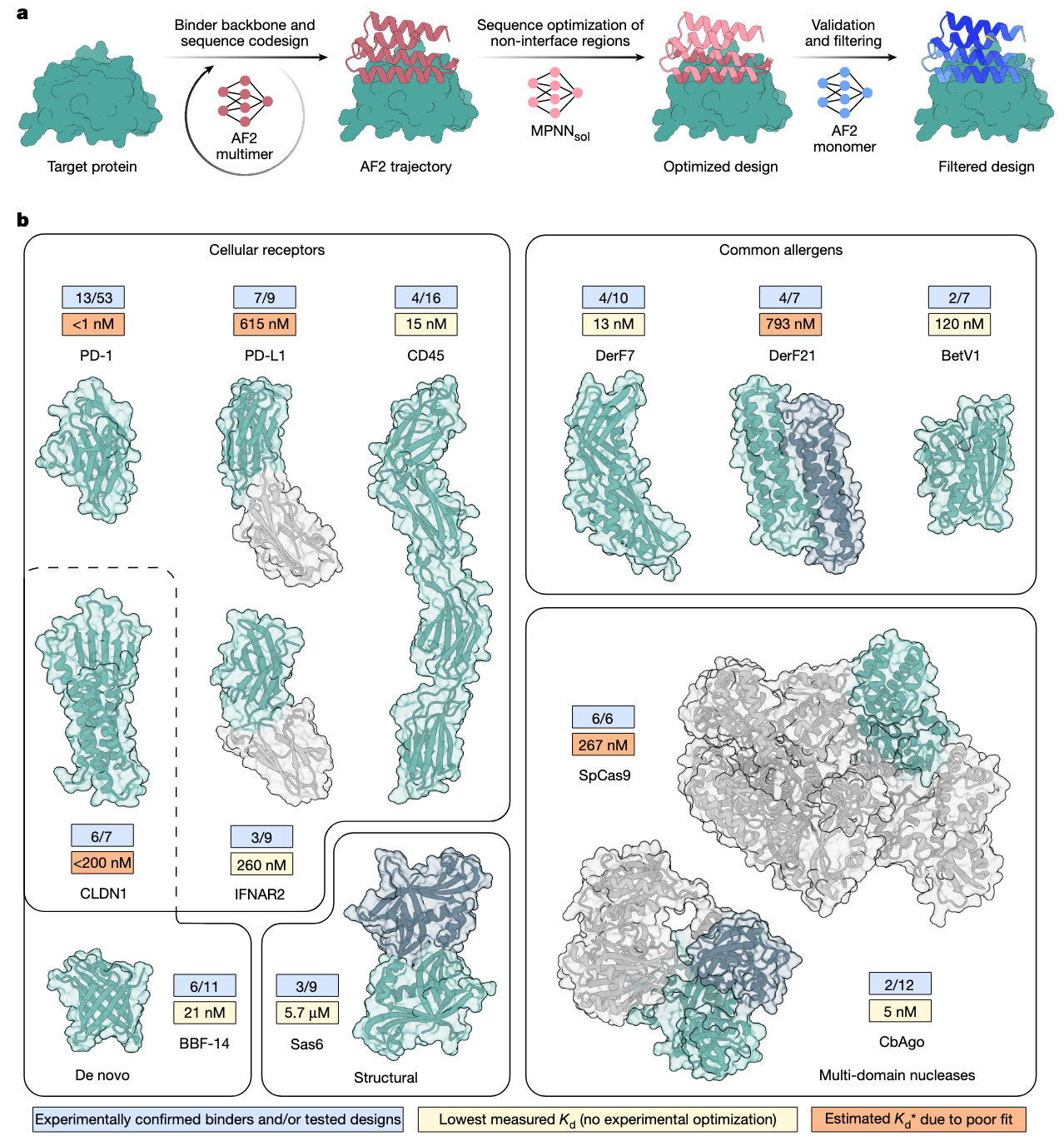
图 1|BindCraft 实现的从头结合子设计流程:a,展示 BindCraft 结合子设计的示意图,先通过 AF2 multimer 生成初始骨架与序列,再利用 MPNNsol 优化其表面与核心区域,同时保持结合界面不变,最终依据 AF2 monomer 预测结果筛选设计;b,结合子设计所针对的蛋白靶点概览,绿色区域为设计中使用的部分,灰色区域为未使用部分,蓝框为 SPR 实验中观察到结合现象的成功设计数与测试总数,黄框表示未经序列优化的最高亲和力结合子的实测
2 高精度的 de novo 蛋白结合子设计
该研究的目标是构建一个高效、易用且自动化的蛋白结合子设计流程,能够依托 AlphaFold2(AF2)实现高精度设计,并将实验筛选需求降至最低。具体来说,BindCraft 采用了 ColabDesign 中对 AF2 的实现方式,将“幻想生成”(hallucination)得到的结合子序列反向传播通过 AF2 网络权重,计算误差梯度以优化序列,从而满足特定设计标准(详见方法部分)。这一思路与此前的 hallucination 方法一致。
通过反复迭代网络,BindCraft 能够同时生成结合子的结构、序列与界面(见 Fig. 1a)。相较于如 RFdiffusion 或 RIFdock 这类在设计过程中固定靶点主链结构的方法,BindCraft 在每一轮迭代中都会重新预测结合子–靶点复合物结构,这使得结合子与靶点的主链与侧链都可具备一定程度的柔性,从而更好地适配结合位点。设计中得到的靶点主链位移均方根差(r.m.s.d.Cα)范围在 0.5 Å 到 5.5 Å 之间(见扩展图 Fig. 1a)。
若想进一步增加柔性,还可通过掩盖输入模板的序列,仅提供 Cα 坐标作为输入(见补充图 Fig. 1a)。为避免生成过多 α-螺旋结构的结合子,流程中引入了“负螺旋损失函数(negative helicity loss)”,从而可以设计出完全由 β-折叠构成的结合子(见扩展图 Fig. 1b),但同时也会降低一定的 in silico 成功率(见补充图 Fig. 1b、c)。
在初始结合子的设计中,BindCraft 采用了 AF2-multimer,因为该模型是在蛋白质复合物上训练的,因此在模拟蛋白–蛋白相互作用(PPI)方面更为精准。值得注意的是,AF2-multimer 所生成的结合界面平均大出 20%,其 loop 区域占比更高、模型置信度也更强(见补充图 Fig. 1d)。为避免序列过拟合至单一模型,设计中使用了 AF2-multimer 的全部五组训练权重。
不过,以往研究表明,AF2-hallucination 所生成的蛋白质在实验中可能出现低表达量。为此,BindCraft 在保持界面结构不变的前提下,进一步利用 MPNNsol(消息传递神经网络) 对结合子的核心与表面序列进行优化(见 Fig. 1a),随后采用 AF2-monomer 对优化后的结合子进行再次预测。由于 AF2-monomer 专门在单体蛋白上训练,能有效降低 PPI 预测偏差,因此非常适合用于界面质量的最终筛选。
考虑到深度学习模型可能偶尔生成不符合物理规律的结构,最终筛选步骤还结合了 AF2 的置信度评分指标 和 基于物理建模的 Rosetta 评分指标(详见方法)。
不同靶点的设计成功率不尽相同,初始 AF2 预测轨迹中约有 16.8%–62.7% 达到满意置信度标准,而经 MPNNsol 优化后、再由 AF2-monomer 复合预测的结果中,有 0.6%–65.9% 最终通过了全部计算过滤条件(见补充图 Fig. 1e)。整体 in silico 成功率受靶点类型与结合子长度影响(见补充图 Fig. 1f)。
与最先进方法 RFdiffusion 相比,BindCraft 在多个靶点与结合子长度组合中展现出相似的设计时间与成功率(见扩展图 Fig. 1c)。值得注意的是,两者在结合界面上的氨基酸分布存在差异,RFdiffusion 设计结果中大体积氨基酸的占比偏低(见扩展图 Fig. 1d)。
研究对 12 个靶点进行了基准评估,以检验其通用性(见 Fig. 1b)。所设计的结合子在序列与结构上都展现出广泛的多样性(见补充图 Fig. 2a 及补充数据 1、2),平均模板建模评分(TM-score)为 0.62,与最近的 PDB 结构比对的序列同源性为 14.4%(见扩展图 Fig. 1e),表明这些设计在保留部分已知结构构件的同时,也探索了大量全新折叠,符合紧凑型支架结构所预期的有限结构多样性特征。
BindCraft 所生成的结合子在几何与化学互补性上表现突出(见补充数据 1),能够构建全新的高亲和界面(见扩展图 Fig. 1e 与补充图 Fig. 2b)。为验证界面的新颖性,研究者使用 PPIRef 数据库将设计界面与 PDB 中已知相互作用进行比较,所有靶点的平均 TM-score 为 0.15,显示出其显著区别于天然 PPI 的特征。
上述所有步骤均被整合为自动化工作流,其参数已针对不同靶点进行优化,确保流程具备良好的通用性。这使得即便是不具备蛋白质设计经验的研究团队,也能按需生成结合子用于任意应用。
通过最大程度地减少人工干预,BindCraft 有效降低了高质量蛋白结合子设计的门槛,让蛋白质设计这一曾高度专业化的任务,得以在更广泛的科研群体中普及与应用。
3 靶向细胞表面受体的结合子设计与验证
为验证所构建设计流程的性能,研究者设计了针对多种治疗相关的细胞表面受体的结合子,并在体外测试其结合活性。首先靶向的是人类 PD-1 蛋白,这是一种表达于 T 细胞表面的关键免疫检查点受体。共纯化并筛选了 53 个结合子设计,采用二价 Fc 融合构建体,通过生物层干涉法(BLI)测试其结合能力,结果有 13 个显示出结合信号,其中最优设计的表观解离常数
受此结果鼓舞,为减少实验筛选量,后续靶点仅测试较少设计。随后设计了针对 PD-L1 与干扰素 2 受体(IFNAR2)的结合子,两者均为重要的免疫信号调控因子。针对 PD-L1 的 9 个设计中有 7 个显示出结合信号(Supplementary Fig. 3a),而在 9 个 IFNAR2 设计中有 3 个表现出结合能力(Fig. 1b)。PD-L1 最优结合子 binder4 的
IFNAR2 的最优结合子 binder5 在 SPR 中表现出 260 nM 的亲和力(Fig. 2e,f),具有典型 α 螺旋信号、高稳定性(Supplementary Fig. 3c)以及单体性质(Extended Data Fig. 2e)。将其与 IFNAR2 的天然配体 IFNA2 测试结合竞争能力,结果显示其确实占据 IFNA2 的结合位点(Extended Data Fig. 2f),但主要识别不同区域(Extended Data Fig. 2g)。为评估设计的特异性,还将各最优结合子与其他具有免疫球蛋白样折叠的受体进行测试,尽管这些蛋白结构相似(Supplementary Data 2),但未发现脱靶结合现象(Extended Data Fig. 2h),且 AF2 的 i_pTM 指标能有效区分靶向与非靶向结合。
这些结果表明,借助计算设计流程,可以直接获得具纳摩尔亲和力的结合子,无需大量筛选步骤,尤其是在靶点结合位点已知的前提下。为进一步验证该流程是否适用于缺乏明确结合位点信息的细胞外受体,研究者选择结构复杂的 CD45 为靶点,其胞外结构域包含四个免疫球蛋白样结构(d1–d4),在最小亚型中存在高度糖基化现象。共测试了 16 个结合子设计,其中有 4 个显示出结合信号(Fig. 1b)。最优结合子 binder1 的

图 2|靶向细胞表面受体的结合子设计与功能验证:a,结合子 binder2 与 PD-1 的复合物设计模型;b,代表性的 BLI 传感图,显示 binder2(二价 Fc 融合形式)与 PD-1 的结合动力学;c,结合子 binder4 与 PD-L1 的复合物设计模型;d,SPR 测定 PD-L1 与 binder4 之间的结合亲和力;e,结合子 binder5 与 IFNAR2 的复合物设计模型;f,SPR 测定 IFNAR2 与 binder5 的结合亲和力;g,结合子 binder1 与 CD45 的复合物设计模型;h,SPR 拟合获得 binder1 的结合亲和力;i,CpE 毒性机制及其被 CLDN1 结合子抑制的示意图;j,CLDN1 结合子 binder12 与可溶性 CLDN1 模拟物之间的 SPR 单周期动力学分析;k,细胞实验显示 binder9、binder12 及 CpE 抑制剂对 CpE 毒性的浓度依赖性抑制效果,柱状图表示两个重复实验的均值,误差棒表示标准差;l,MST 测定显示在与 binder12 预孵育后,CpE 与 CLDN1 野生型的结合被阻断,所示数据来自一次代表性实验;图 i 使用 BioRender (https://biorender.com) 绘制。
4 靶向未探索结合位点的结合子设计
膜蛋白在生物学与治疗中具有重要意义,尤其是那些缺乏明确胞外结构域的蛋白。然而,这类靶点在结合子设计中尤具挑战性,因其难以进行实验验证与高通量筛选。通过计算设计生成的保留天然表位的可溶性模拟物提供了一种前景可期的解决方案,能够用于潜在结合子的快速预筛。为验证此策略,研究者选择了claudin 蛋白家族作为目标,这类蛋白对维持上皮与内皮紧密连接屏障至关重要。claudin 蛋白可被**产气荚膜梭菌肠毒素(CpE)**特异性靶向,CpE 会在膜上形成穿透孔洞,最终导致细胞死亡。
研究者提出,若能设计出与 CpE 竞争相同结合位点的结合子,则可减轻其毒性作用(见 Fig. 2i)。基于 claudin-1 的可溶性模拟物(sCLDN1),设计了一批结合子,靶向其胞外区域,并使用两种模拟物变体进行结合预筛(Supplementary Fig. 4a,b)。在测试的 7 个结合子中,除 binder17 外,其余均能结合含有 CLDN1 原始表位的 sCLDN1-14 与 sCLDN1-18(Supplementary Fig. 4a),其中binder12 显示出最强的结合信号,并表现出纳摩尔级亲和力(Fig. 2j)。
进一步在 CLDN1 野生型(WT)背景下,采用细胞毒性实验评估其功能。结果显示,binder9 与 binder12 能有效抑制 CpE 介导的细胞毒性,并对表达 CLDN1 WT 的细胞起到浓度依赖性的保护作用,其效果与已知 CpE 抑制剂相当(Fig. 2k 和 Supplementary Fig. 4c)。值得注意的是,这两个结合子源自同一设计路径,且具有相同的界面残基。为验证其作用确实是通过与 CLDN1 WT 的直接结合实现的,研究者采用微量热泳动技术(MST),发现 CpE 与 binder12 均可结合 CLDN1 WT,且在 binder12 与 CLDN1 WT 预孵育后,CpE 的结合被明显阻断,表明存在竞争关系(Fig. 2l 和 Extended Data Fig. 2i)。然而,这些结合子无法保护表达 CLDN4 的细胞免于 CpE 毒性(Supplementary Fig. 4d,e),可能因 CpE 对 CLDN4 的亲和力约为 CLDN1 的 400 倍。
这些结果表明,利用可溶性模拟物可有效发现可调节膜蛋白功能的结合子。为评估该设计流程是否具备通用性,研究者进一步选择了一个无天然序列同源物的蛋白作为靶点 —— 从头设计的 β-桶蛋白 BBF-14。β-桶结构通常不被视为蛋白互作的结构基础。研究者从高评分设计中纯化出 11 个结合子,其中 6 个显示结合能力(Fig. 1b)。最优结合子 binder4 由混合 α-β 拓扑构成,结合界面由分裂的 β 折叠片和螺旋元件共同形成。其中 β 折叠的界面结合并不依赖主链氢键,而是由侧链相互作用维系。SPR 测得其对 BBF-14 的
为验证设计的准确性,研究者解析了 BBF-14 与 binder4 的复合物结构(Extended Data Fig. 3c,d;Extended Data Table 1)。将结构对齐至 BBF-14 后,binder4 的主链 Cα 原子均方根偏差(r.m.s.d.)为 1.7 Å,确认其折叠结构与设计结合模式均高度准确(Extended Data Fig. 3c)。该结果强调了仅基于结构信息(而非已知结合位点或协同进化数据)亦可成功设计高亲和结合子。
最后,研究者选择进化中保守的结构蛋白 SAS-6 作为设计靶点,SAS-6 寡聚体是真核细胞中中心粒生物合成所必需的。使用 BindCraft 设计多个结合子,并实验测试了评分最高的 9 个。binder4 对 SAS-6 单体的亲和力为 5.7 μM,对二聚体的亲和力为 4.2 μM(Extended Data Fig. 3e–g),表明其可与寡聚形式兼容。该结合子靶向的区域与已报道的单抗 MBCRS6-15 重叠(Extended Data Fig. 3h),而该抗体已知可诱导 SAS-6 从环状结构转变为螺旋结构。
研究者推测,BindCraft 能实现按需结合子设计,不仅适用于结构复杂或无已知表位的靶点,也可用于探索高阶蛋白复合体中的功能机制。
5 掩蔽变应原表位的结合子设计
变应性鼻炎和季节性过敏在部分国家中影响着多达 50% 的人群。当前治疗手段主要集中在通过免疫抑制剂和单克隆抗体抑制全身炎症反应。然而,中和过敏反应有望成为更有效的管理策略。变应原通常是具有不同折叠方式、生物功能和高电荷表面的多样蛋白。由于水疏性结合位点更适合计算设计,因此高电荷表面的变应原更具挑战性。
为测试 BindCraft 靶向变应原的能力,研究者设计了针对尘螨变应原 Der f7、Der f21,以及主要的白桦花粉变应原 Bet v1 的结合子,后者与多达 95% 的白桦相关过敏病例相关。共对 Der f7 测试了 10 个设计,发现其中 4 个结合子具有结合能力(Fig. 1b),其中binder2 显示最高亲和力,
类似地,对 Der f21 测试了 7 个结合子,其中 4 个显示结合能力(Fig. 1b),其中表现最佳的 binder10 表观亲和力为 793 nM(Fig. 3c)。尽管在溶液中为二聚体(Extended Data Fig. 4d),其 2.6 Å 分辨率的晶体结构验证了其以 1:1 模式结合 Der f21 表面一个带有高电荷的螺旋区域(Extended Data Fig. 4e)。该结合子的 r.m.s.d.Cα 为 3.1 Å,主要由界面酪氨酸残基的不同旋转异构体构象引起(Fig. 3d)。Der f21 的突变分析显示这些结合子识别的表位与过敏者血清中 IgE 所识别的区域不同。
对于白桦变应原 Bet v1,测试了 7 个设计,发现其中两个具有结合能力(Fig. 1b),binder2 的亲和力为 120 nM(SPR 测定)(Fig. 3e),在溶液中呈二聚体(Extended Data Fig. 4f),但其与 Bet v1 的复合物质量为 27.8 kDa,表明为 1:1 结合模式(Fig. 3f)。该结合子具有扭曲的螺旋拓扑结构,其 C 端螺旋插入 Bet v1 的配体结合口袋(文献 28)。为评估其特异性,研究者将每个最优结合子分别与三种变应原孵育,即便在 10 μM 浓度下,亦未观察到交叉结合(Extended Data Fig. 4g),显示其具高度特异性。
此前已有研究开发了结合 Bet v1 三个免疫原性表位的抗体混合物,用于阻断过敏反应(文献 29)。其冷冻电镜(cryo-EM)结构显示,该研究的 binder2 靶向已知由 REGN5713 抗体识别的表位(Fig. 3g)。为验证此结果,将 REGN5713 固定在 SPR 芯片上加载 Bet v1 变应原,检测发现 REGN5714 可结合,而 binder2 不可结合,进一步证实其与 REGN5713 靶向相同表位(Fig. 3h)。
最后,为验证 binder2 是否能与患者血清中的抗 Bet v1 IgE 竞争结合位点,研究者在三名对白桦过敏、具有高效价抗 Bet v1 IgE 的患者血清中进行阻断型酶联免疫吸附实验(blocking ELISA)。用生物素标记的 Bet v1 与 REGN 抗体混合物或设计的 binder2 预孵育(Fig. 3i),结果显示 REGN 抗体可阻断高达 90% 的 IgE 结合,而 binder2 在其中两位供体中可阻断达 50%,与单抗阻断效率相当,表明从头设计的结合子在中和过敏反应方面具有治疗潜力。
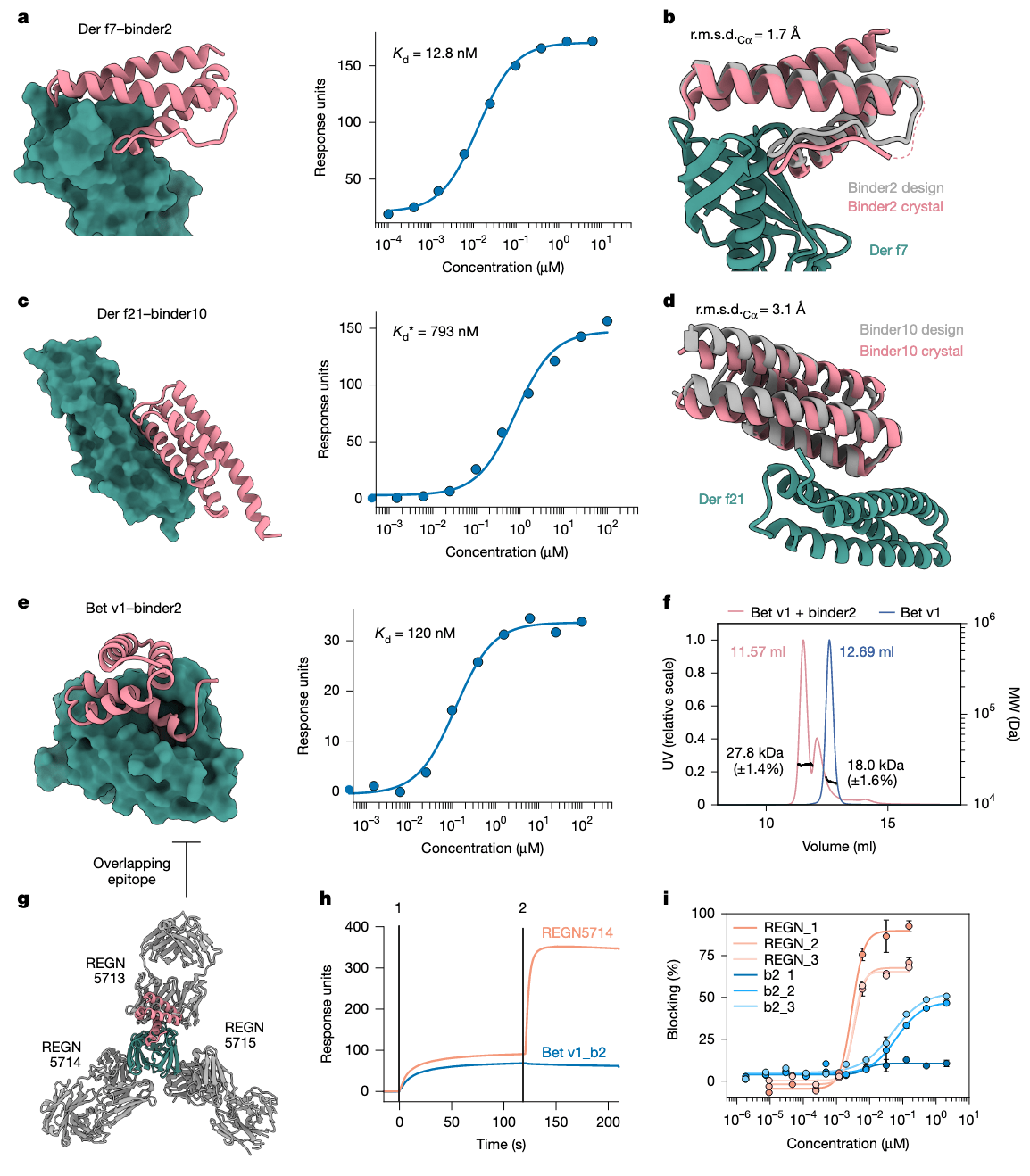
图 3|遮蔽常见变应原表位的结合子设计与验证:a,左图:binder2 针对尘螨变应原 Der f7 的设计模型;右图:binder2 的 SPR 结合亲和力拟合曲线;b,Der f7–binder2 复合物晶体结构(彩色)与设计模型(灰色)叠合;c,左图:binder10 针对尘螨变应原 Der f21 的设计模型;右图:binder10 的 SPR 结合亲和力拟合曲线;d,Der f21–binder10 复合物晶体结构(彩色)与设计模型(灰色)叠合;e,左图:binder2 针对白桦变应原 Bet v1 的设计模型;右图:binder2 的 SPR 结合亲和力拟合曲线;f,SEC–MALS 分析 Bet v1(蓝色,预期分子量 18.5 kDa)与 Bet v1 和 binder2 混合物(橙色,预期分子量 29.3 kDa);g,Bet v1 与商业抗体混合物 REGN 结合的冷冻电镜结构(PDB 7MXL);h,竞争结合实验显示 REGN5714 能结合已固定的 REGN5713–Bet v1 复合物,而 binder2 无法结合,验证其结合位点重合;i,阻断型 ELISA 检测 REGN 抗体混合物(橙色)或 binder2(蓝色)阻断 Bet v1 与来自三名白桦过敏患者血清中 IgE 结合的能力,数字后缀表示个体患者血清编号,数据点为两个技术重复的平均值,误差棒表示标准差。
6 调控多结构域核酸酶的结合子设计
由于核酸结合界面通常具有大面积、高电荷且凸出的表面结构,难以被小分子药物有效靶向,因此长期以来被认为是“难以药物化”的靶点。为验证设计流程能否适用于此类界面,研究者聚焦于链球菌来源的多结构域 CRISPR–Cas9 核酸酶(SpCas9)。尽管 SpCas9 已广泛用于基因编辑(因其良好的可编程性),但其最初在细菌中充当对抗噬菌体的免疫防御系统。而噬菌体也进化出抗 CRISPR 蛋白(Acr),通过阻断核酸结合位点抑制 Cas 功能。
研究者设想是否可以设计人工 Acr 蛋白来模拟这一功能。设计工作聚焦于 SpCas9 的 REC1 结构域,该结构域包含一个高电荷的向导 RNA 结合口袋(Fig. 4a)。测试的 6 个结合子中全部能够结合 SpCas9 蛋白的全长 apo 构象(Supplementary Fig. 5a)。其中表现最优的 binder3 和 binder10 在 SPR 中表现出约 300 nM 的表观亲和力,尽管受限于实验条件难以获得完整的滴定曲线。
为验证其结合模式,研究者尝试解析 binder3 与 binder10 与 SpCas9 apo 酶的 cryo-EM 复合物结构。尽管原始数据质量较高,结合子在密度图中也可清晰识别(Extended Data Fig. 5a),但由于靶点区域分辨率较差,未能建立原子模型(Extended Data Fig. 5b),可能是 apo Cas9 构象动态性强的结果。然而,在 REC1 区域仍能观察到明确密度,两个结合子均可稳定嵌入设计位点,验证了其结合模式(Fig. 4b,c 和 Extended Data Fig. 5c,d)。
进一步在 HEK293T 细胞中共转染 SpCas9 系统与设计的结合子或天然 Acrs,发现设计的结合子可显著降低基因编辑效率(Fig. 4d)。其抑制效果优于 AcrIIC2(后者通过阻碍向导 RNA 加载实现作用机制),但不及作用于靶 DNA 结合的 AcrIIA2 与 AcrIIA4,这两者几乎完全阻断编辑活性(Supplementary Fig. 5b)。这表明各 Acr 的抑制策略差异显著,也说明 BindCraft 可设计出具有全新机制的 Cas9 抑制子。
为拓展到其他大型核酸酶,研究者又设计了针对**Clostridium butyricum 的多结构域 Argonaute 核酸酶(CbAgo)**的结合子。与 Cas9 类似,CbAgo 利用小寡核苷酸引导靶向切割入侵 DNA。迄今尚无 CbAgo 的天然抑制因子被报道。
研究者设计结合子靶向 CbAgo 的 N-PIWI 通道或 PAZ 结构域(Fig. 4e),并测试其对 CbAgo 介导的靶 DNA 切割活性的影响。在测试的 12 个结合子中,有两个结合子 显著抑制 CbAgo 活性(Fig. 4f)。在单独使用 0.4 μM CbAgo 时,其催化速率常数(
通过 SEC 分析验证 binder2 可与 CbAgo 形成稳定复合物(Extended Data Fig. 5f)。引入 guide DNA(gDNA)后该复合物不稳定,说明 binder2 占据了 gDNA 的结合通道(Extended Data Fig. 5g,h)。
综上,以上结果表明,BindCraft 可用于设计靶向复杂核酸结合界面与沟槽的蛋白结合子,为开发新型蛋白抑制剂、基因编辑调控器及基础研究工具开辟了新路径。
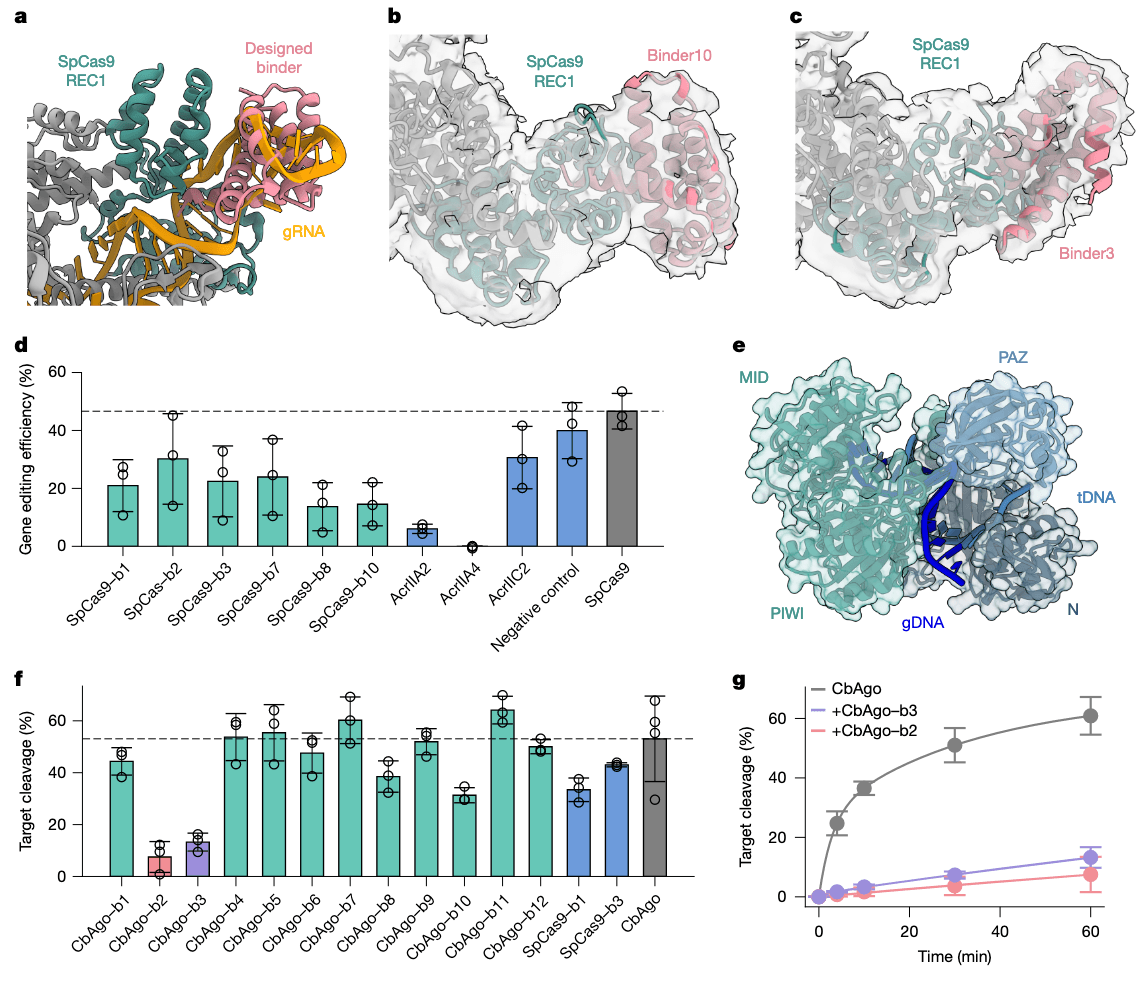
图 4|利用从头设计的结合子靶向核酸导向的多结构域核酸酶的核酸互作界面:a,放大展示 SpCas9 的 REC1 结构域及其与 guide RNA 的结合状态(PDB 4ZT0),设计的结合子叠加在其结合口袋中;b,binder3 与 SpCas9 apo 构象结合的冷冻电镜结构,REC1 结构域以绿色高亮,其余 SpCas9 以灰色表示,叠加灰色密度图显示 cryo-EM 密度;c,binder10 与 SpCas9 apo 构象结合的 cryo-EM 结构,标注方式同 b;d,在无结合子(灰色柱,虚线)或加入设计结合子(绿色柱)或天然 Acr(蓝色柱)条件下,SpCas9 在 HEK293T 细胞中的基因编辑活性比较;e,Clostridium butyricum Argonaute 核酸酶与 gDNA 和 tDNA 的复合物结构(PDB 6QZK),PAZ 结构域与 N+PIWI 结构域分别以浅蓝与深蓝高亮,作为结合子设计靶点;f,在无结合子(灰色柱,虚线)或添加设计结合子(绿色柱)或 Cas9 结合子(蓝色柱)条件下,CbAgo-gDNA 介导的目标 DNA 切割活性;柱状图表示三次重复实验的平均值,误差棒表示标准差;g,在无结合子(灰线)或加入设计结合子 binder2(粉线)、binder3(紫线)条件下,CbAgo-gDNA 介导的 DNA 切割曲线,数据点为三次测量的平均值,误差棒为标准差。
7 用于基因递送的 AAV 靶向重定向设计
腺相关病毒(AAV)衍生的病毒载体因其天然将遗传物质递送至细胞和组织的能力,极大拓展了基因治疗的应用前景。然而,AAV 在细胞类型、组织乃至器官层面的靶向性较差,通常需要高剂量使用才能达到治疗效果,这也提高了脱靶风险与免疫原性。已有多种策略尝试通过插入多肽片段或受体结合元件(如 DARPins)来改变 AAV 衣壳的嗜性(tropism),但这些方法通常依赖文库筛选或免疫接种实验,对靶点位点的控制较弱。
该研究提出,BindCraft 可高效设计微型蛋白结合子,实现 AAV 对细胞特异性受体的重定向(Fig. 5a)。得益于其高设计成功率,这些结合子有望直接用于细胞内的 AAV 转导测试,绕过传统的生化预筛步骤,为快速开发靶向特定细胞或组织的 AAV 载体提供新平台。
传统的 AAV 重定向方法通常将功能分子插入至衣壳蛋白三聚轴附近的 VR-IV 或 VR-VIII 变异区,或融合于病毒衣壳蛋白 VP2 的 N 端。该研究基于大规模 AAV 衣壳功能突变研究,探索了一个替代插入位点 —— 位于 AAV 衣壳 VR-V 区域的第 497 和 498 位氨基酸之间,靠近三聚对称轴(Fig. 5b)。以 AAV6-cmv-GFP 为起始载体,引入点突变以消除其对肝素和唾液酸的天然结合能力(Fig. 5a 中标注为 knockout)。随后设计了针对 HER2 与 PD-L1 的结合子,结合子经过 N 末与 C 末距离惩罚(distance loss)优化,以便实现更好地嵌入衣壳结构,两端均加入短的 –(GSG)₁– 延伸(Fig. 5b)。
为了同时评估设计的 AAV 在产量和转导效率上的表现,开发了一种小规模并行筛选体系,直接将包装细胞的上清液加入到靶细胞中进行测试(Extended Data Fig. 6a,b)。该方法识别出一个可靶向 HER2 的重编程 AAV,以及四个能靶向 PD-L1 的 AAV 变体,它们均对稳定表达相应受体的 HEK293 细胞表现出增强的特异性(Fig. 5c 和 Extended Data Fig. 6c)。
对两个最优变体 HER2–b1 和 PD-L1–b202 进行表征(Fig. 5d,e),发现它们在转导表达目标受体的细胞时具有明显增强的特异性(Fig. 5f)。当利用抗体阻断其靶向的受体结合位点时,PD-L1 靶向型 AAV 的转导能力也被抑制,说明其转导依赖于所设计的结合子与目标受体的结合(Fig. 5g)。
这些结果表明,BindCraft 可实现 AAV 向特定细胞类型的高效靶向重定向,并为病毒载体的精准基因递送提供了创新设计思路。
图 5|通过 AAV 实现靶向基因递送的工程设计:a,示意图展示将细胞类型特异性受体的微型蛋白结合子插入 AAV-cmv-GFP 衣壳中,从而实现靶向重定向,替代其对细胞表面糖类的天然结合方式;b,靶向重定向 AAV 粒子的嵌合组装模型,展示带有(粉色)和不带有(绿色)插入结合子的衣壳蛋白以特定化学计量比组合;c,将包装细胞上清液转移至稳定表达 HER2 或 PD-L1 的 HEK293 细胞后,使用流式细胞术检测不同 AAV 变体的转导效率。信噪比表示为靶细胞与非靶细胞转导效率的“× 倍”变化。对比组为野生型 AAV6-cmv-GFP(WT)与带有敲除突变(KO)的衣壳变体;每组测量进行三次重复(n = 3),误差棒表示 95% 置信区间;d,binder1 靶向 HER2 的设计模型;e,binder202 靶向 PD-L1 的设计模型;f,在标准感染量(MOI)1 × 10⁵ vg/cell 下,带有 binder1 或 binder202 的 AAV 变体在各自稳定过表达靶受体的 HEK293 细胞中的转导率热图,同时展示 KO 与 WT 对照;g,携带 binder202 的 AAV 转导稳定表达 PD-L1 的 HEK293 细胞的情况,下方柱状图显示靶向 binder202 结合位点的抗 PD-L1 抗体可阻断该转导过程。图 a 使用 BioRender (https://biorender.com) 绘制。
8 结论
从头设计蛋白质–蛋白质相互作用(PPI)一直是蛋白设计领域中的核心难题,主要原因在于对分子识别决定因素的理解仍不充分。随着基于深度学习的结构预测网络(如 AF2)的发展,该领域迎来了变革,极大提升了对具备理想生化特征的从头设计蛋白的筛选准确性。
该研究介绍了一种基于反向传播穿越 AF2 网络的稳健设计流程,该策略在先前研究中已有探索,并在该研究中被进一步扩展以用于蛋白结合子的幻觉生成(hallucination)。与现有大多数方法不同,BindCraft 允许目标蛋白结构的柔性变化,这对捕捉结合诱导的构象变化至关重要,是实现有效分子识别的关键。
研究者在 12 个具有挑战性的靶点上系统评估了 BindCraft 的表现,设计出的结合子普遍具有纳摩尔级亲和力,成功率从 10% 到 100% 不等,平均成功率达 46.3%,对于一个纯计算流程而言,这一结果相当显著。与现有先进方法(如 RFdiffusion)和近期发布但闭源的 AlphaProteo 结合子设计流程相比,BindCraft 所需的实验筛选量更低。
值得一提的是,BindCraft 设计的一款结合子近期在全球性结合子设计竞赛中获得第一名,其对挑战性的 EGFR 靶点展现出 82 nM 的亲和力。这不仅体现了结构设计的准确性,也证明该方法可以用于功能化蛋白的构建。例如,研究者利用设计的结合子显著抑制了白桦变应原 Bet v1 与患者血清中特异 IgE 的结合。虽然单一结合子的阻断效果中等,但通过覆盖更广泛的表位或可进一步增强疗效。得益于其高稳定性,从头设计的结合子在治疗应用中有望成为抗体的替代方案。
然而,由于结合子为人工合成且较大(60–240 个氨基酸),其免疫原性与递送效率仍是潜在问题,尽管已有临床前模型正在逐步解决这些挑战。此外,BindCraft 高实验成功率使得设计蛋白可直接用于目标功能的筛选,例如该研究中 AAV 的靶向重定向,即展示了该平台在特异性病毒载体开发中的应用潜力,有望用于实现疾病相关细胞或组织的精准基因递送,同时降低脱靶风险。
尽管成果显著,BindCraft 仍存在一些局限性。首先,反向传播穿越 AF2 的过程对 GPU 资源消耗巨大;其次,使用 AF2 单体模式对设计结果进行筛选时,可能会误剔某些高亲和结合子。此外,虽然尝试使用新版 AlphaFold3 进行筛选,但其预测结果仍存在大量假阳性(Extended Data Fig. 7c)。AF2 本身对点突变的敏感性较低,这在 PPI 设计中可能带来问题,但可通过如 Rosetta 等正交物理评分方法缓解。最后,BindCraft 使用 AF2 的 i_pTM 指标进行排序,尽管该指标已成为有效的二分类预测结合活性的工具(Extended Data Fig. 7a,b),但其与结合亲和力不具显著相关性(Supplementary Fig. 6)。
尽管如此,BindCraft 依然代表了在精确结合子功能化设计方面的重要飞跃。作者相信,通过不断迭代优化这一流程,未来将实现“一设计即一成功结合子”的目标,彻底摆脱实验筛选需求。这将使蛋白结合子的快速生成成为可能,广泛应用于科研、生物技术与治疗等领域,即使使用者不具备蛋白设计背景也能高效开展相关研究。