bioRxiv 2024 | Boltz-1: 通用生物分子相互作用建模

Boltz-1 是首个开源且商业可用的通用生物分子复合物结构预测模型,具备与 AlphaFold3 同等级别的精度。通过创新的多序列比对算法和推理引导技术 Boltz-steering,提升了预测的准确性与物理合理性。Boltz-1 及其扩展版本 Boltz-1x 在多项基准测试中表现出色,推动了生物分子结构预测的开放与进步。
获取详情及资源:
- 论文:Wohlwend, Jeremy, Gabriele Corso, Saro Passaro, et al. “Boltz-1 Democratizing Biomolecular Interaction Modeling.” bioRxiv, March 6, 2025. https://doi.org/10.1101/2024.11.19.624167.
- 代码:https://github.com/jwohlwend/boltz

0 摘要
生物分子相互作用的理解是药物发现和蛋白质设计的重要基础。Boltz-1是一款开源深度学习模型,结合了创新的架构设计、速度优化和数据处理技术,实现了与AlphaFold3相当的生物分子复合物三维结构预测精度。在多个标准基准测试中表现优异,达到先进商业模型水平。Boltz-steering是一种推理引导技术,能有效修正模型中的非物理和错误预测。通过公开训练代码、模型权重及数据集,Boltz-1为生物分子建模领域提供了高效开放的平台,促进全球科研协作与进展。
1 引言
生物分子相互作用驱动着几乎所有的生物机制,对这些相互作用的理解推动了新疗法的开发和疾病机制的发现。2020年,AlphaFold2展示了深度学习模型在大规模蛋白序列的单链结构预测中达到了实验精度。然而,如何在三维空间建模生物分子复合物仍是一个重要难题。近年来,研究界在这一领域取得了显著进展,特别是深度生成模型在模拟不同生物分子相互作用方面表现出色,例如DiffDock相较传统分子对接方法有显著提升,最新的AlphaFold3则在任意生物分子复合物结构预测中达到了前所未有的精度。本文介绍了Boltz-1,这是首个完全开放且商业可用的模型,达到了AlphaFold3报告的精度水平。通过在MIT许可证下免费发布训练和推理代码、模型权重、数据集和基准测试,Boltz-1为全球科研人员和开发者提供了强大的工具,促进实验验证和创新。整体上,Boltz-1遵循了Abramson等人提出的架构框架,同时引入了多项创新改进,包括:
- 引入了新算法,更高效且稳健地配对多序列比对(MSA),在训练时裁剪结构,并支持基于用户定义的结合口袋进行条件预测;
- 优化了模型架构中表示的流动方式以及扩散训练和推理过程;
- 重新设计了置信度模型,不仅在架构组件上进行了改进,还将任务重新定义为对模型主干层的微调。
接下来的章节将详细介绍这些改进内容,并与其他公开模型进行性能对比。实验结果表明,Boltz-1在多种结构和评测指标上与先进商业模型表现相当。此外,针对基于机器学习的结构预测方法常见的不符合物理规律的问题——如内部几何结构错误、手性问题、空间阻碍以及链重叠等“幻觉”现象——提出了一种推理时引导技术Boltz-steering,几乎解决了所有这类物理性问题,同时保持了模型准确性。采用该技术的模型称为Boltz-1x。作为一个动态更新的开源项目,本文及其关联的GitHub仓库将持续由核心团队和社区贡献者进行改进。该项目及其代码库旨在推动对生物分子相互作用的理解,并促进新型生物分子的设计。
2 数据处理流程
Boltz-1使用的蛋白质以氨基酸序列表示,配体以SMILES字符串(及共价键)表示,核酸则以基因组序列表示。输入数据进一步通过多序列比对(MSA)和预测的分子构象进行增强。与AlphaFold3不同,Boltz-1未使用输入模板,因其对大型模型性能影响有限。
本节首先介绍结构训练数据及MSA与构象数据的获取过程,并说明验证集和测试集的整理方法。随后重点描述三项关键的数据整理和增强算法创新:
- 利用分类学信息对多聚体蛋白复合物的MSA进行配对的新算法(见2.3节);
- 结合先前研究中空间裁剪和连续裁剪策略的统一裁剪算法(见2.4节);
- 针对常见应用场景设计的鲁棒结合口袋条件化算法(见2.5节)。
2.1 数据来源与处理
训练数据选用截至2021年9月30日发布的所有PDB结构(与AlphaFold3相同的训练截止日期),且分辨率至少达到9Å。通过解析结构的mmCIF文件,提取生物组装体1。对于每条聚合物链,使用参考序列并与结构中的残基进行比对。配体部分则利用CCD字典生成构象,并匹配结构中的原子。在配体共价结合且结构中未出现该原子的情况下,移除该原子。最后,按照AlphaFold3的数据清洗流程执行,包括排除指定配体列表、确保残基数量最小阈值以及移除冲突链条。
多序列比对(MSA)与分子构象体的构建过程,以及蛋白质结构预测模型的训练流程。针对整个PDB数据集,采用ColabFold搜索工具进行MSA构建,该工具基于MMseqs2实现,使用默认参数(版本信息包括uniref30 2302和colabfold envdb 202108)。随后,通过UniProt提供的分类注释,对所有UniRef序列进行了分类标签的赋予。对于模型输入的初始分子构象体,预先使用RDKit的ETKDGv3方法针对所有CCD编码计算了单一的构象体。
在结构预测模型的训练过程中,模型共训练了68,000步,批量大小设为128。训练的前53,000步中,使用了384个token和3456个原子的裁剪尺寸,训练数据均匀采自PDB数据集和OpenFold蒸馏数据集(后者约包含27万个结构,且使用了其提供的MSA)。训练后15,000步则仅采样自PDB结构,裁剪尺寸增加到512个token和4608个原子。与AlphaFold3相比,其采用了类似的网络架构,但训练步数达到近150,000步,批量大小为256,计算时间约为本文方法的四倍。这一显著的计算效率提升归功于本文在后续部分详细介绍的多项创新技术。
2.2 验证集与测试集的构建
针对当前缺乏统一的全原子结构标准测试集问题,发布了一套全新的PDB数据划分方案,旨在帮助学术界形成稳定且一致的全原子结构预测基准。该训练、验证和测试集划分策略主要借鉴了Abramson等人的方法。
首先,使用mmseqs的easy-cluster命令基于序列相似度(最低40%序列同一性)对PDB中的蛋白质序列进行聚类。接着,从PDB中筛选符合以下条件的结构:初次发布于2021年9月30日之前(不含)且2023年1月13日(含)之前,解析度低于4.5Å,所有链的蛋白质序列均不属于任何训练集中的聚类(即2021年9月30日之前的序列聚类),且小分子部分要么不存在,要么至少存在一个小分子与训练集中的小分子Tanimoto相似度不超过0.8(小分子定义为含有两个以上重原子且不在配体排除名单中的非聚合物实体)。经过此步筛选,共得到1728个结构。
对这批结构进一步筛选细化,步骤包括:保留含有RNA或DNA的结构(126个);迭代添加含有小分子或离子的结构,条件是其蛋白链属于未见过的新聚类(新增330个);迭代添加多聚体结构,且蛋白链同样属于新聚类,之后随机保留通过筛选结构的50%(新增231个);迭代添加单体结构,链属于新聚类,随机保留通过结构的30%(新增57个)。最终汇总得到744个结构。
最终保留蛋白质、RNA、DNA链总残基数不超过1024的结构,形成553个样本的验证集。
测试集采用相似流程构建,区别在于蛋白质和配体的相似性排除考虑了2023年1月13日之前发布的所有结构(涵盖训练和验证集),筛选条件为发布时间晚于2023年1月13日,且总残基数在100至2000之间。最终测试集包含593个结构。
2.3 密集型多序列比对配对算法
多序列比对(MSA)能够揭示在进化过程中共进化的氨基酸残基,这些残基通常在三维空间中相距较近。然而,针对蛋白质-蛋白质相互作用提取此类信号更具挑战性,因为大多数蛋白质序列通常是单独测序或报告的。为近似配对这些序列,研究人员常利用序列所关联的分类学信息。
文中提出的算法3介绍了一种基于分类学信息进行MSA配对的方法,该方法旨在保持MSA的密度(密度对于模型复杂度极为关键,因为模型复杂度随MSA行数线性增长),同时在配对序列所带来的信号强度与单个链内的序列冗余之间实现合理的平衡。
2.4 统一裁剪算法
为有效训练不同大小的蛋白质复合体,诸如AlphaFold2和AlphaFold3等方法通常在训练时对结构进行裁剪,限定最大原子数、残基数或token数。常见的裁剪技术主要有两种:一种是连续裁剪,即选择序列中连续的残基或完整分子片段;另一种是空间裁剪,根据与中心token的空间距离选择token。这两种方法各有优点,能够为模型提供不同的训练信号,因此在实际应用中往往结合使用,比如Abramson等人的工作。
然而,文中指出这两种方法代表了裁剪策略的两个极端,训练中引入更多样化的裁剪策略更为有效。基于此,提出了一种新的裁剪算法(算法4),该算法通过定义“邻域”来实现连续裁剪与空间裁剪的直接插值。邻域指的是围绕特定token的序列中一定长度的连续片段(或完整的非聚合物实体)。裁剪时,依据邻域中心token与裁剪中心的距离逐步加入邻域。如果邻域大小设为零,该方法即为纯空间裁剪;若邻域大小为最大token预算的一半,则等同于连续裁剪。
实验表明,随机均匀采样邻域大小(范围从0到40个token)作为训练样本的裁剪参数,能够带来更好的训练效果。
2.5 鲁棒的结合位点条件化
在许多实际应用中,研究人员通常对蛋白质的结合位点已有一定的先验知识,因此使模型能够基于结合位点信息进行条件化建模非常有价值。AlphaFold3通过微调模型,新增一个针对所有结合位点-配体对的token特征实现了结合位点条件化,其中结合位点定义为距离配体6Å内的所有残基。这种方法虽然有效,但存在一些限制:需要维护两个模型(有条件化和无条件化版本),且假设需明确指定所有距离配体6Å内的残基,而现实场景中用户往往只知道关键残基,且完整的相互作用残基集合高度依赖于配体构象,常常无法准确获知。
为解决上述问题,提出了一种不同的结合位点条件化策略,设计目标包括:(1)只需维护一个统一模型;(2)对部分指定的相互作用残基具有鲁棒性;(3)支持针对蛋白质或核酸等聚合物结合剂的作用位点指定。在训练过程中,30%的迭代随机选择一个结合剂,依据几何分布随机决定暴露的结合位点残基数目,并从距离结合剂6Å内至少含一个重原子的残基中随机抽取。该信息被编码为额外的one-hot token特征输入模型。此训练流程在算法5中有详细描述。
3 建模
模型架构与训练基于Abramson等人补充材料中描述的AlphaFold3进行了复现。AlphaFold3是一种扩散模型,采用多分辨率的基于Transformer的网络对原子坐标进行去噪处理。模型在两个分辨率层级上运行:重原子层和token层。token的定义因分子类型而异,蛋白质链中为氨基酸,RNA和DNA中为核酸碱基,其他分子及修饰残基或碱基则为单个重原子。
去噪Transformer的核心之上,AlphaFold3设计了一个中央主干结构(central trunk),用于初始化token表示及确定Transformer的注意力对偏置(attention pair bias)。该主干结构计算开销较大,主要原因在于它以token对作为基础的“计算单元”,并对这些对进行轴向注意力操作,导致计算复杂度随着输入token数量的三次方增长。为降低计算负担,主干结构设计为与具体的扩散时间或输入结构无关,使得每个复合体仅需运行一次该部分。
在此基础架构之上,团队设计并测试了多种潜在的替代方案。以下章节将重点介绍那些带来性能提升并最终被纳入Boltz-1.2版本的改进。鉴于训练完整规模模型所需的巨大计算资源,这些改动主要在较小规模的模型架构上进行了验证。尽管未能呈现针对最终全尺寸模型的直接消融实验,但预计这些改进同样适用。
3.1 架构改进
MSA模块
对AlphaFold3中MSA模块(对应算法8)的操作顺序进行了调整,以便更有效地实现单体表示(single representation)与对表示(pair representation)之间的互相更新。具体而言,将原先的操作顺序:
OuterProductMean → PairWeightedAveraging → MSATransition → TriangleUpdates → PairTransition
修改为:
PairWeightedAveraging → MSATransition → OuterProductMean → TriangleUpdates → PairTransition。
其中,OuterProductMean负责将单体表示的信息传递到对表示,通过调整顺序,使得MSATransition中更新的单体表示能直接传递到对表示,从而增强信息流动。
Transformer层
Abramson等人描述的DiffusionTransformer层(对应算法23)中,隐藏表示的更新顺序为:
a ← AttentionPairBias(a) + ConditionedTransitionBlock(a)。
这种设计存在两个问题:一是缺少残差连接,可能导致反向传播难度增加;二是AttentionPairBias中学习的变换无法在同一层被ConditionedTransitionBlock利用。针对这些问题,调整为以下更新顺序:
a ← a + AttentionPairBias(a)
a ← a + ConditionedTransitionBlock(a)。
这种顺序引入了残差连接,改善了梯度传递,同时使得AttentionPairBias的输出能够直接作为ConditionedTransitionBlock的输入,提升了模型的表达能力。
3.2 训练与推理流程
Kabsch扩散插值
AlphaFold3相较于AlphaFold2的一个关键变化是其去噪模型不具备旋转和平移的等变性(AlphaFold2结构模块基于等变的IPA设计)。为了提升去噪模型对旋转和平移变换的鲁棒性,训练和推理时对输入结构随机进行平移和旋转操作。Abramson等人采用在计算均方误差(MSE)损失前,将预测的去噪坐标与真实坐标进行刚性对齐的做法,以降低去噪损失因这些变换产生的方差。
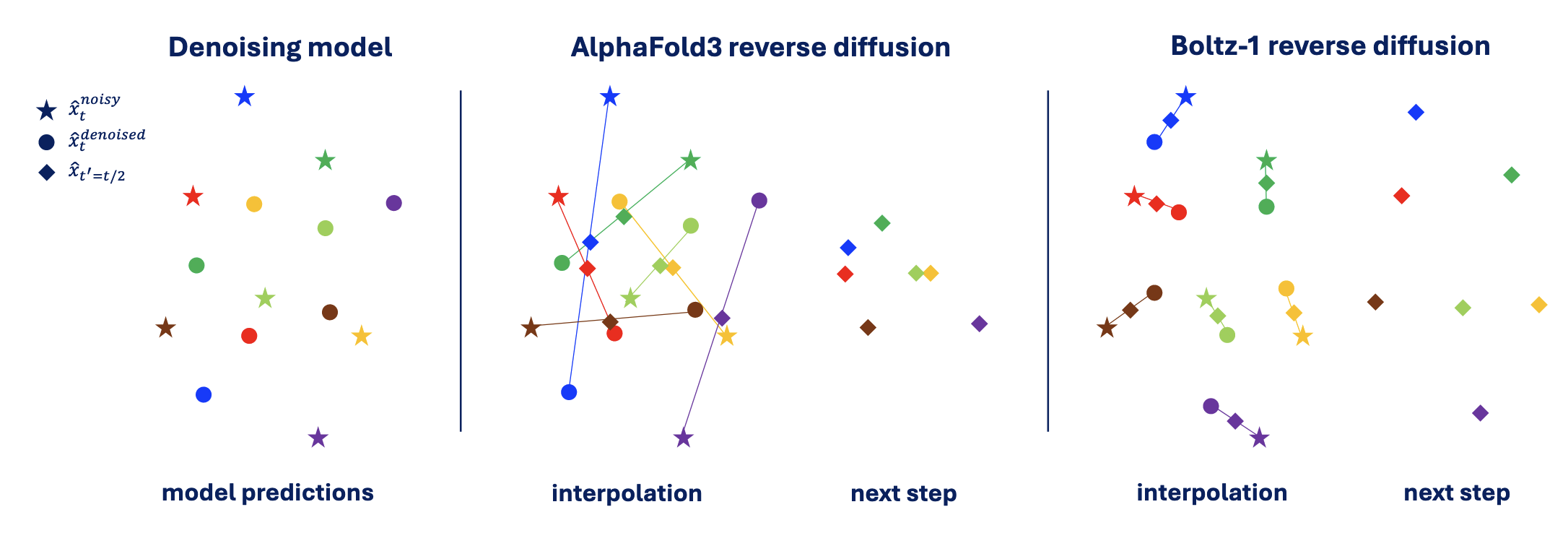
然而,仅靠这种方法存在理论上的问题。存在某些函数在训练时可使刚性对齐的MSE损失趋近于零,但在推理时却无法采样出真实合理的构象。例如,假设模型对任何合理范围内加噪(如噪声标准差10倍以内)的结构输入都预测相同的结构坐标x,当噪声超出此范围时则预测零向量。该模型训练时损失极低,但推理时因噪声变化导致预测不稳定,常常偏离真实分布,出现无效构象。这种现象源于去噪与噪声结构之间线性插值时,可能产生距离真实结构较远的中间状态。
为解决此问题,文中在推理过程的每一步插值前,利用Kabsch算法对噪声结构和去噪结构进行刚性对齐。这样,插值基于两者之间的最小距离投影,保证插值结构在理论假设下更接近去噪结果而非噪声输入。实验证明,该改进在训练子集数据时对防止过拟合尤为有效,但对于最终训练的完整Boltz-1模型,去噪输出已足够接近投影,Kabsch对齐的重要性有所减弱。
扩散损失权重
扩散损失的加权采用了符合EDM框架的表达式:
而非AlphaFold3中使用的:
这一调整更符合当前的理论分析,优化了损失函数的表现。
3.3 置信度模型
AlphaFold3在训练时将置信度模型与主干网络及去噪模型一同训练,但中断了从置信度任务到其余模型部分的所有梯度传递。相比之下,本文选择将结构预测模型与置信度模型分开训练,这样能够更好地独立调试各个组件,并在置信度预测任务中实现多项关键改进。
在AlphaFold3中,置信度模型由四层PairFormer组成,输入包括主干网络输出的最终单体(single)和对(pair)token表示,以及反向扩散预测的token两两距离编码。接着通过线性映射,预测晶体结构中每个原子是否解析、原子级LDDT(局部距离差异测试)和token对级别的PAE(预测误差估计)与PDE(预测方向误差)。
主干架构与初始化
观察发现,置信度模型的输入输出结构与主干网络高度相似,主干网络同样通过循环利用自身最终表示输出供去噪模型使用的表达式表示。借鉴大语言模型社区中通过微调预训练生成模型主干来训练奖励模型的做法,置信度模型被设计为包含完整的主干组件,并用训练好的主干权重进行初始化。因此,置信度模型包含AtomAttentionEncoder、MSAModule和48层的PairFormerModule。此外,仍然将预测的构象作为pairwise token距离矩阵的编码输入,通过线性层对最终的PairFormer表示进行置信度解码。
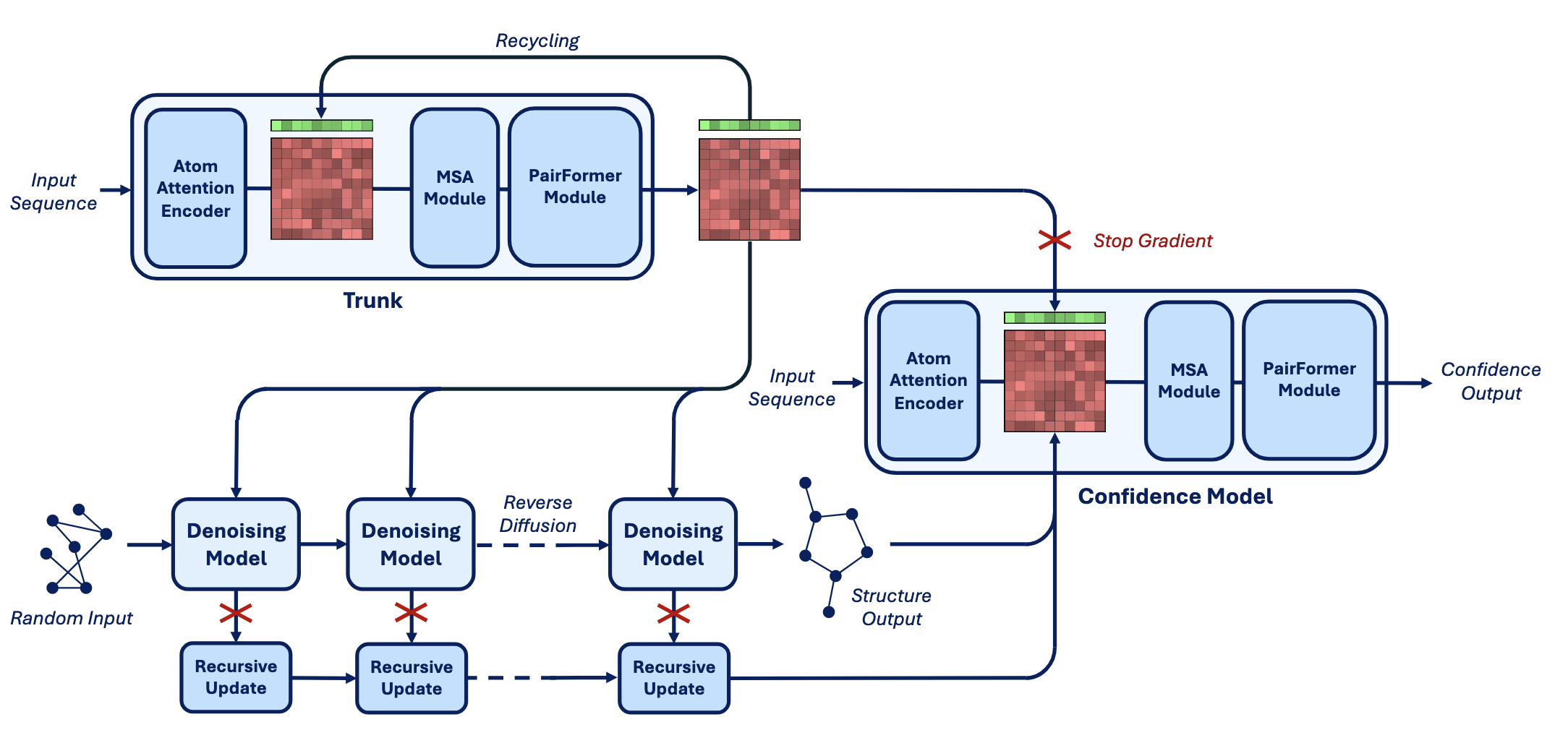
扩散模型特征
除了主干网络的表示,置信度模型还接收经过学习的、对每一步反向扩散中最终token表示的聚合结果。这些表示通过时间条件的递归模块在反向扩散轨迹上聚合,并与主干token级特征串联输入置信度模型起始层。进一步地,模型改进了token级特征向pairwise表示的输入方式,增加了线性变换后的token特征的逐元素乘积。
整体流程与训练
完整推理流程详见算法1,示意图见图3。训练时,置信度模型中借用自主干的组件参数初始化为主干的最终权重(采用指数移动平均),其他网络组件权重随机初始化,但最终层权重置零,以避免扰动预训练主干中已具备的丰富表达。

3.4 优化策略
为了加速模型运行并降低内存消耗,采用了多种计算技术,具体实现细节可参考公开代码库。
序列局部原子表示
AtomAttentionEncoder和AtomAttentionDecoder中的对原子表示进行pair-biased Transformer计算时,注意力机制被限制为序列局部,即每32个原子组成一个块,仅关注序列空间中距离最近的128个原子。实现上,开发了GPU高效的序列局部注意力计算,预先映射每16个token块到对应的key和query序列嵌入,再并行处理32×128的块状稀疏注意力,保持矩阵稠密,提升计算效率。
注意力偏置共享与缓存
去噪模型需针对每个扩散时间步对每个样本重复运行,而主干网络仅需运行一次并输出表示供所有去噪步骤使用。去噪模型中计算token和原子Transformer的注意力对偏置是最耗时部分,但这些偏置既不依赖于具体输入结构,也不依赖扩散时间步。具体包括AtomAttentionEncoder、AtomAttentionDecoder和DiffusionTransformer中所有层的注意力偏置及AtomAttentionEncoder中的中间单体和对原子表示。因此,这些部分只需计算一次,并在所有样本和反向扩散轨迹中共享,显著降低了计算成本,代价是需要额外的内存存储这些缓存。
贪心对称性校正
在验证和置信度模型训练时,需确定预测结构与真实结构之间的最优对齐,考虑相同链的排列组合及链内对称原子排序。由于排列组合数量随复合体规模指数增长,穷举不可行。设计了一种层次化贪心匹配算法:先搜索最优链分配,再在此基础上对每个配体或残基的原子扰动进行贪心选择。具体为,计算每个对称链不改变内原子排序时的全局LDDT,再对每个配体或核苷酸单元单独寻找能最大提升全局LDDT的扰动并应用。由于未被扰动的元素对的LDDT不变,计算仅需聚焦变动的距离矩阵行列。实际中限制链分配扰动不超过100种,原子扰动不超过1000种。
MSA模块与三角注意力的分块处理
为提升内存效率,引入分块策略以显著减少推理峰值内存消耗。借鉴OpenFold对三角注意力层的分块实现,将其推广应用到MSA模块,重点分块操作发生在过渡层、对权重平均层和外积层。这使模型在处理更大输入时保持可扩展性,且速度相近。
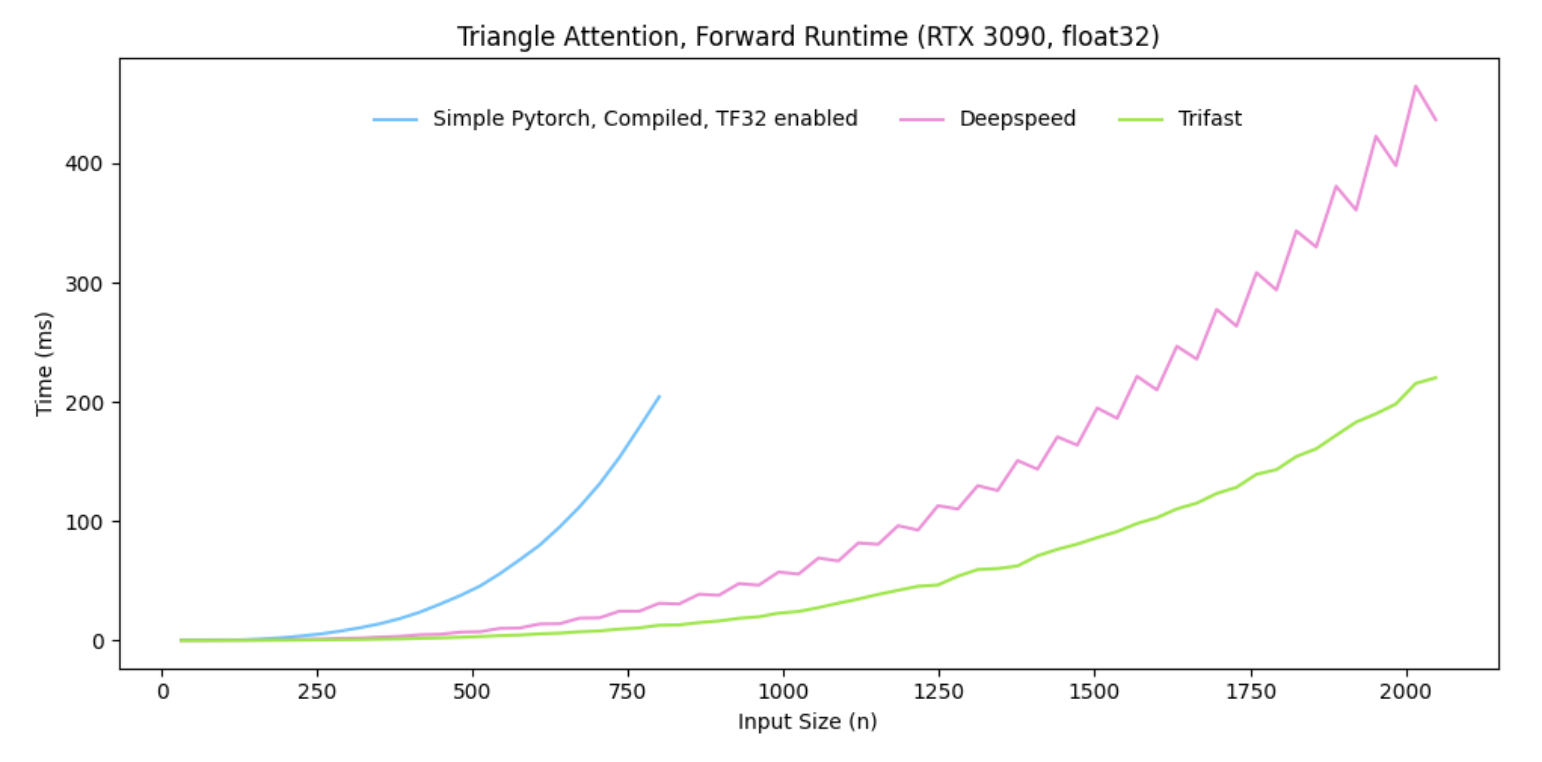
Trifast内核
PairFormer使用的三角自注意力计算复杂度为O(n³),其中内存消耗往往成为限制输入规模的瓶颈,中等规模时运行时间也较长。常用解决方案是采用分块计算。本文实现了基于Triton的内核,通过分块在线softmax优化该计算,内存复杂度降低至O(n²),并充分利用GPU架构加速实际输入规模下的计算。此内核为FlashAttention的扩展,支持内部偏置项。与PyTorch编译版本及其他高效内核的前向运行时对比见图4,代码托管地址:https://github.com/latkins/trifast。
4 Boltz 引导技术
4.1 引言
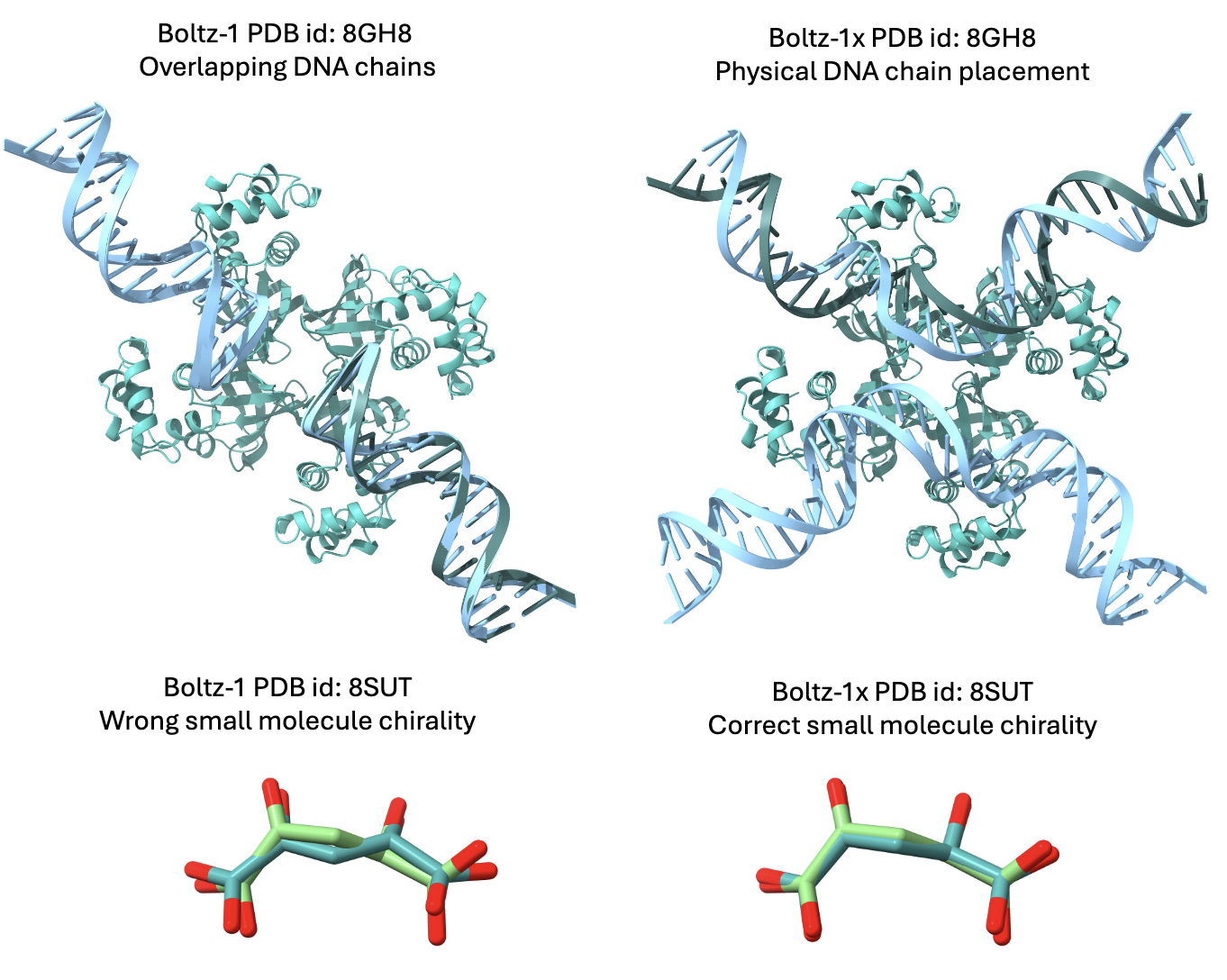
对Boltz-1若干预测结果的视觉检查发现,模型输出存在幻觉现象,其中最显著的问题是多个链结构几乎重叠在一起。类似的行为在AlphaFold3中也被观察到。此外,与之前基于机器学习的结构预测方法一样,Boltz-1也会产生部分不符合物理规律的结构,如原子间的空间冲突、轻微偏离的键长和键角、不正确的手性中心及立体键构型,以及预测的芳香环非平面化等。虽然这些问题对几何准确性的评价影响有限,但却极大限制了预测结构在后续应用中的实用性,例如分子动力学模拟等计算分析。
为解决上述问题,提出了一种新的推理阶段引导技术——Boltz-steering。该方法通过引入一个物理启发的势函数,调整原扩散模型定义的结构分布,从而“倾斜”模型采样分布:
其中,
一种采样该倾斜分布的思路是先从扩散过程采样一批粒子
相关工作
在生物分子结构领域,已有其他研究利用推理阶段势函数引导扩散过程。例如,RFDiffusion通过对每个时间步的x_0预测计算势函数,并施加梯度更新以促进特定对称蛋白质结构和链间接触的形成。Ishitani和Moriwaki则采用RDKit构象作为参考,基于预测构象与参考构象间键长、键角和手性体积的均方根误差(RMSE)定义损失,并通过梯度更新优化配体结构。相比之下,Boltz-steering采用基于距离界限的平底势(flat-bottom potential),旨在约束势函数处于合理构象范围内,而非强制匹配RDKit构象,同时扩展到其他物理性质,不仅使用梯度更新,还结合重新采样。DecompDiff也在反向扩散中利用梯度更新提升生成分子构象的物理合理性。
4.2 方法
Boltz-steering基于Singhal等人提出的Feynman-Kac(FK)引导框架,采用了一种不同且更有效的策略。在扩散反向过程的每个中间时间步,通过势函数
具体来说,在反向扩散过程开始时,先从初始倾斜分布
中采样;随后每个时间步
进行采样。
势函数
其中,
由于直接从倾斜转移核采样不可行,FK引导框架采用序贯蒙特卡洛(SMC)方法,在每个时间步生成多个样本(粒子),并根据重要性权重重采样。为简化表示,令采样算法为
其中
进一步,为了更有效地引导轨迹采样到低能量构象,采用了Bansal等人提出的反向通用引导(backwards universal guidance),将提议分布定义为
其中
对应地,重要性权重调整为
实际应用中,每个时间步都引入引导操作,但仅每隔3个时间步进行一次重采样,以确保足够的探索性。完整的Boltz-steering算法详见算法2。
4.3 约束势能
整体势能
其中,碰撞势能(clash potential)仅在时间点

四面体原子手性(Tetrahedral Atom Chirality)
对一个手性中心
这里,
键的立体化学(Bond Stereochemistry)
考虑一键
其中,
平面双键(Planar Double Bonds)
对于碳原子间的平面双键
内部几何(Internal Geometry)
为了确保模型生成的配体构象具有物理合理的距离几何,基于 RDKit 生成的上下界矩阵,定义了“平底”势能。对于一个包含
该势能确保所有原子对的距离均落在合理的范围内,避免生成非物理构象。
空间位阻碰撞(Steric Clash)
为防止空间碰撞,限制不同非键合链的原子间距离必须大于原子范德华半径之和的
其中,
链间重叠(Overlapping Chains)
为了防止链间重叠,定义了基于对称链重心间距离的时间依赖势能:
这里,
共价键链(Covalently Bonded Chains)
为了保证模型尊重共价键连接,定义势能约束共价键连接的不同链上原子间距离不超过
5 结果
本文在两个基准数据集上评估了模型性能:一是经过清理的近期 PDB 结构的多样性测试集(详见第 2.2 节),二是 CASP15 竞赛,这是最近一次涵盖蛋白质结构预测的社区竞赛,首次将 RNA 和配体结构纳入评估范围。两个基准均包含蛋白质复合物、核酸及小分子等多样化结构,适合作为评估像 Boltz-1 这样能够预测任意生物大分子结构的模型的试验平台。
在 CASP15 数据集中,筛选标准包括:(1)未被取消的竞赛目标,(2)具备对应的 PDB 编号以获得真实晶体结构,(3)化学计量信息中的链数与提供的链数匹配,(4)总残基数少于 2000。最终选取了 76 个结构。对于测试集,则剔除了含有共价结合配体的结构,因为当前 Chai-1 公开版本无法设置此类配体。两套数据集还排除了在 A100 80GB GPU 上因内存不足或其他原因导致计算失败的结构,最终留下 66 个 CASP15 结构和 541 个测试集结构用于评估。
在基准对比方面,模型的表现与当前最先进的生物大分子结构预测模型 AlphaFold3 和 Chai-1 进行了比较。后两者均由专有商业许可发布,且不公开训练代码和流程。Chai-1 模型通过 chai lab 软件包(版本 0.2.1)运行。所有模型均采用 200 步采样、10 轮循环,并输出 5 个预测结果。多序列比对(MSA)使用了最多包含 16384 条序列的预计算数据。由于 Chai-1 需要对序列来源进行注释,所有 Uniref 序列标注为 uniref90,其他序列标注为 bfd uniclust。尝试过替代注释方式,但未发现对模型性能有明显影响。
评估标准
为了评估模型在多样化生物大分子和结构上的表现,采用了多项成熟的指标,具体包括:
-
平均全原子 LDDT:衡量所有生物大分子局部结构的准确性。
-
平均 DockQ 成功率:定义为 DockQ > 0.23 的预测比例,用以衡量蛋白质-蛋白质相互作用预测的优良程度。
-
平均蛋白-配体界面 LDDT:评估配体与结合口袋的相互作用质量,是 CASP15 官方的配体类别评估指标。
-
配体结合口袋对齐 RMSD 小于 2 Å 的配体比例:广泛采用的分子对接准确度衡量标准。
-
构象的物理质量,通过 PoseBusters 提供的一组物理规则检测构象的合理性,包括:
(a) 配体键长是否在 RDKit 设定的合理范围内;
(b) 配体键角(1-3 原子距离)是否合理;
(c) 配体内部是否存在原子间碰撞(距离低于合理下限);
(d) 四面体原子的手性是否保持;
(e) 键的立体化学是否保持;
(f) 不同非键合链的原子间是否存在碰撞,要求距离大于范德华半径之和的 0.75 倍。
所有指标均使用 OpenStructure 2.8.0 计算。LDDT-PLI、DockQ 和配体 RMSD 的成功率均覆盖所有蛋白-蛋白和蛋白-配体界面,先对每个复合物内的界面结果求平均,再跨复合物取平均。为保证公平比较,所有方法均生成 5 个样本,并分别评估“最佳预测(oracle)”与“最高置信度预测(top-1)”。
为推动领域进展并促进基准的广泛采用,所有模型的输入、输出、评估结果及聚合脚本均已公开,下载说明见官方 GitHub 仓库。
结果总结
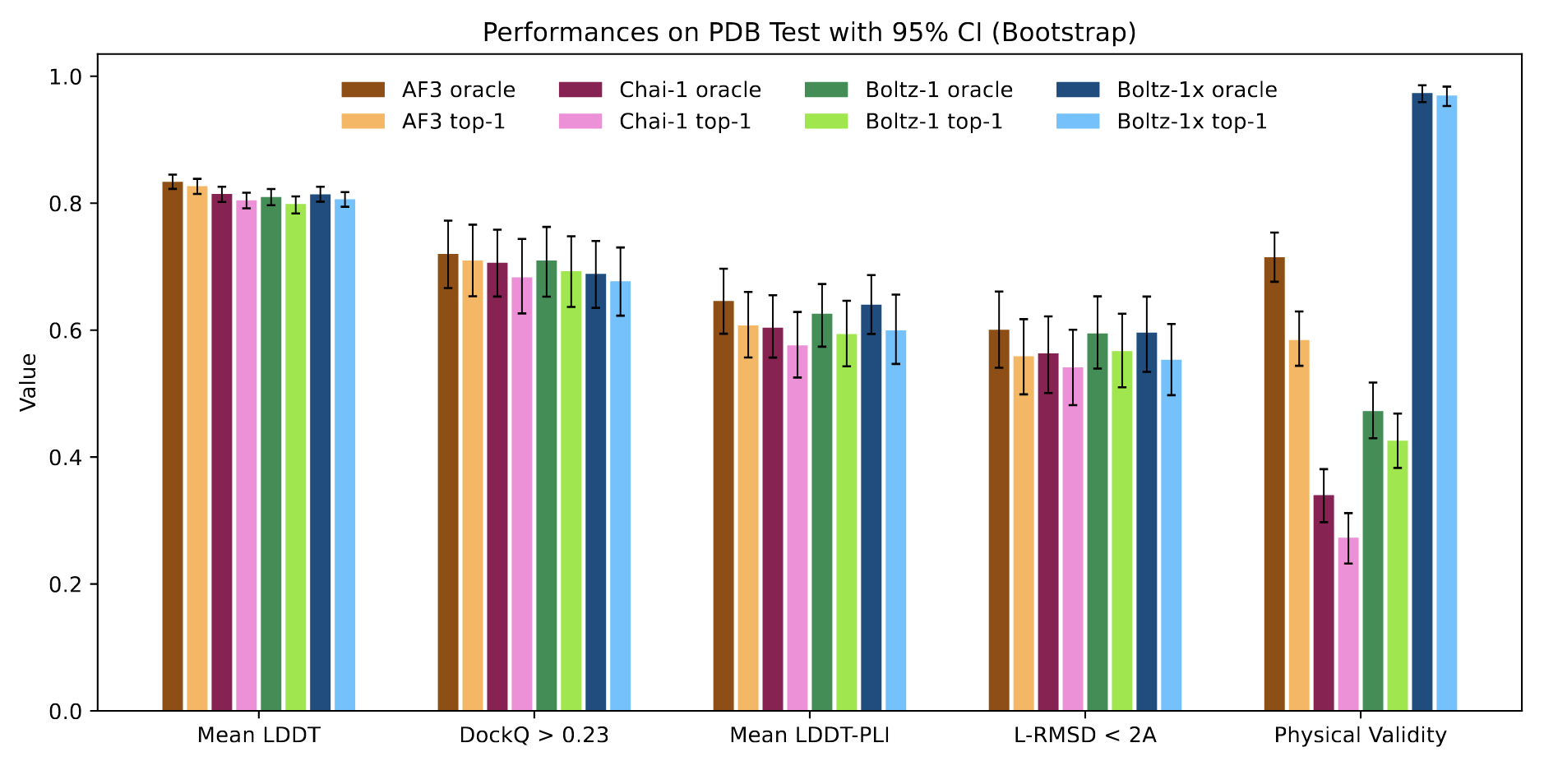
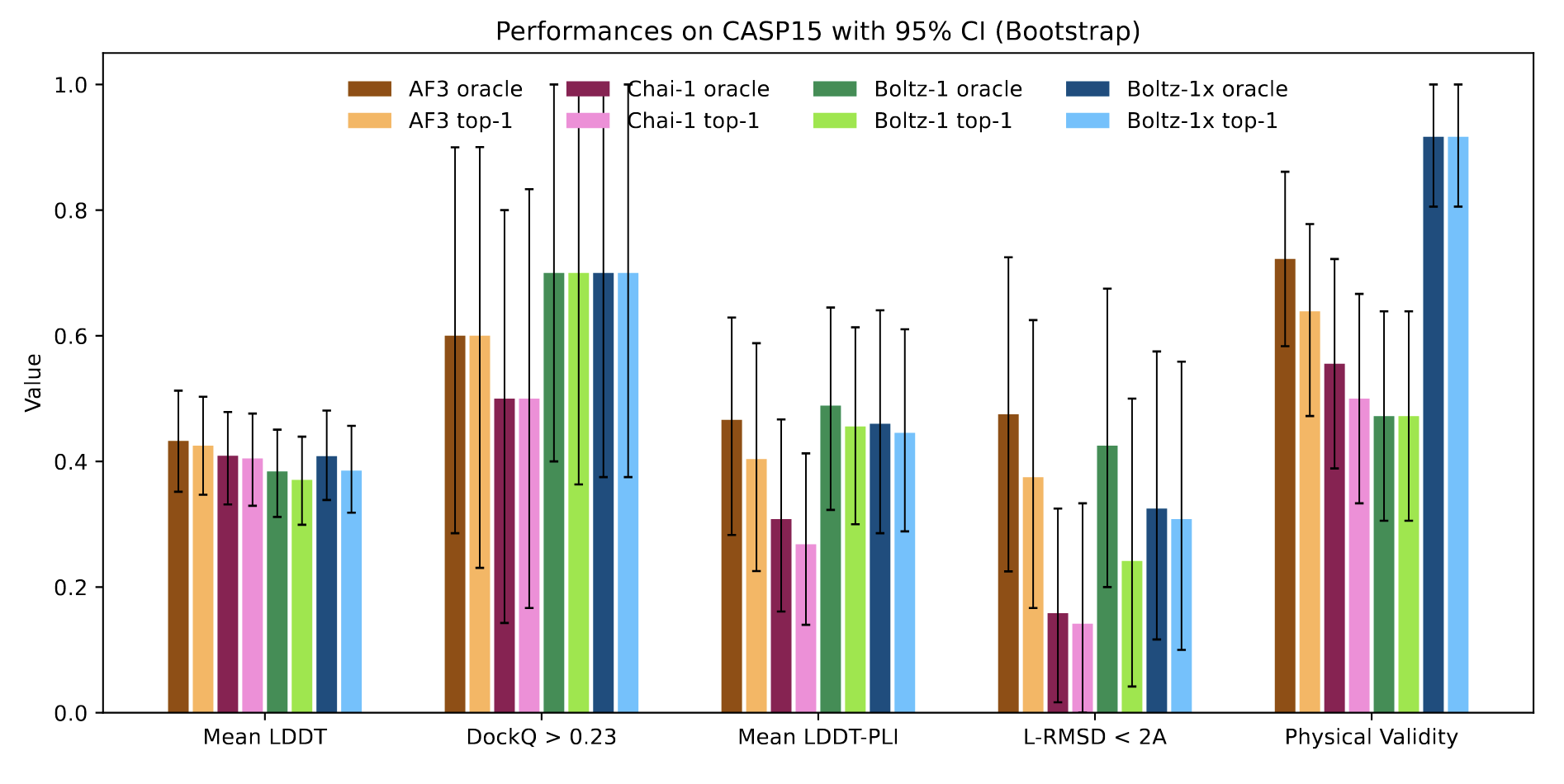
AlphaFold3、Chai-1 与 Boltz-1 在 CASP15 和测试集的多个指标上表现相当。AlphaFold3 在平均 LDDT 指标上稍占优势,可能因其额外的 RNA 和 DNA 蒸馏数据集提升了对核酸复合物的处理能力。
蛋白-蛋白相互作用方面,三者表现相近。AlphaFold3 在测试集中以略高比例的 DockQ > 0.23 界面领先,但差异均在置信区间内。蛋白-配体指标中,AlphaFold3 和 Boltz-1 在平均 LDDT-PLI 和配体 RMSD < 2 Å 的比例上优于 Chai-1,但差异同样在置信区间范围内。
整体上,Boltz-1 在蛋白-蛋白与蛋白-配体相互作用的预测准确性方面,与领先模型 AlphaFold3 和 Chai-1 不相上下。
物理质量检测方面,测试集中 Boltz-1 的 top-1 构象有 57% 未通过物理合理性检查,提示存在较严重的物理问题。Chai-1 和 AlphaFold3 通过率分别约为 27% 和 58%。而 Boltz-1x 版本则显著提升,通过率达到 97%,且性能维持与其他模型相当的水平。
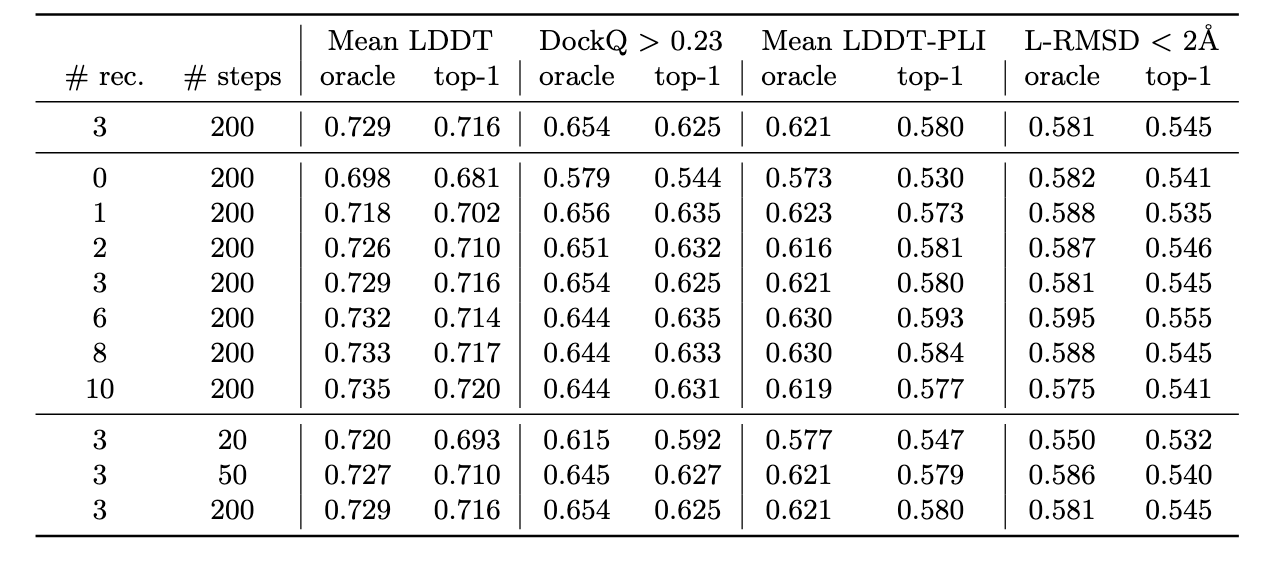
消融实验(表 1)显示,随着循环次数和扩散步数的增加,模型性能整体单调提升,且在 3 次循环和 50 步扩散后趋于稳定。
示例展示(图 1)展示了测试集中两个难点目标,Boltz-1 表现优异,TM 评分约达 95%。
6 结论
本文介绍了 Boltz-1,这是首个实现与 AlphaFold3 相当准确度、且完全开源且商业可访问的生物大分子复合物三维结构预测模型。为实现这一目标,团队复现并扩展了 AlphaFold3 的技术方案,在模型架构、数据清洗、训练及推理流程上引入多项创新。通过在多样化测试集和 CASP15 基准上的实证验证,Boltz-1 展现出与当前最先进的结构预测方法 AlphaFold3 和 Chai-1 相当的性能。
此外,推出了 Boltz-1x,该版本利用一种名为 Boltz-steering 的新推理技术,显著提升了预测构象的物理合理性,同时保持了 Boltz-1 的几何精度。
Boltz-1 和 Boltz-1x 的开源发布,标志着先进生物大分子建模工具民主化的关键进展,极大拓宽了其跨领域的适用性。通过 MIT 许可自由提供训练与推理代码、模型权重及数据集,旨在赋能科研人员和机构,推动基于 Boltz 模型的创新实验。期待 Boltz 系列成为研究者构建的基础平台,促进协作,加速对生物大分子相互作用的理解,并推动药物设计、结构生物学等领域的突破发展。