JCIM 2025 | Freedom Space 3.0: 利用机器学习辅助筛选可合成的小分子
今天介绍的是发表在 JCIM 2025 的一项最新研究——Freedom Space 3.0:利用机器学习辅助筛选可合成的小分子。在药物发现中,超大规模化学数据集已成为探索化学空间的重要工具,然而传统组合化学生成的分子往往面临合成可行性不足的问题。Freedom Space 3.0通过引入机器学习模型,在分子枚举之前对构建模块进行预筛选,显著提高了候选分子的质量与合成成功率。该数据集包含约50亿个分子,由十种经过验证的化学反应生成,并与Enamine REAL Space形成互补。研究团队不仅通过计算方法系统评估了分子的理化性质、多样性与合成可及性,还在实验层面验证了732个分子的合成,成功率超过81%。这一成果表明,基于机器学习的策略为解决超大规模化学库设计中的瓶颈提供了有效路径,为早期先导化合物发现与优化开辟了新的可能。

获取详情及资源:
- 📄 论文: https://doi.org/10.1021/acs.jcim.5c01912
- 💻 代码: https://github.com/ChemSpace-LLC/Freedom_Space_3
- 📊 数据: https://chem-space.com/compounds/freedom-space
0 摘要
机器学习(ML)的进步正在深刻改变化学空间的探索方式,使得能够根据特定应用需求定制子空间。该研究介绍了Chemspace Freedom Space 3.0的开发过程——一个通过ML筛选构建模块而生成的、可合成小分子化学库。该方法在枚举之前,利用基于定制分子表征训练的模型对构建模块进行精炼筛选,从而提升所得分子的质量与合成可行性。Freedom Space 3.0包含约50亿个分子,其生成依赖于十种经过充分验证的化学转化,并与Enamine REAL Space互为补充。研究进一步通过计算评估了这些分子的理化性质、化学多样性以及合成可及性。同时,实验验证显示,在对700个分子进行合成尝试中,4−6周内的成功率超过80%,充分证明了Freedom Space 3.0在加速先导化合物发现与后续优化流程中的潜力。
1 引言
随着更先进的计算方法与硬件的发展,超大规模化学数据集已成为药物发现中不可或缺的重要工具。由于其规模和多样性,利用这些数据集能够显著提升发现新型且高效先导化合物的概率,从而加速药物候选物的筛选。药物样化学空间的估算规模超过
除了企业内部库之外,美国NIH−NCI团队开发了公开可用的Synthetically Accessible Virtual Inventory (SAVI)。瑞士伯尔尼大学Reymond课题组提出了化学空间项目,通过算法对分子骨架进行穷举枚举,随后在价键规则与稳定性约束下精炼,形成公开数据库GDB,其最新版本GDB-17包含1660亿个、最多17个重原子的分子。商业化学数据集中,较为知名的包括Enamine REAL Space(700亿分子)、Wuxi GalaXi(120亿)、OTAVA CHEMriya(120亿)、Mcule ULTIMATE(1.4亿)与eMolecules eXplore(7万亿)。
然而,组合化学数据集的主要缺陷在于分子的可合成性。特定官能团的存在并不能保证反应在预设条件下顺利进行,可能出现低收率、副产物、纯化困难甚至完全无法反应等问题,从而限制了数据集的应用价值。为解决这一问题,近年来兴起了计算机辅助合成预测(CASP),依赖机器学习与人工智能来预测反应的可行性、收率与最佳条件。CASP已在逆合成、反应产物预测、选择性预测以及反应条件与试剂预测中展现出显著应用价值。准确预测反应收率在有机化学中尤为关键,尤其是在药物发现与开发阶段,它不仅影响单次反应的试剂选择,更直接决定虚拟分子生成的质量。然而,由于化学反应的复杂性(包括起始试剂、反应条件与人为操作等因素),收率预测仍然极具挑战。尽管在高通量实验(HTE)数据集中已有显著进展,但在文献数据上的表现大幅下降,原因在于数据噪声和实验细节缺失。
在应对合成可行性问题时,基于高质量数据训练的收率预测模型用于按需合成化合物集合的构建,被认为是相对可靠的方法。然而,这类预测需要逐一评估集合中每个产物,对于数十亿规模的分子集合而言,其应用受到明显限制。相比之下,Enamine REAL通过使用验证过的内部构建模块与合成流程,保证了>80%的合成成功率,且在3−4周内即可完成。然而,这一方法高度依赖Enamine所提供的模块,若要引入新模块则需投入大量时间与精力。
因此,开发一种能够简化构建模块筛选过程的新方法,并从任何商品化目录中快速挑选可合成模块,显得尤为重要。该研究提出了Freedom Space 3.0,一个基于机器学习试剂筛选策略生成的超大规模组合化学空间。研究利用Enamine的构建模块实验数据训练模型,从商业目录中预筛选适合的模块,并在验证过的化学转化体系中应用。这一方法无需对每个模块进行额外实验验证,就能高效生成大规模、多样化且合成可行的化学数据集。Freedom Space 3.0为药物早期发现提供了重要资源,在先导化合物发现与优化中具有显著应用潜力。
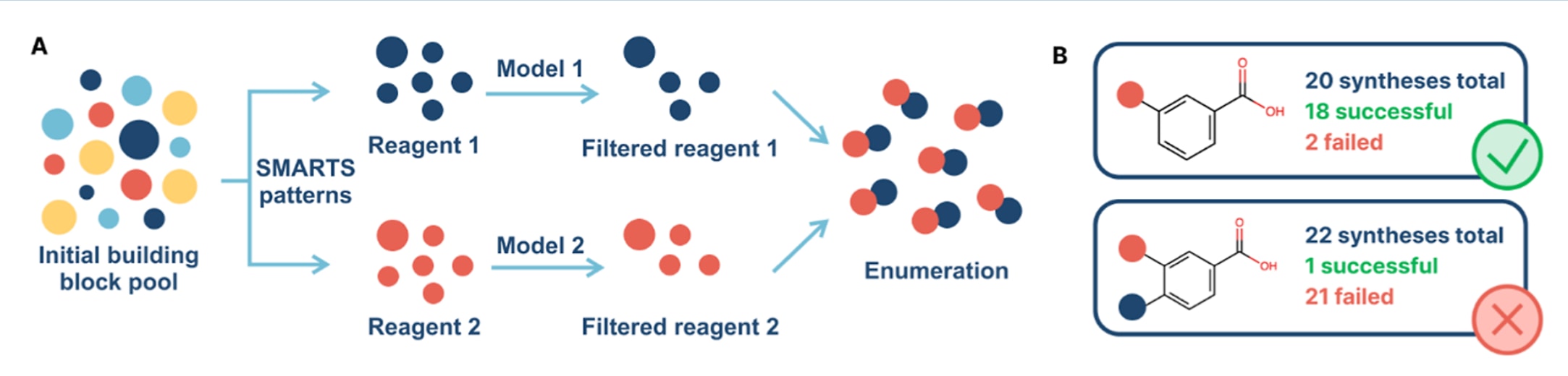
图1 | (A) 基于机器学习的反应过滤工作流程。 需注意,每一种反应方案中的每个试剂都由独立模型进行筛选。(B) 训练数据示例,其中所示分子与数值仅用于说明,并非实际训练集的一部分。
2 结果与讨论
2.1 Freedom Space 3.0的构建
前一版本的Freedom Space包含2.01亿个分子,其构建依赖于人工筛选的构建模块。尽管有经验的合成化学家的专业知识极为宝贵,但这种方式的通量受到人工分析效率的限制。Freedom Space 3.0的目标是提供更大规模的化合物集合,并设计一条自动化工作流程,通过机器学习辅助的试剂筛选来处理任意化合物库,从而实现对试剂的高效筛选。该工作流程(如图1A所示)旨在简化可合成化学空间的生成过程,避免依赖耗时的实验验证。
具体步骤包括:首先建立一个经过整理的构建模块/试剂集合,来源于可信供应商提供的商业化合物。随后,通过基于SMARTS的模式匹配方法,为每种目标反应选择相应的试剂子集,以确保其化学合理性。接着,这些初始试剂池会进一步由机器学习二分类模型筛选,模型预测每个试剂在对应反应体系中成功参与的概率,仅保留被判定为合适的试剂。最后,基于这些筛选后的试剂进行组合枚举,生成具有高合成可行性的组合化学数据集。
该流程的核心优势在于高质量实验数据的可得性,使得机器学习过滤器的开发成为可能。这一数据集由Enamine提供,包含其REAL Space的详细实验统计信息:包括每个试剂在特定合成方案中的成功与失败次数、使用的具体反应条件以及总体产率。对于每一个“试剂−反应”对,数据集中均提供了二元标签,指示该试剂是否适合用于REAL衍生物的枚举(如图1B所示)。这份经精心注释的数据集成为监督式机器学习的基础,保证了筛选模型的可靠性。
2.2 化学转化的选择
为构建Freedom Space 3.0,研究团队需要挑选合适的化学反应。选择标准主要包括:是否拥有足够成功实验以训练机器学习过滤器、对应构建模块的现货可得性以及最终产物的多样性。因此,最终确定了七种化学转化用于训练ML模型,同时额外引入三种依赖人工筛选的转化,因为它们缺乏足够的训练数据。这三类人工筛选的反应用于增加分子骨架的多样性,特别是引入取代吡rimidine与quinazolin-4(3H)-one骨架。在十种反应中,有三类涉及三元试剂组装,其余则由两个试剂组合而成。所有化学转化及其对应试剂的详细描述见补充信息中的表S1。
2.3 基于机器学习的过滤器构建
在该研究中开发的机器学习模型被用作过滤器,用于精炼初始的构建模块集合。初始集合来自Chemspace库存化合物库,通过内部设定的SMARTS模式筛选得到。其核心目标是剔除在目标转化中可能表现不佳的试剂,从而保证生成的组合化学空间中富含合成可行的分子。Enamine提供的数据包含试剂的SMILES、该试剂对应的实验总数以及成功实验的次数。其中,成功实验被定义为产物纯度高于90%,并由LC/MS或¹H NMR确认,这使得数据能够涵盖纯化过程中可能出现的问题。在七种机器学习过滤的化学转化中,六种是双试剂反应,一种为三试剂反应,因此训练的模型数分别为2个和3个,因为每个试剂必须单独过滤。所有模型均被设计为分类器,用于预测某一试剂在特定合成反应中的成功概率。经过多种方法的评估,最终选择了定向消息传递神经网络(DMPNN)作为模型架构,因为其在预测任务中 consistently 表现优于其他方法,能够有效学习复杂的结构−性质关系,具有高精度与强泛化性。每个过滤器由6个模型组成,通过平均预测结果实现集成。与以往方法不同的一点在于,该研究采用了基于合成子(synthon)的分子表征,而非传统的SMILES。这种分离图结构不仅提供了反应中心位点的信息,还包含相应的官能团特征,使模型能够对共享相同synthon的构建模块做出区分,例如区分芳环上不同卤素原子的反应性。
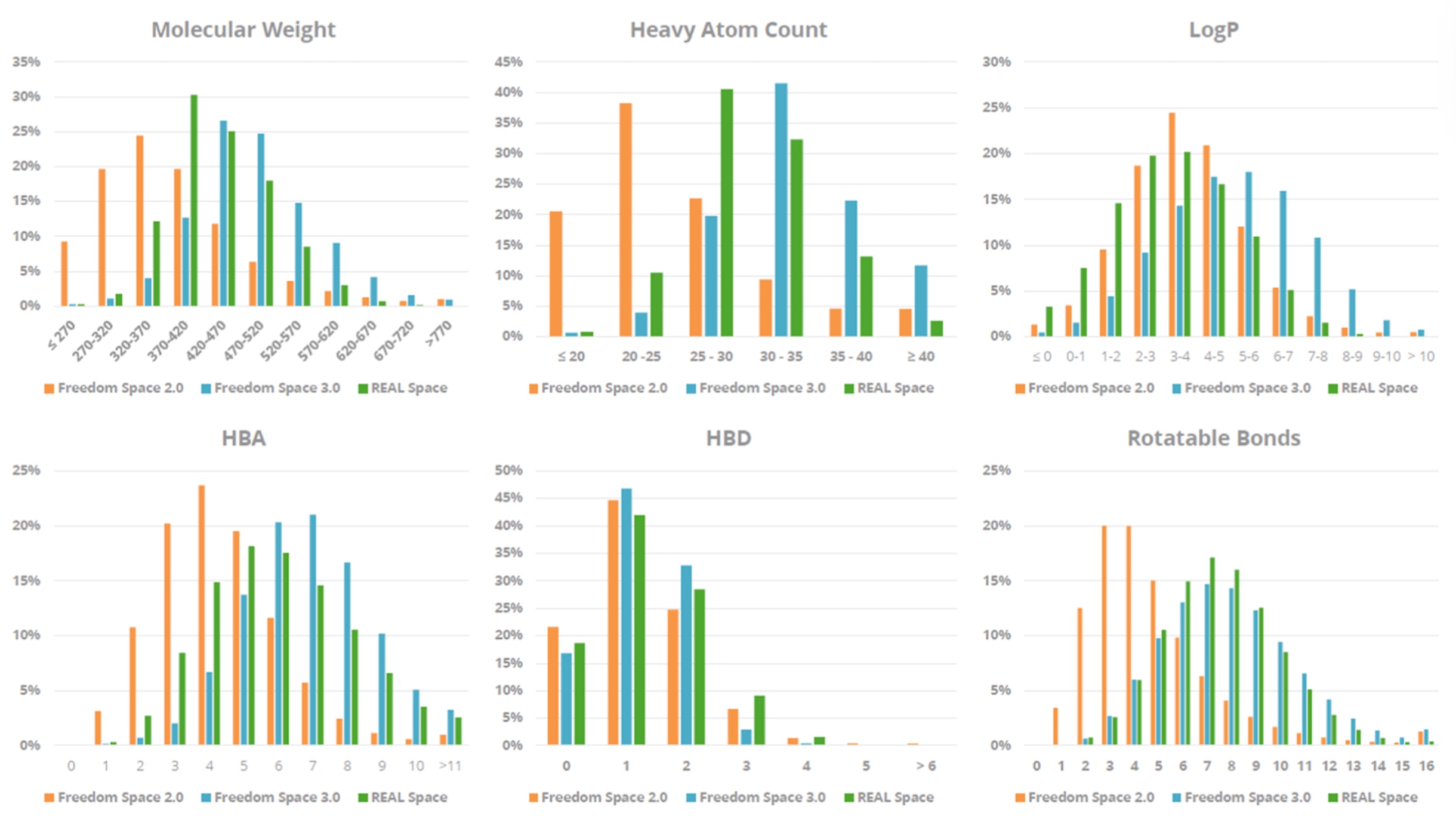
图2 | Freedom Space 3.0的理化性质概况(分子量、重原子数(非氢原子数)、LogP(溶质在正辛醇与水之间的分配系数对数)、HBA(氢键受体数)、HBD(氢键供体数)以及可旋转键数(非环状、与非末端重原子相连的单键数)),并与Freedom Space 2.0及Enamine REAL Space(版本:2025年5月)进行对比。所有性质均由RDKit计算获得。
2.4 构建模块的选择
Freedom Space 3.0的设计目标是补充Enamine REAL Space。因此,除了Enamine,还从其他供应商处选择试剂。供应商的筛选标准包括:可靠性、目录信息的时效性以及交付效率,最终还会结合价格与交付周期进行过滤。得到的初始构建模块清单随后根据所选合成反应进行分类,这一步利用SMARTS模式来保证所需官能团的存在,同时排除部分不兼容的官能团。接下来,这些子集被逐一处理以生成对应的synthon,构建分子表征,并通过训练好的模型进行过滤。
2.5 组合化学空间的生成
在ML辅助的构建模块筛选之后,组装得到基于synthon的Freedom Space 3.0,规模约90亿个分子。为保证该数据集对计算资源有限的研究团队依然可用,还生成了一个枚举版本,包含50亿个分子。由于三试剂反应产物占据相当比例,研究团队对其分子量与重原子数作了限制,以避免分子过大,同时通过使用多样化的试剂集合保持结构多样性。最终,Freedom Space 3.0既可作为枚举数据集,也可作为synthon格式发布,兼容传统与基于synthon的探索方法。
2.6 理化性质与多样性分析
为评估Freedom Space 3.0的理化性质分布与结构多样性,研究团队分别独立计算并与参考数据集(Freedom Space 2.0与Enamine REAL Space)对比。结果显示,Freedom Space 3.0中常见分子描述符的分布(见图2)呈高斯状,表明其覆盖了从小片段类分子到延伸至**超越“Rule of Five”(bRo5)**区域的广阔化学空间。这种多样性使其适用于药物发现的多个方向,包括基于片段的设计和蛋白−蛋白相互作用的调控。与Freedom Space 2.0相比,3.0版本的分子普遍具有更大的分子量、更高的重原子数、更多的可旋转键等规模相关特征,这一变化主要源于引入了三试剂反应,天然会生成更大、更复杂的产物。此外,扩展的构建模块集合和反应类型也推动了数据集规模从2.0版本的2.01亿个显著增长至3.0版本的90亿个。
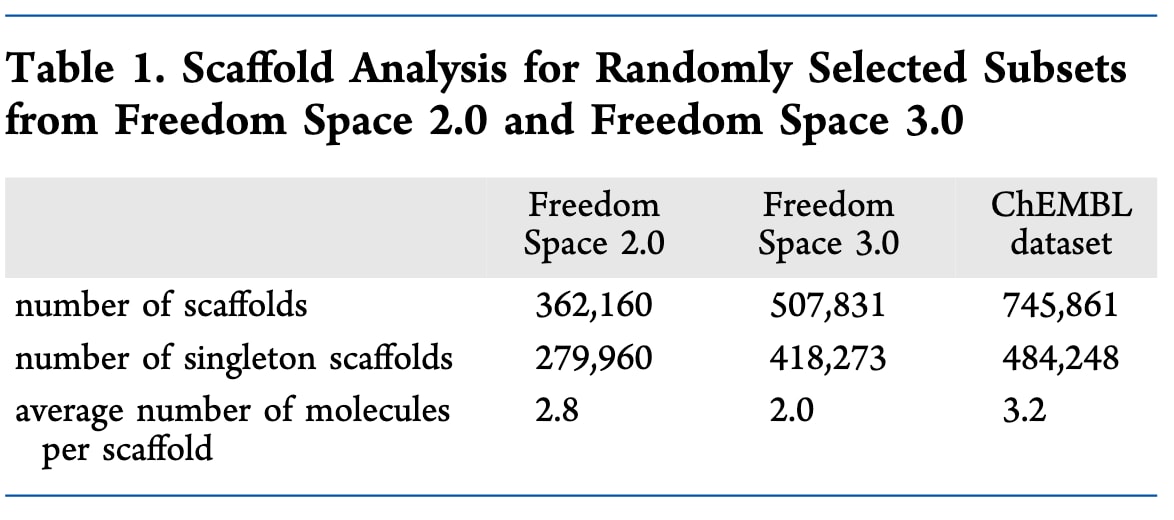
进一步利用Murcko骨架分析评估了不同版本的多样性与覆盖率。对随机抽取的100万个分子生成Murcko骨架,结果显示:Freedom Space 2.0中有362,160个独特骨架,而Freedom Space 3.0中则增加到507,831个,提升约1.4倍。单体骨架(仅对应一个分子的Murcko骨架)比例在2.0中为77%,在3.0中为82%,反映了后者的更高多样性。最常见的骨架也从2.0的苯环(20,702个分子)转变为3.0的N4-苄基-N2-苯基喹唑啉-2,4-二胺(5,888个分子),体现出分子复杂性的提升。对比ChEMBL数据库(version 35, 2,474,590个分子)的Murcko骨架生成结果,进一步说明了3.0的覆盖范围。利用UMAP投影(基于Morgan指纹与Tanimoto相似度)可视化结果(见图3)显示,Freedom Space 3.0与ChEMBL有部分重叠,表明其包含生物相关骨架,同时还拓展至更广阔的化学空间,相较2.0表现出更高的结构多样性和新型化学型。
表1 | Freedom Space 2.0与Freedom Space 3.0中随机抽取子集的骨架分析结果
2.7 化合物合成可及性的计算评估
延续先前的工作,研究团队对Freedom Space 3.0中的分子进行了合成可及性的计算评估。主要使用了SAscore、RAscore以及AiZynthFinder的逆合成路径预测三种方法。考虑到数据集中包含双试剂和三试剂反应产物,研究分别对两类分子独立评估,以更好展示其性质。具体操作是:从双试剂和三试剂产物中各随机抽取100万个分子,用于SAscore与RAscore评估;而AiZynthFinder因计算量更大,仅抽取其中的5万个分子进行分析。
结果显示(SAscore范围为1至10,10表示难以合成),大部分分子分布在2−4之间,表明具有良好合成性。RAscore范围为0到1,表示找到逆合成路线的概率。多数Freedom分子的概率高于80%,其中双试剂产物仅有3.2%低于0.4,而三试剂产物这一比例为13.4%,说明后者合成难度相对更高。使用AiZynthFinder时,双试剂产物仅1.5%的查询失败生成逆合成路径,而三试剂产物为5.3%,进一步验证了前述趋势。综合三种方法结果可见,三试剂产物整体评分稍差,但总体上超过96%的分子都能生成逆合成路径,说明Freedom Space 3.0中的分子具有高度可合成性。当然,这一结论仍需通过实验验证加以确认。
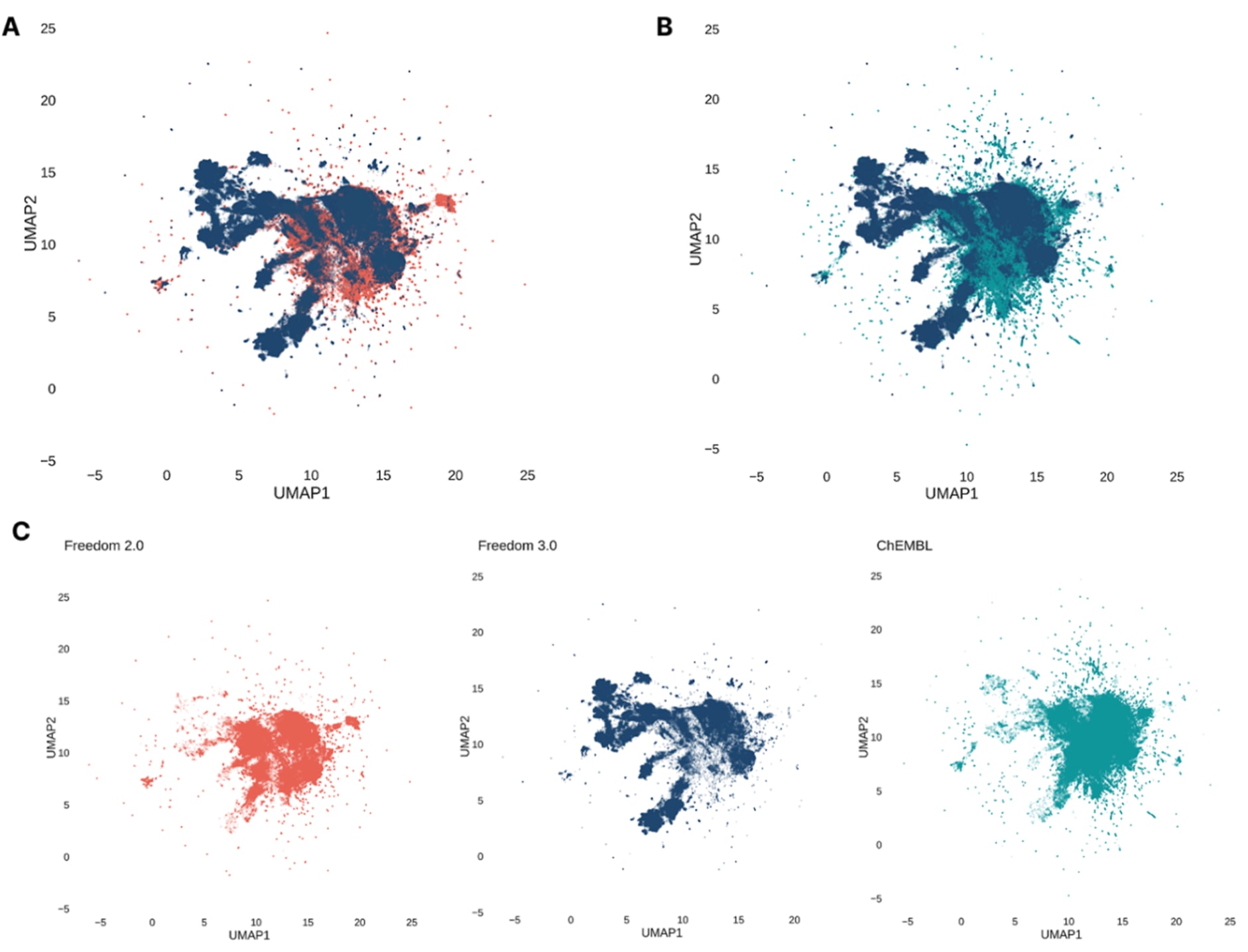
图3 | Freedom Space 2.0(·)、Freedom Space 3.0(·)以及ChEMBL数据库(·)骨架的分析结果。 对于ChEMBL数据集,其骨架基于整个数据库计算。(A) Freedom Space 2.0与Freedom Space 3.0的UMAP表示重叠情况;(B) Freedom Space 3.0与ChEMBL35的UMAP表示重叠情况;(C) Freedom Space 2.0、Freedom Space 3.0与ChEMBL35数据库的UMAP嵌入可视化结果。
2.8 合成可及性的实验验证
Freedom Space 3.0已向科研社区开放,用于先导发现与扩展研究,研究人员可通过FTP、chem-space.com门户及BioSolveIT的infiniSee工具访问。从研究工作流中筛选出的分子被送至Chemspace,由内部算法识别其起始试剂与反应类型,并由Enamine德国实验室执行合成。实验遵循标准验证过的合成步骤,采用瓶式反应,产物经制备型HPLC纯化,LC/MS和¹H NMR确认纯度通常超过90%。
共对732个分子开展实验合成(来自10个订单),平均成功率为81%,与计算结果一致。具体订单的成功率有所差异,源于抽样自Freedom空间的不同区域。值得注意的是,机器学习过滤反应与人工过滤反应的表现相当,展示了自动化流程的有效性。合成成功率最低的是Suzuki偶联反应,但这与模型表现无关,而是由于该类反应本身对无氧、无水条件要求严格。随着更多实验数据的积累,这些结论将进一步得到完善。
此外,还对三个不同化合物集合的订单履行情况和时间线做了深入分析。结果表明:起始试剂从供应商到达实验室(M1)仅需6−8天;M2阶段(合成、纯化与质控)在4−5周内完成,其中3周时已有50−70%的分子完成合成;M3阶段(最终产品发货)仅需3−5天。整体时间表符合业界标准交付周期,证明Freedom Space具备高交付能力。
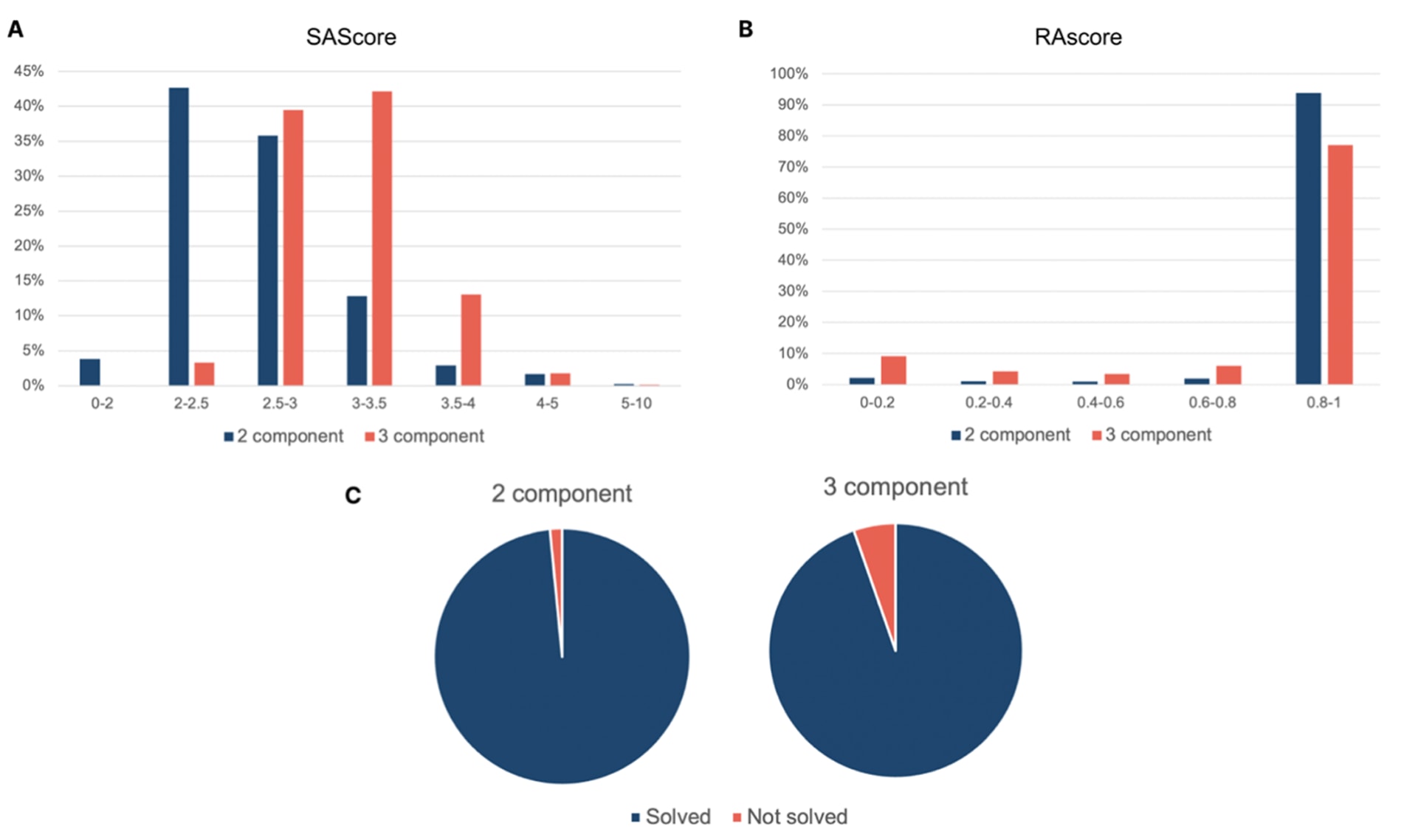
图4 | Freedom Space 3.0分子合成可及性的计算评估(分别从双组分与三组分子空间中各随机抽取5万个分子),基于SAscore(A)、RAscore(B)与AiZynthFinder(C)的结果。 “Solved”表示成功生成逆合成路径,“Not solved”表示在指定参数下生成失败。(随机抽样细节及完整的合成可及性评估设置见方法部分)
2.9 Freedom化合物与定制合成及Enamine REAL的对比分析
为了突出Freedom Space 3.0的实际优势,研究团队对其与其他选项进行了基准对比。对比对象包括:①来自DNA编码文库(DEL)筛选的分子进行的定制合成,②Enamine REAL Space中的分子。由于DEL分子常通过组合化学获得,与REAL和Freedom的设计理念一致,因此能保证对比的公平性。
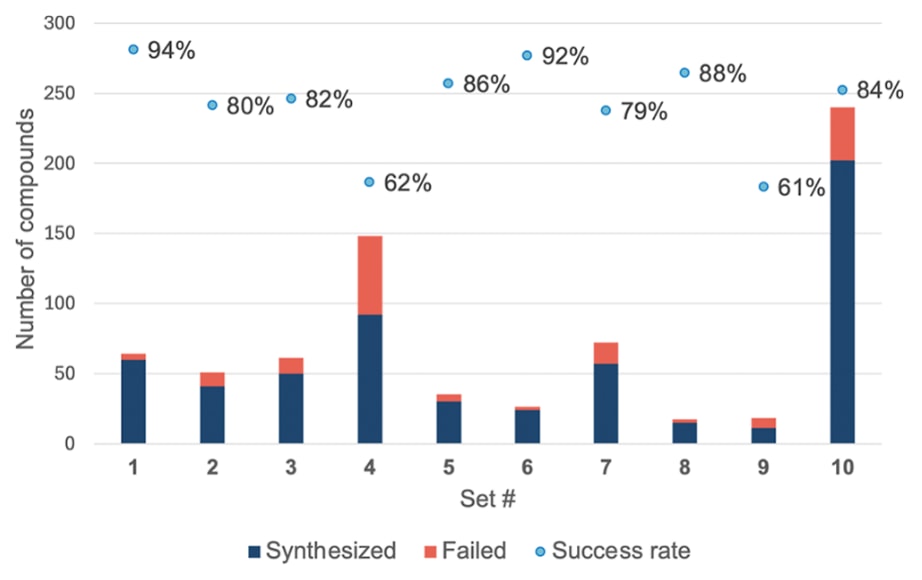
图5 | Freedom Space 3.0化合物的合成成功率。
分析聚焦三个关键指标:单个分子的合成成本、交付周期与合成成功率。结果显示,Freedom化合物在这三项指标上位于Enamine REAL与全定制合成之间。相较REAL,其成本与交付周期略高,原因在于需要外部采购试剂;但成功率保持在可比水平,这得益于机器学习过滤的应用。REAL化合物多依赖自动化并行化学合成,而Freedom分子由实验室化学家合成,这也有助于维持较高的成功率。整体而言,Freedom化合物为先导发现与扩展提供了一种可靠且可及的选择。

图6 | Freedom Space 3.0化合物的订单履行情况与时间线。
表2 | 三个关键参数(价格、交付周期与成功率)在三类来源化合物间的对比:定制的Off-DNA合成、Chemspace Freedom Space与Enamine REAL Space。

3 结论
Freedom Space 3.0在可合成化学库的设计上迈出了重要一步,它通过机器学习在分子枚举之前精炼试剂筛选,显著提升了化合物集合的合成可行性。全面的计算分析表明,Freedom Space 3.0在理化性质、化学多样性与合成可及性之间达到了理想平衡,使其成为先导发现与扩展的重要资源。实验验证显示,在732个分子中平均合成成功率为81%。这些结果充分证明,基于机器学习的设计有潜力克服传统超大规模化学库构建中的限制。所描述的工作流程还可推广至基于专有化合物集合的自动化可合成化学数据集构建。
未来展望
Freedom Space 3.0已成为学术界与工业界广泛认可的可合成数据集,并在众多研究项目中得到应用,实验合成成功率保持在约80%。在其成功基础上,研究团队正积极推进Freedom Space 4.0的开发。新版本将沿用类似的工作流程,但在化学转化与试剂范围上显著扩展。当前正评估约50种反应,试剂集合规模也将扩大近两倍。新的化学转化与试剂将进一步提升化合物库的规模与多样性,使Freedom Space 4.0能够更好地满足研究界的期待。