Cell Biomaterials 2025 | AMP-Diffusion: 生成式潜在扩散语言建模驱动的抗感染合成肽
今天介绍发表在 Cell Biomaterials 的一项最新研究 —— AMP-Diffusion:生成式潜在扩散语言建模驱动的抗感染合成肽。随着抗菌耐药性(AMR)问题日益严峻,寻找新型抗感染策略已成为全球健康的关键挑战。抗菌肽(AMPs)因作用机制多样、难以诱导耐药,被认为是传统抗生素的有力替代,但其设计仍受限于庞大序列空间和复杂的结构–功能关系。研究团队提出了 AMP-Diffusion 模型,基于蛋白语言模型 ESM-2 的潜在嵌入空间,通过去噪扩散过程生成具有潜在活性的 AMP 序列。经过深度学习预测与筛选,合成的 46 条候选肽展现出广谱抗菌活性,其中多条对多重耐药菌株同样有效,且大部分具有低细胞毒性。在小鼠感染模型中,优势肽的疗效可与多黏菌素 B 和左氧氟沙星相媲美。该研究不仅展示了生成式 AI 在药物设计中的巨大潜力,也为应对 AMR 危机提供了全新的解决思路。

获取详情及资源:
- 📄 论文: https://doi.org/10.1016/j.celbio.2025.100183
- 💻 代码: https://github.com/programmablebio/amp-diffusion
0 摘要
生成式人工智能(AI)为肽分子设计提供了强有力的新途径,但这一过程仍面临巨大挑战,包括庞大的序列空间、复杂的结构–活性关系,以及在保持抗菌效力的同时降低毒性的需求。 该研究提出了一种新方法——AMP-Diffusion,该模型基于抗菌肽(AMP)序列并结合蛋白质语言模型(pLMs)的嵌入进行微调,从而实现潜在扩散建模。AMP-Diffusion能够系统性探索序列空间,并加速抗生素候选分子的发现。
在研究中,研究团队共生成了5万条候选序列,随后利用自研的深度学习模型APEX进行筛选与排序,并合成了其中排名靠前的46条。实验结果表明,这些肽分子表现出广谱抗菌活性,对多重耐药菌株同样有效,同时展现出低细胞毒性。机制研究揭示,其主要作用模式为破坏细胞膜通透性和引起膜去极化。在小鼠的临床前模型中,优势肽能显著降低细菌负荷,其疗效可与多黏菌素B和左氧氟沙星相媲美,且未检测到不良反应。
因此,AMP-Diffusion为新型抗生素的设计提供了一个稳健且高效的平台。
1 引言
近年来,生成式人工智能(AI) 取得了重大进展,其中去噪扩散模型已成为计算机视觉、自然语言处理乃至蛋白设计等多个领域的有效工具。该类模型通过逐步将高斯噪声恢复为原始信号,为具有特定结构与功能特征的蛋白设计提供了全新思路。在蛋白设计中,基于扩散的方法如RFDiffusion已成功用于设计蛋白单体、结合物及对称寡聚体,展示了在探索庞大构象空间方面的优势。同样,基于图的扩散模型在抗体设计与蛋白–配体对接等特定任务中也取得了进展,通过对蛋白结构与相互作用进行图表示优化分子构型,从而提升抗体亲和力或配体对接性能。
除扩散方法外,蛋白语言模型(pLMs) 已成为分析与设计蛋白序列的重要工具。诸如ESM-2、ProtT5等模型在数百万天然蛋白序列上进行预训练,借助Transformer架构直接从序列中捕捉基本理化与功能属性。这一能力扩展到从头蛋白设计,能够生成保持功能性与生物活性的序列。例如,ProtGPT2和ProGen采用自回归预训练,而EvoDiff则引入扩散以支持新序列生成与支架设计。
在应用层面,抗菌肽(AMPs) 因其多样化作用机制(包括膜破坏、代谢抑制与免疫调节)被认为是传统抗生素的有力替代方案,在应对抗菌耐药性(AMR)方面具有巨大潜力。然而,AMP的发现与优化仍具挑战性。近年来,机器学习与深度学习在AMP生成中已发挥关键作用,通过识别大规模数据集中的功能模式显著加速了研究进展。
基于此背景,该研究提出了假设:结合表示学习与生成式语言建模有望实现稳健的AMP生成,并精确调控其理化与功能特性。为此,研究开发了AMP-Diffusion,一种预训练潜在扩散模型,通过在预训练的ESM-2-650M嵌入空间中施加高斯噪声完成前向/逆向扩散,从而生成具有功能性的AMP序列。与其他AMP扩散模型不同,AMP-Diffusion无需在蛋白潜在空间上额外训练,而是直接依赖经过广泛验证的ESM-2潜在表示,被公认为基于序列理解的“黄金标准”。此外,AMP-Diffusion在去噪器结构中复用了ESM-2的注意力层权重,使生成结果不仅在统计上合理,更能符合ESM-2学习到的复杂生物学规则。
最后,研究通过广泛的实验验证AMP-Diffusion生成的肽,包括结构特征、作用机制、细胞毒性以及动物感染模型中的疗效,系统性地证明了该模型的有效性与应用潜力。
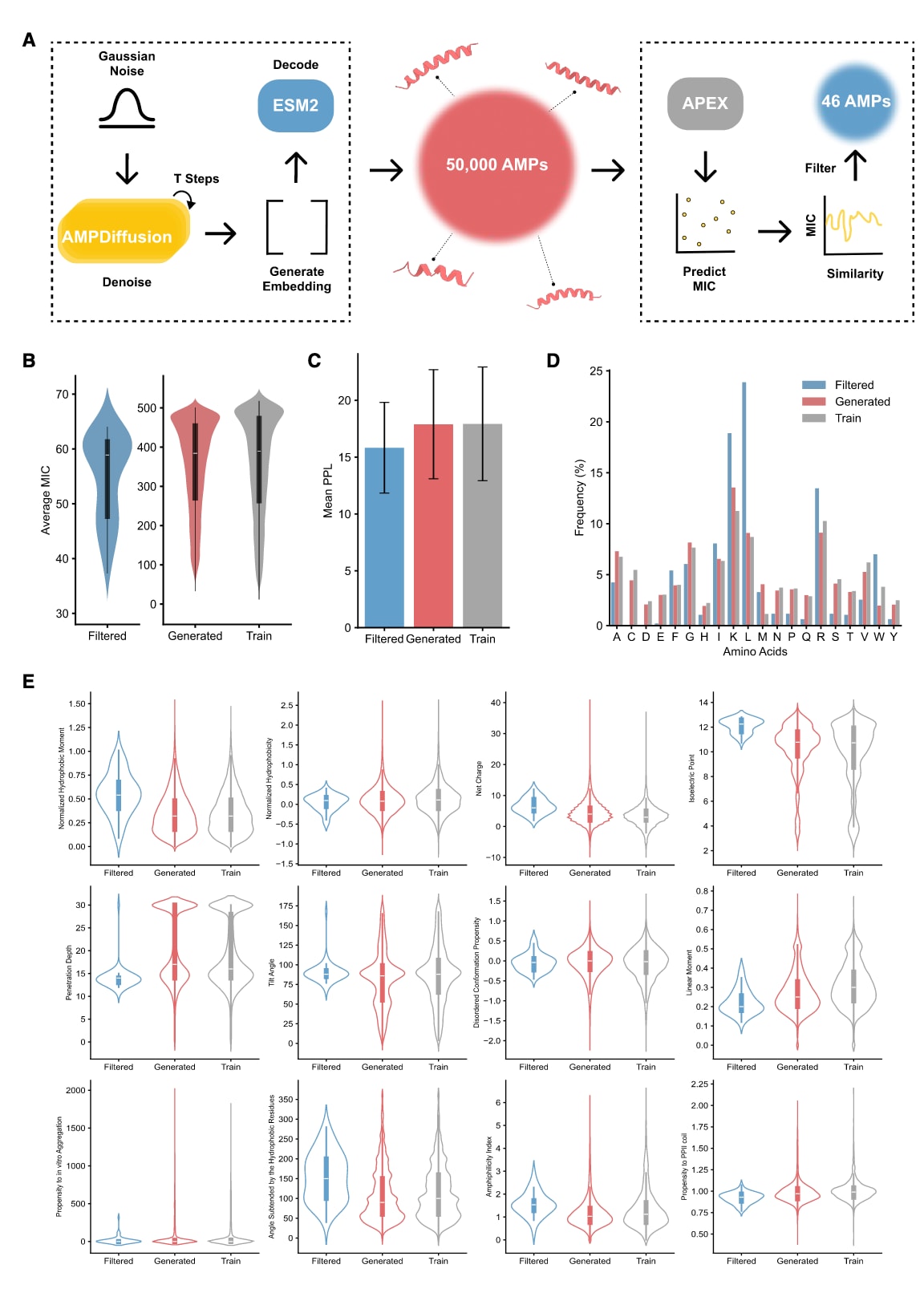
图1 | AMP-Diffusion模型及生成肽的特征 A,生成与筛选流程。 AMP-Diffusion模型以随机高斯噪声为输入,共生成50,000条候选序列。生成的嵌入矩阵M通过ESM-2-650M语言模型头解码为氨基酸序列。随后,所有序列均使用APEX模型预测其对11种细菌的最小抑菌浓度(MIC)。基于平均MIC值(μmol L⁻¹)与序列同一性标准,最终筛选得到46条候选肽。B,MIC值分布。 小提琴图展示了在筛选集、生成集与训练集中的平均MIC分布(单位:μmol L⁻¹)。C,困惑度(PPL)对比。 柱状图比较了筛选集、生成集与训练集的PPL值,误差条表示±1标准差。D,氨基酸组成分布。 展示了三组数据(筛选集、生成集与训练集)的氨基酸分布差异。E,理化性质分布。 对比了三组数据在关键理化特征上的分布情况。
2 结果与讨论
2.1 计算框架
作者开发了 AMP-Diffusion,一种专为抗菌肽(AMP)生成而设计的潜在空间扩散模型。该模型通过在 ESM-2嵌入的潜在空间中应用扩散过程,逐步加入并去除噪声,从而生成功能性AMP序列。AMP-Diffusion 在一个整合数据集上进行训练,该数据集来自 DRAMP 3.0、APD3 和 DBAASP 数据库(Dataset S1)。
评估结果表明,AMP-Diffusion 生成的肽在 伪困惑度(pseudo-perplexity) 和 氨基酸残基多样性 上与实验验证的AMP高度一致。这些生成序列还表现出与天然AMP相当的理化性质,表明其具有生物学合理性和潜在的实验可验证性。更多模型推导与细节参见 Chen 等人(参考文献27)。
在该研究中,研究者假设经过预训练的 AMP-Diffusion 模型能够生成功能强效的AMP。从初始的 50,000 条候选序列(Dataset S2)中,研究者基于三个标准筛选了46条肽用于实验验证:(1) 预测的抗菌活性较高,(2) 与现有AMP相似度较低,(3) 序列多样性广泛(图1A,详细筛选标准见方法部分的计算过滤步骤)。结果显示,生成序列的 最低抑菌浓度(MIC)分布 与训练集相当,表明模型成功复现了训练集AMP的MIC特征(图1B与图S1)。
为了评估生成肽的“天然性”,研究者使用了 ProGen2 ——一个在数百万天然蛋白质序列上预训练的蛋白语言模型。天然性通过 困惑度(perplexity) 进行量化,值越低代表天然性越高。结果显示,研究者生成的序列困惑度为 17.90(过滤后为15.83),与训练集的 17.93 非常接近(双尾Mann–Whitney U检验 p = 0.0357)(图1C),这表明 AMP-Diffusion 生成的肽具备与天然AMP相当的天然性。
2.2 预测肽的组成与理化特性
生成的AMP库在氨基酸组成上与训练集高度相似,说明模型成功捕捉到了已知AMP的组成特征(图1D)。有趣的是,经过过滤的生成序列子集表现出独特模式,赖氨酸(K)、亮氨酸(L)和精氨酸(R)的频率显著升高。已知这些残基在以下方面至关重要:
- K 和 R:与带负电的细菌膜进行初始静电相互作用;
- K、L 和 R:决定两亲性,有助于有效与脂质膜相互作用。
因此,带正电和脂肪族残基的富集直接塑造了AMP-Diffusion预测肽的理化特性(图1E与图S2),使得过滤后的子集表现出更强的两亲性与电荷特征,具有更高的结构趋向性和增强的抗菌活性。
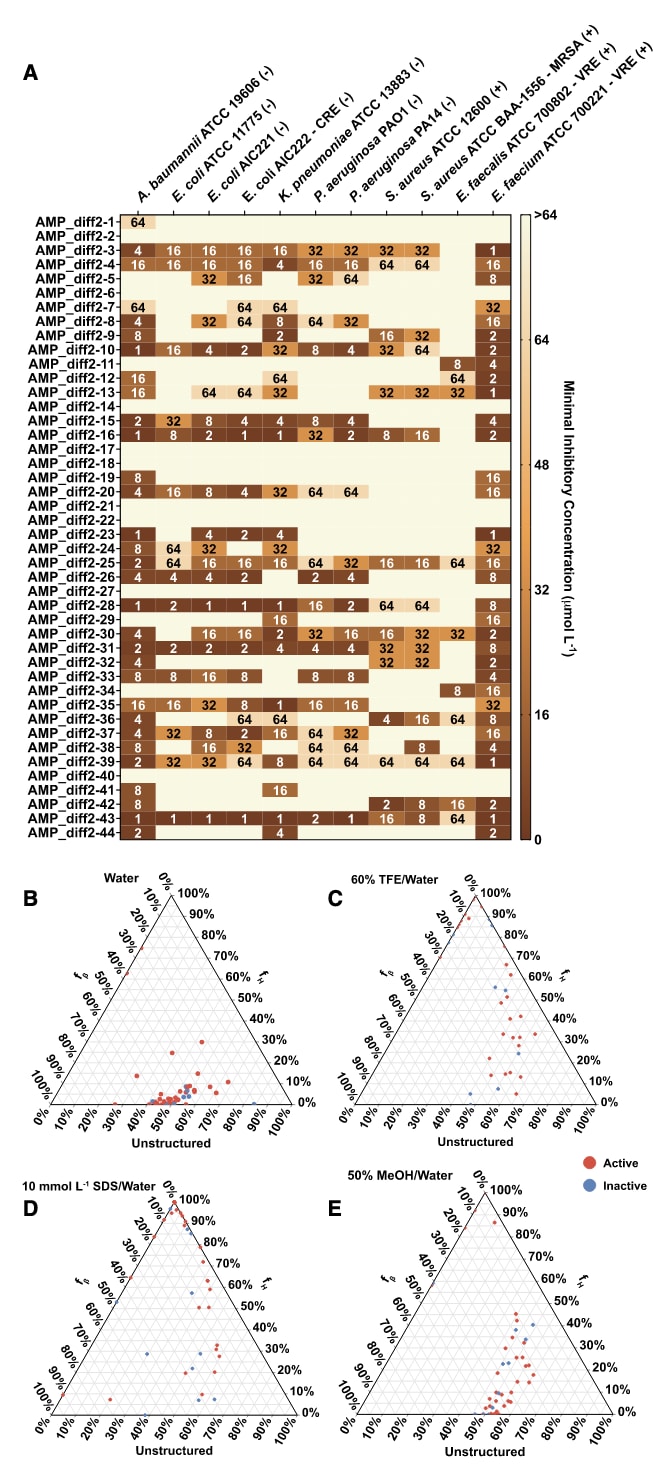
图2 | AMP-Diffusion生成肽的抗菌活性与二级结构 (A) 合成肽对11种临床相关病原体的抗菌活性(μmol L⁻¹)热图,其中包括4株对常规抗生素耐药的菌株。实验中,将约10⁶个细菌细胞与系列稀释的肽(1–128 μmol L⁻¹)在37 °C下孵育。处理后1天,在酶标仪上测定600 nm处的光密度以评估肽存在条件下的细菌生长情况。热图中的MIC值取自每个条件下三次重复实验的众数。(B) 三元图展示了每条肽(50 μmol L⁻¹)在四种不同溶剂中的二级结构比例:水、60% 三氟乙醇(TFE)/水、十二烷基硫酸钠(SDS, 10 mmol L⁻¹)/水、50% 甲醇(MeOH)/水(参见图S3)。二级结构比例通过BeStSel服务器计算。红点表示具有抗菌活性的肽,蓝点表示无活性的肽(参见图S4)。
2.3 体外抗菌活性
为验证模型的预测准确性,研究者合成并实验测试了 46 条肽序列。合成对象的选择基于预测的抗菌活性和序列多样性,以确保生成肽空间的广泛代表性(详见方法部分的 APEX 模型与计算过滤)。结果显示,46 条合成肽中有 35 条(76%)对至少一种临床相关的 11 种细菌病原体具有活性,其中包括 ESKAPEE 病原体清单中的成员(图2A)。大多数肽表现出广谱活性,其中 鲍曼不动杆菌(A. baumannii ATCC 19606) 和 耐万古霉素的屎肠球菌(E. faecium ATCC 700221) 是最为敏感的菌株。
2.4 二级结构
AMPs 通常在与细菌膜相互作用时形成 α-螺旋构象。这种结构转变受到 两亲性、净电荷、疏水性和肽长度 的影响。为评估不同生化环境下的二级结构,研究者在以下条件下测试了每条肽:
- 水溶液(模拟细胞外环境);
- 三氟乙醇/水 (3:2, v/v)(诱导螺旋);
- 甲醇/水 (1:1, v/v)(诱导 β-结构);
- 10 mmol L⁻¹ 十二烷基硫酸钠 (SDS) 胶束溶液(模拟脂质双层环境)。
结果表明,与已知AMP一致,这些肽因其较高的赖氨酸(K)、亮氨酸(L)和精氨酸(R)含量而表现出 螺旋–无规卷曲转变:
- 在螺旋诱导溶液和脂质双层环境中形成 α-螺旋;
- 在水和甲醇/水混合液中保持无规卷曲状态(图2B–2E, 图S3)。
2.5 作用机制
细菌膜是大多数AMP的常见作用靶点,这是因为AMP与脂质双层存在 非特异性相互作用。AMP的抗菌活性受到其氨基酸组成、分布,以及理化特性(如两亲性、疏水性)的影响。
首先,研究者检测了其对鲍曼不动杆菌外膜的通透作用,采用 NPN(1-苯胺基萘)荧光探针进行测试。NPN 在脂质环境中的荧光强度高于水环境,因此可用于评估膜完整性。实验结果显示,所有测试肽的活性至少与对照抗生素 多黏菌素B 和 左氧氟沙星 相当。其中 AMP-diff2-1、-13、-32、-33 和 -38 表现出尤为强烈的外膜通透作用(图3A, 图S4A)。这表明这些肽在破坏细菌膜方面优于许多已报道的AMP以及来源于人类蛋白、已灭绝生物或细菌宏基因组的加密肽(EPs)。
接着,研究者评估了其对 胞质膜去极化作用。使用电位敏感染料 DiSC3-5,其在膜去极化时荧光增强。结果表明,这些肽的去极化作用强于多黏菌素B和左氧氟沙星,与已知的高效去极化EPs相当。AMP-diff2-13、-30、-32、-33 和 -42 是最强的去极化剂(图3B, 图S4B)。
2.6 细胞毒性实验
所有 46 条合成肽均在 人胚胎肾细胞(HEK293T) 中测试其细胞毒性,该细胞系广泛用于毒性研究。结果显示:
- 34 条肽在测试浓度范围(8–128 μmol L⁻¹)内未表现出显著毒性;
- 12 条肽表现出一定毒性,其中仅有 6 条在低于 64 μmol L⁻¹(细菌抑制实验的最高浓度)时即表现出毒性。
这些结果进一步强调了该类肽的 整体安全性良好。此外,通过非线性回归估算了 CC50(导致 50% 细胞毒性的肽剂量)(图3C)。
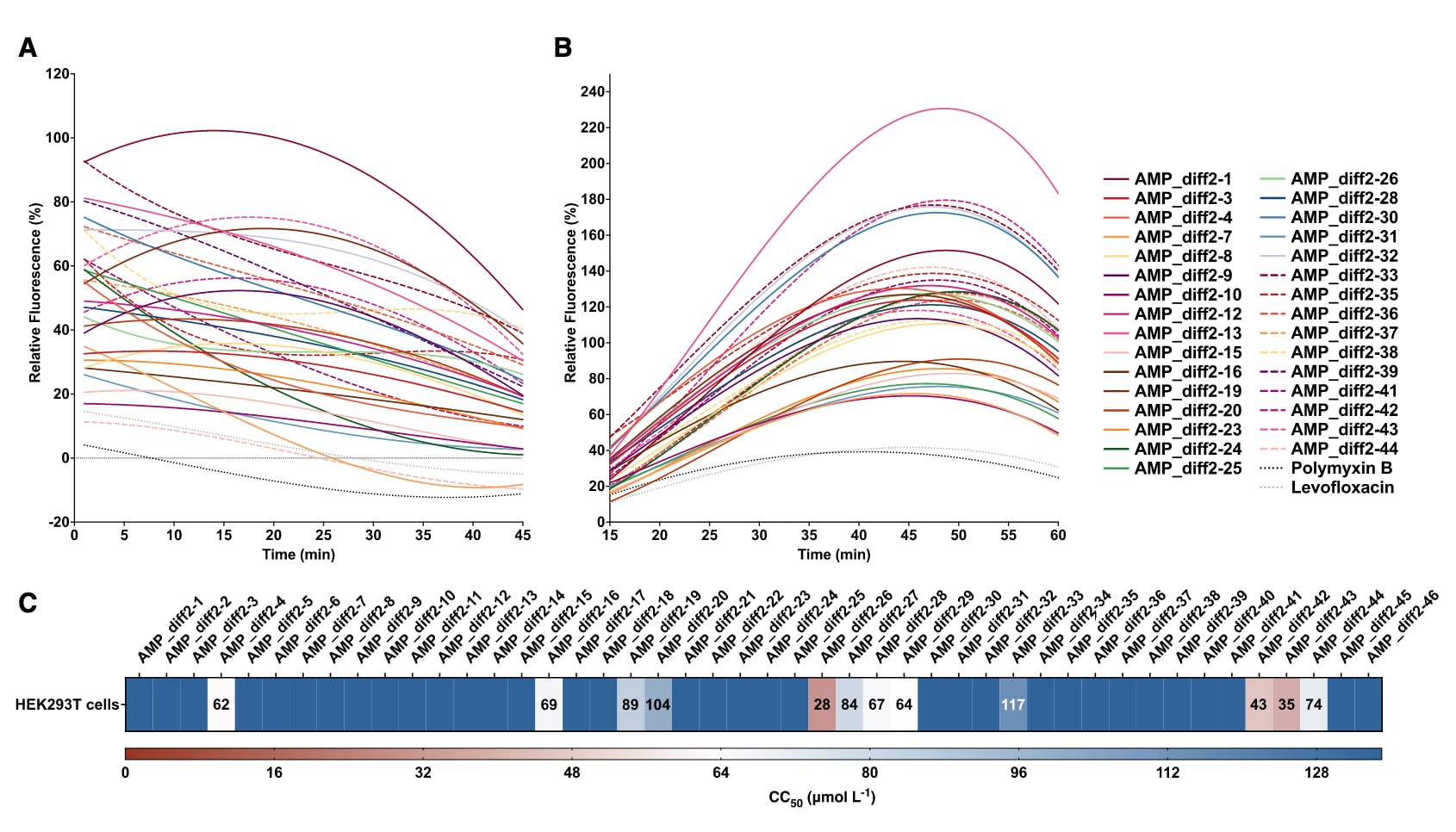
图3 | 合成肽的作用机制与细胞毒性活性 (A, B) 为评估合成肽是否作用于细菌膜,对所有对鲍曼不动杆菌 A. baumannii ATCC 19606 显示活性的肽进行了外膜通透化实验和细胞质膜去极化实验(参见补充图 S4)。荧光探针 1-(N-苯基氨基)萘 (NPN) 被用于检测由6条主要穿透型肽诱导的外膜通透化效应 (A);荧光探针 3,3′-二丙基噻二氰菁碘化物 (DiSC3-5) 则用于评估由6条主要去极化型肽引起的细胞质膜去极化 (B)。图中数值表示两种探针的相对荧光强度,采用非线性拟合方法与未处理对照组(缓冲液+细菌+荧光染料)基线进行比较,并以抗生素多粘菌素B与左氧氟沙星作为参考标准(所有对A. baumannii ATCC 19606 显示抗菌活性的肽的完整结果见补充图 S3)。(C) 通过非线性回归曲线插值剂量–反应数据,确定导致 50% 细胞裂解 (CC50) 的细胞毒性浓度。所有实验均独立重复三次。
2.7 动物模型中的抗感染功效
为评估活性肽在体内是否具有抗感染功效,研究者采用了小鼠皮肤脓肿感染的临床前模型。39–43 其中两条在体外对 鲍曼不动杆菌(A. baumannii ATCC 19606) 表现出高活性的肽(AMP-diff2-16 和 AMP-diff2-43,MIC = 1 μmol L⁻¹),在感染建立后以 单次给药、10 倍 MIC 浓度(10 μmol L⁻¹) 进行测试。每次处理为 20 μL 局部注射,分别对应约 24 μg kg⁻¹(AMP-diff2-16) 和 26 μg kg⁻¹(AMP-diff2-43) 的剂量。作为对照,常用抗生素 多黏菌素B 和 左氧氟沙星 也以相同浓度(10 μmol L⁻¹)给药,对应剂量分别为 13.9 μg kg⁻¹ 和 3.6 μg kg⁻¹。
在该模型中,小鼠通过皮下注射感染鲍曼不动杆菌(图4A)。随后将单剂量的候选肽直接注射至感染部位。两天后,两条肽均使细菌负荷下降了约1.5个数量级(图4B)。四天后,细菌数量下降了2–2.5个数量级,效果与对照抗生素多黏菌素B和左氧氟沙星相当(图4B)。在治疗过程中,小鼠未出现体重变化、皮肤损伤或其他不良反应(图4C)。这些显著的体内结果表明,AMP-Diffusion生成的肽在生理相关条件下展现出与标准抗生素相当的抗感染效果,显示出作为有效抗感染剂的潜力。
2.8 研究局限性
当前版本的 AMP-Diffusion 基于 经典的去噪扩散概率模型(DDPM)框架,1 与更先进的 阐明型扩散模型(EDM44 与 EDM245) 相比,在生成质量与计算效率上仍未优化。此外,AMP-Diffusion 作为无条件扩散模型运行,缺乏条件引导机制。未来研究可探索为加噪序列训练专用分类器,以便基于关键治疗特性(如抑制活性与毒性)来引导生成;或者采用无分类器引导,结合病原体特异性与治疗特性嵌入,生成针对特定细菌株且安全性更高的肽。
研究者认为,离散扩散架构(如 掩码离散扩散 MDLM 和 离散去噪后验预测 DDPP)将是下一代 AMP-Diffusion 模型的理想候选方案。46–48 结合这些创新有望建立新一代框架,进一步优化肽设计,提高抗菌功效与安全性。
另一方面,公开可用的 AMP 数据库存在显著的组成异质性,涵盖作用于多种靶标和分子对象的肽。例如,在 DBAASP 数据库中,研究重点明显失衡,针对 革兰氏阳性和阴性菌 的肽数量远多于针对真菌、病毒、寄生虫或癌细胞的肽。这种多样性还扩展到非生物结构,如脂质双层(许多膜破坏性肽的常见靶点)。虽然这种广度为 AMP 研究提供了全面视角,但其固有的不平衡性会对开发无偏计算模型带来挑战。未来若研究需避免这种异质性,可通过 战略性下采样 构建更均衡、更聚焦于特定生物体或作用机制的数据集。
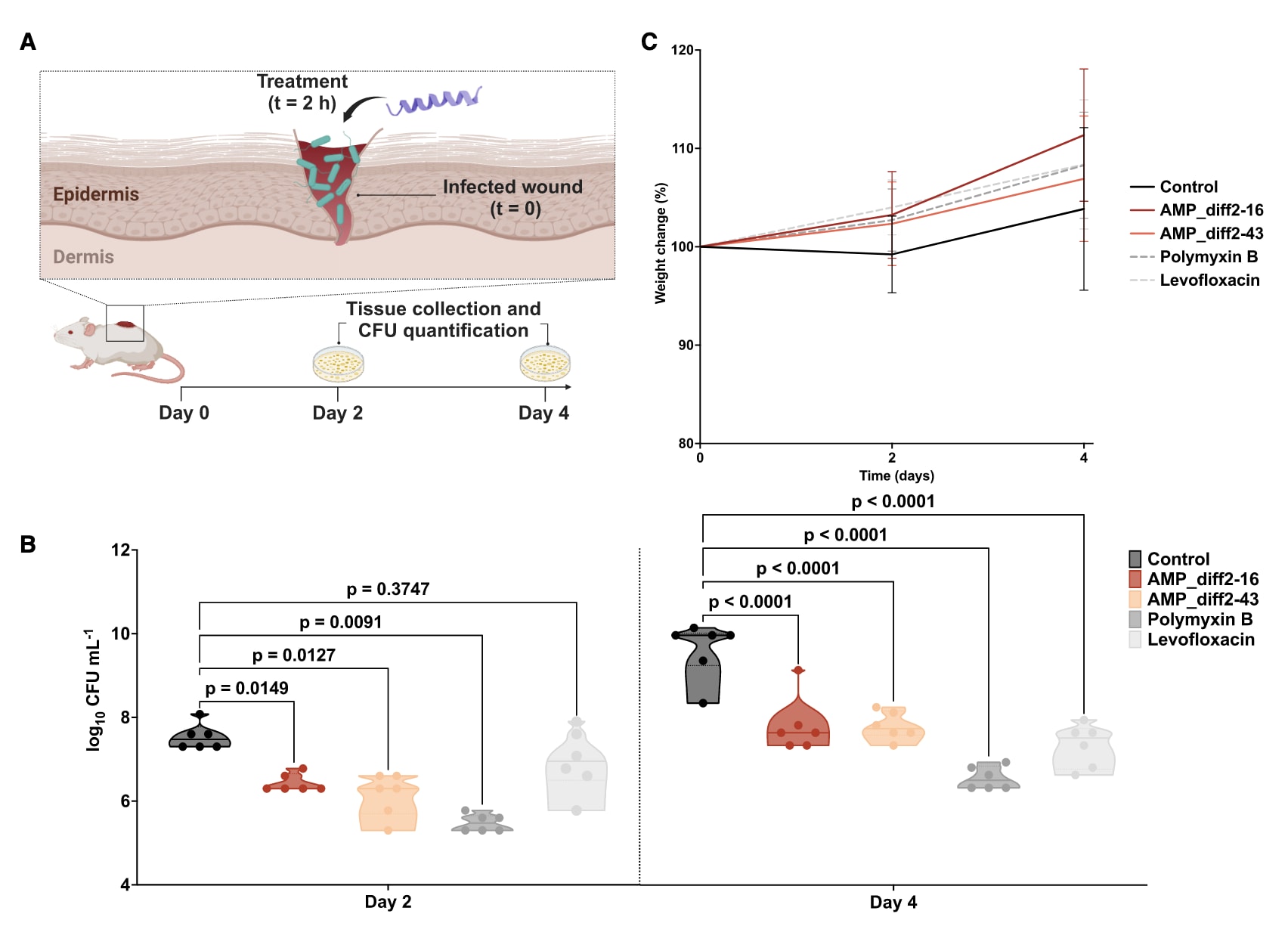
图 4 | 小鼠感染模型中肽的抗感染活性 (A) 皮肤脓肿小鼠模型示意图,用于评估所选肽(n = 6)对 A. baumannii ATCC 19606 的抗菌活性。(B) AMP-diff2-16 和 AMP-diff2-43 两种肽以 10 μmol L⁻¹ 的单次剂量给药,每次处理通过 20 μL 局部注射完成,分别对应约 24 μg kg⁻¹(AMP-diff2-16)和 26 μg kg⁻¹(AMP-diff2-43)。作为对照,抗生素多黏菌素 B 和左氧氟沙星也以相同浓度(10 μmol L⁻¹)给药,剂量分别为 13.9 μg kg⁻¹ 和 3.6 μg kg⁻¹。结果显示,这两种肽在给药后 最长可抑制细菌增殖 4 天,与未处理对照组相比具有显著效果。两种肽的抑菌水平与抗生素对照(多黏菌素 B 和左氧氟沙星)相当。(C) 在皮肤脓肿模型持续的 4 天实验期间,监测小鼠体重,以评估细菌负荷或肽处理可能带来的毒性。B 中的统计学显著性采用 **单因素方差分析(one-way ANOVA)**并结合 Dunnett’s 检验,p 值如图中所示。
3 方法
3.1 AMP-Diffusion
AMP-Diffusion 是一种基于 DDPM 框架 的潜在空间扩散模型,专门用于生成抗菌肽(AMP)。它利用最新的 ESM-2 蛋白语言模型(pLM) 将肽序列映射到连续潜在空间,在前向过程中系统地加入高斯噪声。模型随后训练一个去噪器,从加噪后的输入中重建原始潜在嵌入,并通过最小化预测嵌入与原始嵌入之间的
在生成阶段,反向过程以高斯噪声为起点,逐步去噪以生成与原始数据分布相似的样本。去噪架构采用预训练的 ESM-2-8M 注意力块,并结合位置时间嵌入与**多层感知机(MLP)**进行最终处理。
在该研究中,模型重新基于一个清洗并扩展后的数据集(共19,670条肽,见数据集 S1)进行训练,数据来源包括 DRAMP、DBAASP 与 APD3。训练中引入了 指数滑动平均(EMA)。具体而言,在扩散模型训练的同时,维护一份“慢速更新”的权重副本。在每次训练步骤中,主模型的权重由优化器更新,而 EMA 权重则按如下规则进行更新:
其中,
除上述改动外,其他参数与方法均保持与原始模型架构一致。关于更为全面的技术细节,建议参考原始研究。
3.2 APEX模型
采用了 APEX,一种针对特定细菌菌株的抗菌活性预测器,用于对生成的肽序列进行排序和筛选 https://gitlab.com/machine-biologygroup-public/apex-pathogen 。由于APEX能够预测针对11种病原菌株的抗菌活性,因此在该研究中以其预测的平均最小抑菌浓度(MIC)对AMP-Diffusion生成的肽进行排序。未被多样性和相似性筛选标准(见计算过滤部分)剔除的高分肽被选中用于合成和实验验证。
3.3 计算过滤
为确保入选验证的序列满足以下条件:(a) 具有较高的抗菌活性;(b) 与已知AMPs足够不同;(c) 覆盖较广的序列空间,应用了三重过滤标准:
- 生成肽的平均MIC必须 ≤ 64 μmol L⁻¹;
- 与已知AMP的相似性:采用局部序列比对方法,计算生成肽与来自DRAMP 3.0、DBAASP、APD3以及自有库存的整理AMP集合之间的序列相似性。若相似度 > 0.60,则认为过于接近,予以剔除;
- 对剩余肽进行成对序列相似性比较,若两者相似度 > 0.40,则保留预测平均MIC更低的一条(由APEX估算)。
3.4 ProGen2与困惑度(Perplexity, PPL)
ProGen2 是一组用于蛋白序列生成与理解的蛋白语言模型(pLMs),基于自回归语言建模。其模型规模从 1.51亿到64亿参数不等,以适应不同计算需求。该模型采用标准的Transformer解码器,具备从左到右的因果掩码,并结合旋转位置编码。训练数据包括来自UniRef90、UniProtKB和BFD30的多源蛋白序列,因此能够学习不同蛋白家族和结构间的模式与关系。
困惑度用于衡量概率模型预测样本的能力,在pLMs中常用于评估序列质量。定义如下:
其中
3.5 理化特性
对AMP-Diffusion生成的肽与天然AMP(训练数据)进行了理化性质比较。使用DBAASP服务器计算了包括归一化疏水矩、归一化疏水性、净电荷、等电点、穿透深度、倾角、无序构象倾向、线性矩、体外聚集倾向、疏水残基夹角、两亲性指数及PPII螺旋倾向在内的一系列理化指标。
3.6 肽的合成
肽由AAPPTec公司提供,采用 Fmoc固相肽合成策略合成。
3.7 细菌菌株与培养
在MIC测定和作用机制实验中所使用的菌株包括:鲍曼不动杆菌 Acinetobacter baumannii ATCC 19606、大肠杆菌 Escherichia coli ATCC 11775、E. coli AIC221(E. coli MG1655 phnE_2::FRT,AIC222的对照株)和 E. coli AIC222(E. coli MG1655 pmrA53 phnE_2::FRT,多黏菌素耐药株;耐黏菌素株)、肺炎克雷伯菌 Klebsiella pneumoniae ATCC 13883、铜绿假单胞菌 Pseudomonas aeruginosa PAO1、PA14、ATCC 35032、金黄色葡萄球菌 Staphylococcus aureus ATCC 12600、ATCC 6538、ATCC BAA-1556(耐甲氧西林株)、屎肠球菌 Enterococcus faecalis ATCC 700802(耐万古霉素株)、粪肠球菌 Enterococcus faecium ATCC 700221(耐万古霉素株)。
所有菌株均从冷冻保存物复苏,在相应培养基平板上培养过夜:Luria-Bertani (LB) 培养基(常规菌株)、假单胞菌选择培养基(用于 P. aeruginosa)、以及MacConkey 培养基(用于 A. baumannii),37℃培养过夜。培养后,挑取单个菌落接种于5 mL LB液体培养基,在37℃下摇床培养16 h。次日,将过夜培养物按1:100稀释至5 mL新鲜培养基中,继续培养至对数生长期(OD600 = 0.3–0.5)。
3.8 MIC测定
采用肉汤微量稀释法进行MIC测定。首先将肽溶液加入未处理的聚苯乙烯96孔微量滴定板中,并以无菌水进行两倍系列稀释,最终浓度范围为1–64 μmol L⁻¹。随后,制备密度为
3.9 外膜通透化实验
采用 NPN摄取实验检测肽对细菌外膜的通透化能力。将 A. baumannii ATCC 19606 培养至 OD600 = 0.4,随后以10,000 rpm离心10 min(4℃),用缓冲液洗涤,并重悬于5 mmol L⁻¹ HEPES缓冲液(pH 7.4,含5 mmol L⁻¹葡萄糖)中。取100 μL细菌悬液加入白色96孔板中,再加入4 μL NPN溶液(终浓度0.5 mmol L⁻¹)。随后在各孔中加入稀释的肽溶液,记录45 min内的荧光信号,激发波长λ_ex = 350 nm,发射波长λ_em = 420 nm。
相对荧光强度以未处理对照组(缓冲液+细菌+荧光探针)为基线计算,百分比差值的计算公式如下:
其中
3.10 细胞质膜去极化实验
采用膜电位敏感染料 DiSC3-5评估肽的作用。将 A. baumannii ATCC 19606 在对数中期收获,经洗涤后重悬于 HEPES 缓冲液(pH 7.2),其中含 20 mmol L⁻¹ 葡萄糖和 0.1 mol L⁻¹ KCl,浓度为 0.05 OD600 / mL。随后向细菌悬液中加入 DiSC3-5(终浓度 20 μmol L⁻¹),孵育15 min 以使荧光稳定(表明染料嵌入细菌膜)。然后将肽与细菌悬液按 1:1 比例混合,使其最终浓度相当于各自的MIC。通过监测荧光变化(λ_ex = 622 nm,λ_em = 670 nm)持续 60 min 评估膜去极化。相对荧光以未处理对照组(缓冲液 + 细菌 + 染料)为基线计算,百分比差值公式如下:
3.11 圆二色谱 (CD) 实验
使用 J1500 CD 光谱仪(Jasco)在宾夕法尼亚大学生物化学资源中心 (BCRC) 进行。实验在 25℃ 下进行,每条光谱为三次累积的平均值。采用光程 1.0 mm 的石英比色皿,扫描范围 260–190 nm,扫描速率 50 nm/min,带宽 0.5 nm。肽浓度为 50 μmol L⁻¹,在四种溶剂条件下检测:水、三氟乙醇/水 (3:2, v/v)、SDS 水溶液 (10 mmol L⁻¹) 以及甲醇/水 (1:1, v/v)。测量前分别记录相应基线。应用傅里叶变换滤波以降低背景噪音。二级结构分数由 BeStSel 单光谱分析工具计算。三元图使用 https://www.ternaryplot.com/ 生成并进一步可视化。
3.12 真核细胞培养与细胞毒性实验
从 ATCC 获得 HEK293T 细胞(CRL-3216)。细胞培养于高糖型 DMEM 培养基,补充 1% 青链霉素(抗生素)和 10% 胎牛血清,在 37℃、5% CO₂ 饱和湿润环境中生长。实验前一天,将密度为 50,000 个细胞/mL 的细胞悬液(100 μL)接种于经处理的96孔板(即每孔 5,000 个细胞)。贴壁的 HEK293T 细胞随后暴露于不同浓度的肽 (8–128 μmol L⁻¹) 24 h。孵育后进行 MTT 细胞活力实验:将 0.5 mg/mL 的 MTT 溶液(不含酚红培养基)替换细胞上清液(100 μL/孔),37℃、5% CO₂ 下孵育4 h,生成不溶性 formazan 晶体。随后用 0.04 mol L⁻¹ 盐酸/无水异丙醇溶解,并在 570 nm 处测定吸光度。所有实验均进行三次独立生物学重复。
3.13 皮肤脓肿小鼠感染模型
选用6周龄雌性 CD-1 小鼠。小鼠麻醉后剃背毛,并用乙醇擦拭以清除皮肤表面细菌。随后使用手术刀在皮肤上划痕。取 A. baumannii ATCC 19606 悬液(10⁶ CFU/mL,20 μL,培养至 OD600=0.5,PBS洗涤两次,5000 g 离心3 min)接种至创伤处。感染建立 2 h 后,在创口处给予多肽、多黏菌素B或左氧氟沙星,浓度均为 10 × MIC。感染后第2天和第4天处死小鼠,切取感染皮肤组织,利用震荡破碎仪 (25 Hz,20 min) 匀浆,10倍系列稀释后涂布于 MacConkey 平板,计数 CFU。每组纳入 6 只小鼠。所有动物实验程序经宾夕法尼亚大学 ULAR 审批(protocol 806763)。
3.14 统计分析
所有数据均来源于至少三次独立实验,结果以中位数及上下四分位数表示。图4中的统计显著性通过**单因素方差分析(one-way ANOVA)**计算。所有统计分析均使用 GraphPad Prism v.10.0.3 软件完成。显著性差异以 p 值 表示。