NMI 2025 | GraphPep: 一种基于相互作用图学习的蛋白质–肽复合物评分框架

获取详情及资源:
0 摘要
精确预测蛋白质–肽相互作用对于肽类药物的研发至关重要。然而,由于蛋白质数据银行(Protein Data Bank, PDB)中可用的蛋白–肽复合物结构数量有限,训练出高精度的评分函数一直是个挑战。为解决这一问题,研究提出了一种基于相互作用特征的图神经网络框架——GraphPep,用于蛋白质–肽复合物的评分。与传统方法将原子或残基作为图节点不同,GraphPep以蛋白–肽相互作用为节点进行建模,并在损失函数中关注残基–残基接触关系,而非单一的肽均方根偏差(RMSD)。因此,该模型不仅能够高效捕捉关键的蛋白–肽相互作用特征,还能缓解训练数据稀缺带来的问题。此外,GraphPep通过结合ESM-2蛋白语言模型进一步提升了性能。研究在多个由不同蛋白–肽对接程序及AlphaFold生成的诱饵数据集上进行了广泛验证,并与当前最先进方法进行了比较,结果表明GraphPep在精度与稳健性方面均表现优异。
1 引言
肽与蛋白质之间的相互作用约占所有蛋白质间相互作用的40%,这些相互作用参与了包括免疫反应、基因表达及信号传递等多种细胞功能。因此,蛋白–肽相互作用因其在治疗学上的潜力而受到广泛关注,因为肽分子可以通过干预关键相互作用来调节蛋白质功能。蛋白–肽复合物结构为理解其物理化学相互作用提供了重要基础,但实验测定往往耗时且昂贵,因此计算建模方法显得尤为重要。肽分子本身的柔性特性进一步增加了实验研究的难度,使得其构象与相互作用的精确刻画和预测更具挑战性。
为应对这一问题,研究者开发了多种用于预测蛋白–肽复合物结构的对接方法。对接通常包括两个阶段:采样阶段生成大量候选复合物模型(称为诱饵),评分阶段对这些模型进行评估与排序以筛选最准确的结构。评分函数的精度决定了整个对接方法的表现。然而,由于实验解析的蛋白–肽结构数量有限,针对蛋白–肽相互作用专门设计的评分函数仍然稀缺。目前的评分方法主要包括基于物理势能、统计势能、机器学习以及深度学习四类。物理势能方法通过范德华力、电荷作用或疏水能等能量项来描述分子间相互作用;统计势能方法则通过分析实验结构中的统计信息来推导相互作用势。随着机器学习与深度学习的快速发展,新的评分函数不断涌现。机器学习模型常利用随机森林或支持向量机等算法,通过多样化特征区分接近天然构象与非天然构象,但其性能高度依赖特征工程与训练集质量。深度学习方法主要包括基于卷积神经网络(CNN)与图神经网络(GNN)的两大框架。前者将复合物界面离散化为三维网格进行特征学习,如DOVE与DeepRank模型。然而,CNN在处理三维结构时对输入取向敏感,且局部感受野限制了其捕捉远程相互作用的能力。相比之下,GNN通过节点与边的消息传递学习复杂的相互作用模式,具有旋转不变性与更强的表达能力。
随着蛋白语言模型的发展,将其嵌入特征引入GNN节点以捕捉演化与功能信息,进一步提升了评分性能。现有的GNN评分模型通常以残基或原子为节点,以其接触关系为边,通过网络处理后进行图级预测,如评估诱饵的原生接触比例(Fnat)、局部距离差异测试(LDDT)、DockQ分数,或依据iRMSD与CAPRI标准判断诱饵正确性。然而这些模型仍面临若干问题:其一,在蛋白–肽复合物中,由于分子体积差异显著,较大的蛋白部分往往主导整体预测;其二,图级预测需大量多样诱饵训练,易造成正负样本不平衡;其三,残基级图忽略关键原子相互作用,而原子级图在非结合态受体对接中表现不佳;此外,分子内与分子间相互作用在同一网络中学习,可能削弱对界面相互作用模式的捕捉;最后,PDB中蛋白–肽结构数量稀少,也限制了高精度评分函数的训练。
为解决上述难题,研究提出了GraphPep模型,一种用于蛋白–肽相互作用预测的图神经网络框架。GraphPep以原子与残基层面构建基于相互作用的图结构,将蛋白与肽在界面的相互作用视为节点,当两种相互作用共享同一残基或原子时定义为一条边。通过残差门控图卷积网络进行消息传递后,GraphPep在节点级进行分类,从而显式学习界面相互作用特征。为缓解训练数据稀缺问题,模型在损失函数中关注残基–残基接触,而非仅依赖肽的均方根偏差(RMSD)。GraphPep在多种由不同对接算法及AlphaFold-Multimer生成的诱饵数据集上进行了系统评估,展示了优异的精度与稳健性。此外,在利用AlphaFold3强制采样生成的抗体–肽测试集中,GraphPep同样表现出卓越的预测性能。
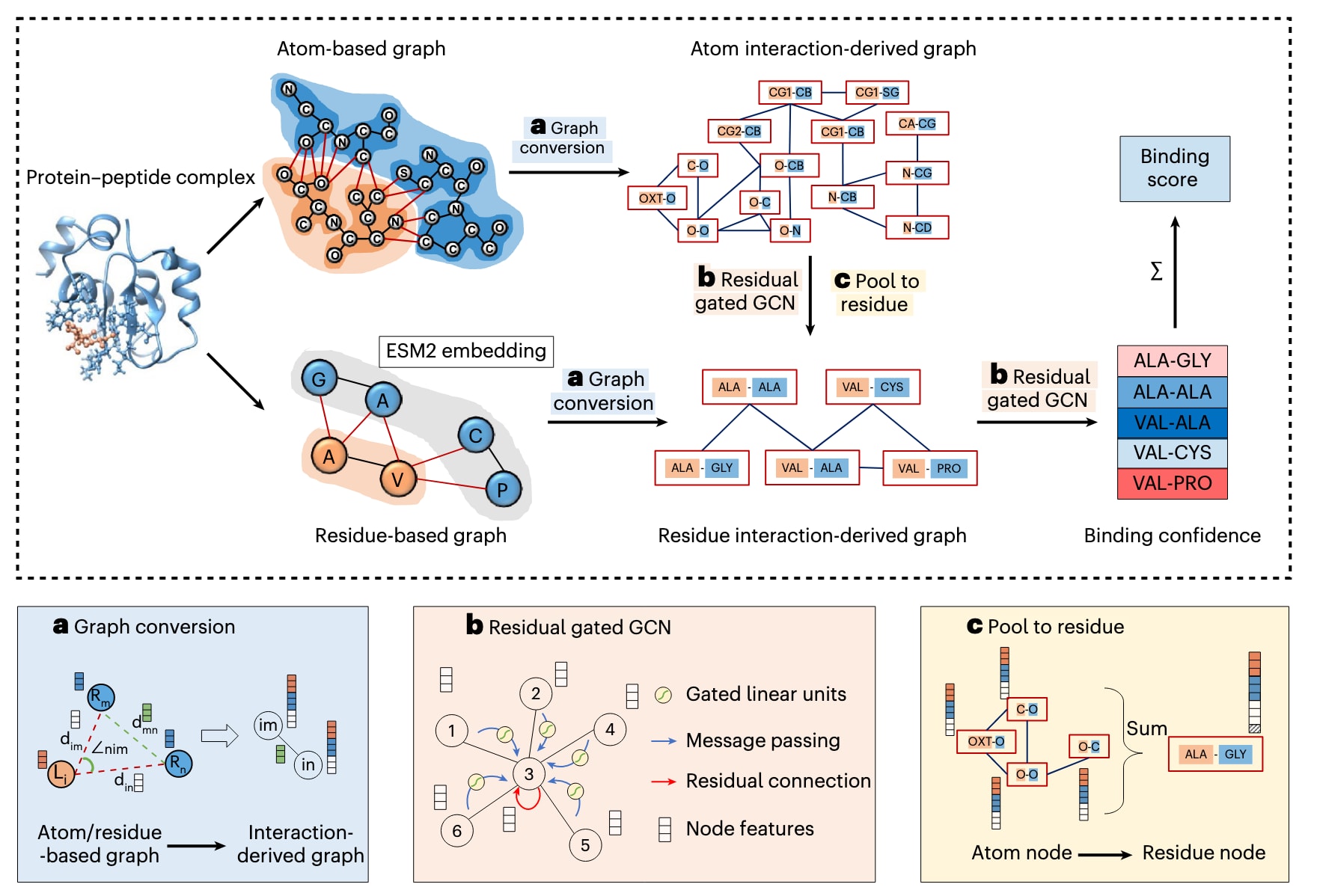
图1 | GraphPep框架示意图。 蛋白–肽复合物的原子层面与残基层面界面特征首先被提取并转换为基于相互作用的衍生图,从而使模型能够感知三角关系结构(triangle-aware),并仅保留代表蛋白–肽相互作用的节点。a,模型在原子层与残基层的相互作用衍生图上均应用残差门控图卷积网络(Residual Gated Graph Convolution)。b,原子层节点的特征被汇聚到对应的残基层节点中,用于进一步表征界面特征。c,最终,GraphPep在残基层图上执行节点分类,并整合各节点的置信度得分,计算出蛋白–肽结合构象的最终结合评分。
2 结果
2.1 GraphPep的工作流程
图1展示了GraphPep的整体工作流程。给定一个蛋白–肽复合物结构后,首先在原子层面与残基层面分别构建两种传统图结构,并嵌入来自ESM(Evolutionary Scale Modeling)蛋白语言模型的特征。接下来,这两种图被转换为基于相互作用的衍生图。在这一过程中,原始图中代表蛋白–肽相互作用的边被重新定义为衍生图中的节点,而在原始图中共享同一顶点的两条边,则在衍生图中形成一条新的边(见图1a)。
在深度学习阶段,模型在原子层与残基层的相互作用衍生图上分别执行残差门控图卷积(Residual Gated Graph Convolution)(见图1b)。随后,更新后的原子层图节点嵌入被汇聚到残基层图节点中,作为额外的节点特征输入(见图1c)。在残基层图中,每个节点对应一个蛋白与肽之间的残基–残基相互作用,其节点嵌入用于计算对应的置信度得分。最终,模型的结合评分通过对不同置信度阈值下的节点数量进行加总获得,从而综合反映蛋白–肽结合界面的整体相互作用强度。
2.2 在由对接程序生成的诱饵数据集上的性能
首先,利用由FlexPepDock、HADDOCK和HPEPDOCK三种对接程序生成的诱饵数据,对GraphPep的性能进行了评估。测试基于两个基准数据集:LEADS-PEP(bound/unbound)与Local_62(unbound/unbound)。在每个诱饵数据集中,GraphPep的表现与相应对接算法自带的评分函数以及InterPepScore方法进行了比较。
2.2.1 结合态基准测试(LEADS-PEP)
GraphPep首先在LEADS-PEP测试集上进行了评估,该数据集包含53个蛋白–肽复合物,对接诱饵结构由FlexPepDock、HADDOCK和HPEPDOCK生成(见补充表1–3)。如图2所示,GraphPep在不同数量的Top预测中,均显著优于三种对接算法自带的评分函数以及基于图深度学习的InterPepScore方法。
对于FlexPepDock生成的诱饵,GraphPep在Top 1、Top 3与Top 5预测中分别获得平均DockQ分数0.479、0.515与0.528,而FlexPepDock的对应分数为0.373、0.469与0.502,InterPepScore为0.405、0.464与0.499(图2a)。值得注意的是,GraphPep在Top 1预测中的表现甚至超过了FlexPepDock与InterPepScore在Top 3预测下的成绩。
在HADDOCK诱饵中,GraphPep同样取得最佳结果,Top 1、Top 3与Top 5的平均DockQ分数分别为0.549、0.613与0.637,而HADDOCK分别为0.438、0.470与0.568,InterPepScore分别为0.535、0.599与0.630(图2b)。
对于包含结合构象最全面且最精确的HPEPDOCK诱饵,GraphPep再次表现出最高的DockQ分数,Top 1、Top 3与Top 5分别达到0.814、0.854与0.860,而HPEPDOCK分别为0.706、0.794与0.816,InterPepScore分别为0.747、0.805与0.825(图2c)。
此外,可以看到InterPepScore在不同诱饵集间表现差异较大,在HADDOCK诱饵上的提升更明显,而GraphPep在三类诱饵集中均取得显著性能提升(图2a–c),显示出其对不同对接程序的良好稳健性。
进一步的个例分析表明,GraphPep在多数复合物中均获得相当或更优的预测结果。在Top 1预测中,GraphPep较FlexPepDock取得更高DockQ分数的案例有33个,较InterPepScore则有29个(图2d与补充表1)。在HADDOCK诱饵中,GraphPep在32个案例中优于HADDOCK,在27个案例中优于InterPepScore(图2e与补充表2)。在HPEPDOCK诱饵中,GraphPep在29个案例中优于HPEPDOCK,同时在29个案例中也超过InterPepScore(图2f与补充表3)。
2.2.2 非结合态基准测试(Local_62)
为进一步评估GraphPep在非结合态受体结构上的表现,研究在包含62个蛋白–肽复合物的Local_62基准数据集上进行了测试(见补充表4与5),其诱饵结构分别由HADDOCK与HPEPDOCK生成。该基准旨在评估受体柔性对GraphPep及其他方法性能的影响,因为缺乏诱导契合(induced-fit)构象的受体结构对模型构成了较大挑战。
在HADDOCK生成的诱饵中,GraphPep在各项评估指标上均优于HADDOCK与InterPepScore(图3a)。在Top 1预测下,GraphPep在中等精度标准下的成功率为32.2%,而HADDOCK与InterPepScore分别为21.0%与16.1%。在高精度模型方面,GraphPep正确预测了4.8%的样本,而InterPepScore仅为1.6%,HADDOCK则未能预测出任何高精度模型。当考虑Top 3与Top 5预测时,GraphPep的性能依然保持领先(图3a)。
图3b展示了不同方法的Top 1 DockQ分数随受体柔性(apo–holo Cα RMSD)的变化关系。从图中可以看出,GraphPep在各个RMSD范围内均能提升预测性能,并且对于apo–holo RMSD≥3 Å的复合物仍能生成中等甚至高质量的预测结果(见补充表4)。这些结果表明GraphPep对受体柔性具有较强的稳健性。
在HPEPDOCK生成的诱饵中,GraphPep同样在不同Top预测数量下优于HPEPDOCK与InterPepScore(图3c)。需要注意的是,HPEPDOCK为刚体对接程序,将蛋白视为固定结构,因此对于受体构象变化较大的复合物,其预测结合模式的难度较高。即便如此,GraphPep在Top 1预测下仍取得了显著提升——中等精度阈值的成功率为61.3%,高精度阈值为19.4%,远超HPEPDOCK的41.9%与11.3%,以及InterPepScore的38.7%与9.7%。类似的性能提升也出现在Top 2与Top 3预测中。
此外,GraphPep在不同程度受体柔性下的Top 1 DockQ分数均优于其他方法,甚至在apo–holo Cα RMSD超过6 Å的复合物中也能实现显著改进(图3d与补充表5),再次证明了GraphPep在应对受体柔性挑战时的稳健性与泛化能力。
2.3 在由ADCP与AFM构建的诱饵数据集上的性能
进一步地,研究在AutoDock CrankPep(ADCP)测试集上评估了GraphPep的性能,该测试集包含99个蛋白–肽复合物(见补充表6)。由于ADCP99原始诱饵集未公开,研究者依据其原始论文所述流程自行生成并在本地进行分析。图4展示了GraphPep及其他方法在由ADCP与AlphaFold-Multimer(AFM)生成的ADCP99基准诱饵集上的表现。对于组合诱饵集,采用了与文献中相同的共识评分策略:当肽序列的多序列比对(MSA)具有非零有效序列数(即Neff-pep > 0)时,选取AFM预测结果;否则根据测试评分函数选择Top 1结构。
从图4可以看出,GraphPep在全部三种诱饵集中均表现优于其他方法。具体而言,在ADCP诱饵集上(图4a),GraphPep在中等精度与高精度标准下的成功率分别为56.6%与31.3%,显著高于ADCP的47.5%与23.2%,以及InterPepScore的51.5%与25.3%。在AFM诱饵集中(图4b),各评分函数间的性能差异较小,可能源于结构多样性有限,但GraphPep在中、高精度标准下仍保持最佳表现。对于组合诱饵集(图4c),所有方法的成功率均有所提升,说明共识策略具有显著效果。其中,GraphPep在该策略下表现最优,成功率达到74.7%,超过InterPepScore(68.7%)与原论文中采用的共识能量方法(71.7%)。在高精度预测方面,GraphPep优势更加突出,成功率为58.6%,而InterPepScore与共识能量方法分别为48.5%与51.5%。
图4d–f展示了GraphPep与其他方法在不同诱饵集上的Top 1 DockQ分数比较。研究重点关注了37个肽序列无有效MSA(即Neff-pep = 0)的复合物,因为在这些情况下评分函数直接决定最终排名。结果显示,GraphPep在大多数样本中均取得相当或更高的DockQ分数(见补充表6)。在ADCP诱饵集中,GraphPep在14个案例中优于ADCP,在18个案例中优于InterPepScore(图4d);在AFM诱饵集中,GraphPep在15个案例中优于AFM,在20个案例中优于InterPepScore(图4e);而在组合诱饵集中,GraphPep在25个案例中取得更高的DockQ分数,进一步验证了其在不同诱饵生成策略下的稳健性与泛化能力。
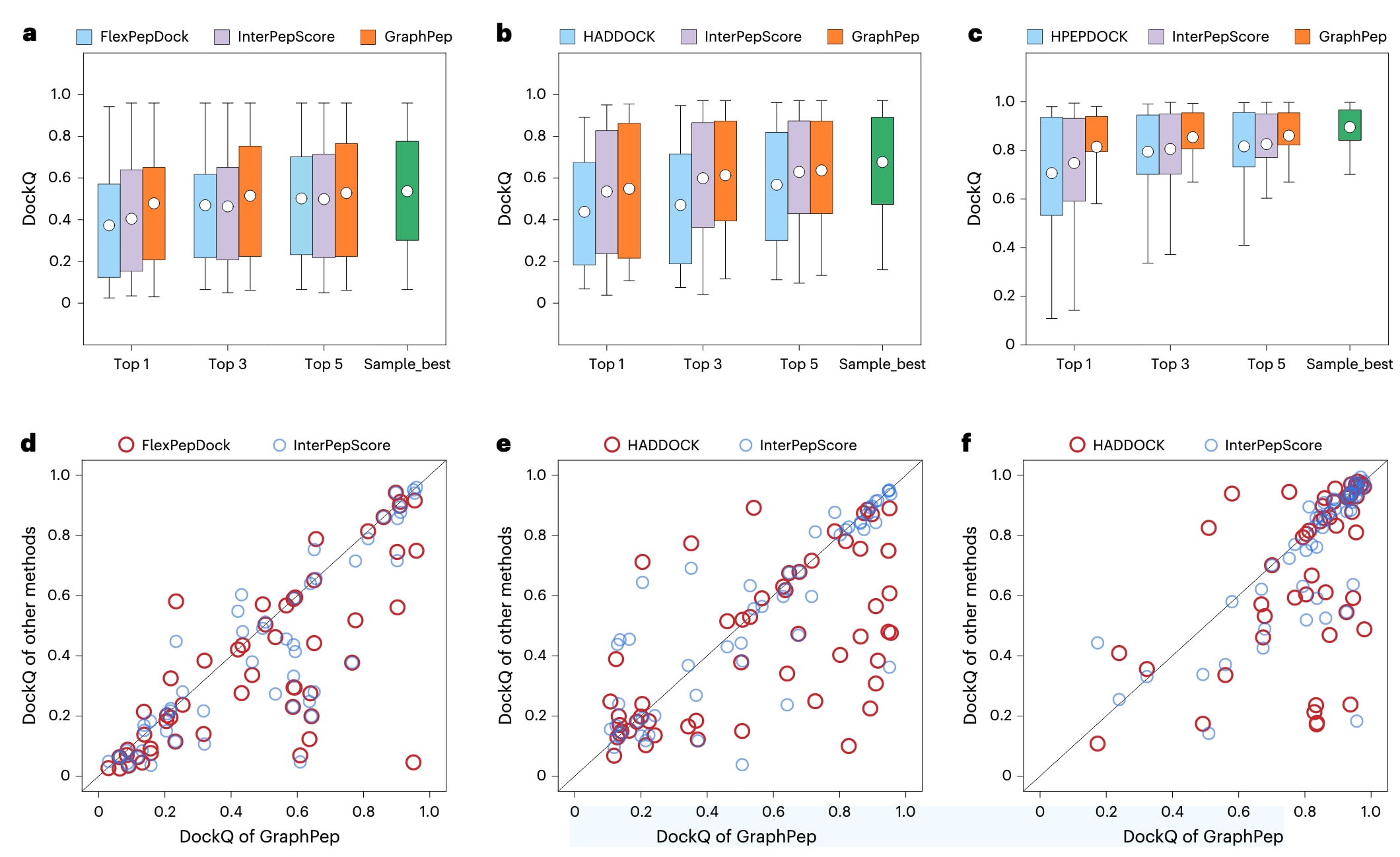
图2 | GraphPep与其他方法在LEADS-PEP结合态测试集上的比较。 a–c,不同方法在由FlexPepDock(a)、HADDOCK(b)与HPEPDOCK(c)生成的诱饵数据集上的Top 1、Top 3与Top 5预测结果(n = 53)的DockQ箱线图。图中圆点表示平均值,箱体边缘表示四分位区间,须线表示1.5倍四分位距。作为参考,同时展示了采样效果最佳诱饵的结果。d–f,GraphPep与其他方法在由FlexPepDock(d)、HADDOCK(e)和HPEPDOCK(f)生成的诱饵数据集上的Top 1 DockQ分数散点图,用以直观比较不同模型的预测精度。
2.4 在由ADCP与AFM构建的诱饵数据集上的性能
进一步地,研究在AutoDock CrankPep(ADCP)测试集上评估了GraphPep的性能,该测试集包含99个蛋白–肽复合物(见补充表6)。由于ADCP99原始诱饵集未公开,研究者依据其原始论文所述流程自行生成并在本地进行分析。图4展示了GraphPep及其他方法在由ADCP与AlphaFold-Multimer(AFM)生成的ADCP99基准诱饵集上的表现。对于组合诱饵集,采用了与文献中相同的共识评分策略:当肽序列的多序列比对(MSA)具有非零有效序列数(即Neff-pep > 0)时,选取AFM预测结果;否则根据测试评分函数选择Top 1结构。
从图4可以看出,GraphPep在全部三种诱饵集中均表现优于其他方法。具体而言,在ADCP诱饵集上(图4a),GraphPep在中等精度与高精度标准下的成功率分别为56.6%与31.3%,显著高于ADCP的47.5%与23.2%,以及InterPepScore的51.5%与25.3%。在AFM诱饵集中(图4b),各评分函数间的性能差异较小,可能源于结构多样性有限,但GraphPep在中、高精度标准下仍保持最佳表现。对于组合诱饵集(图4c),所有方法的成功率均有所提升,说明共识策略具有显著效果。其中,GraphPep在该策略下表现最优,成功率达到74.7%,超过InterPepScore(68.7%)与原论文中采用的共识能量方法(71.7%)。在高精度预测方面,GraphPep优势更加突出,成功率为58.6%,而InterPepScore与共识能量方法分别为48.5%与51.5%。
图4d–f展示了GraphPep与其他方法在不同诱饵集上的Top 1 DockQ分数比较。研究重点关注了37个肽序列无有效MSA(即Neff-pep = 0)的复合物,因为在这些情况下评分函数直接决定最终排名。结果显示,GraphPep在大多数样本中均取得相当或更高的DockQ分数(见补充表6)。在ADCP诱饵集中,GraphPep在14个案例中优于ADCP,在18个案例中优于InterPepScore(图4d);在AFM诱饵集中,GraphPep在15个案例中优于AFM,在20个案例中优于InterPepScore(图4e);而在组合诱饵集中,GraphPep在25个案例中取得更高的DockQ分数,进一步验证了其在不同诱饵生成策略下的稳健性与泛化能力。
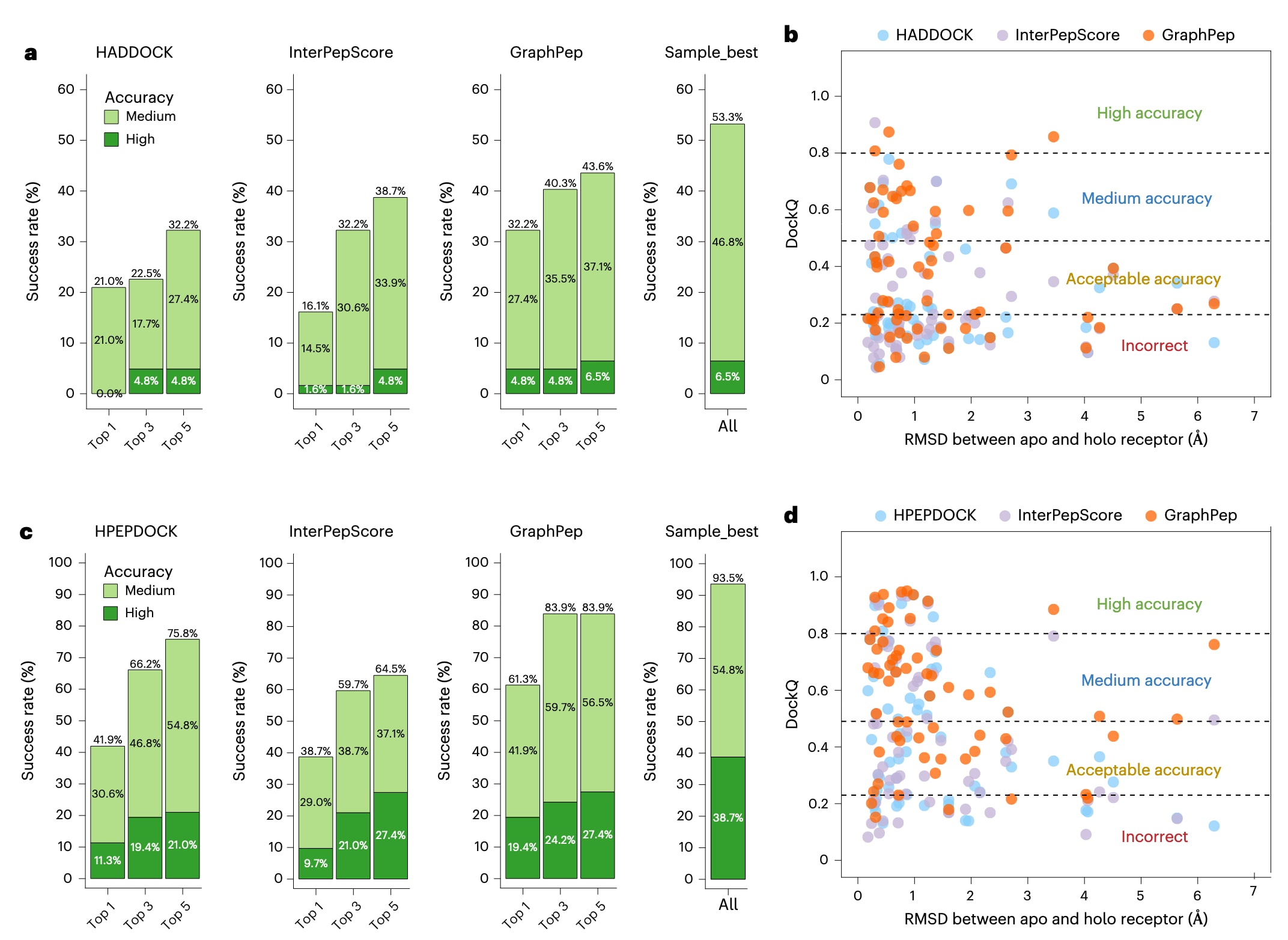
图3 | GraphPep与其他方法在Local_62非结合态测试集上的比较。 a、c,不同方法在由HADDOCK(a)与HPEPDOCK(c)生成的诱饵数据集上的Top 1、Top 3与Top 5预测成功率。作为参考,同时展示了采样效果最佳诱饵的结果。b、d,不同方法在由HADDOCK(b)与HPEPDOCK(d)生成的诱饵数据集上的Top 1 DockQ分数与受体apo与holo结构之间Cα RMSD的散点图。虚线表示DockQ分类区域的阈值,用以区分高精度、中等精度及低精度预测范围。
2.5 在由ADCP与AFM构建的诱饵数据集上的性能
进一步地,研究在AutoDock CrankPep(ADCP)测试集上评估了GraphPep的性能,该测试集包含99个蛋白–肽复合物(见补充表6)。由于ADCP99原始诱饵集未公开,研究者依据其原始论文所述流程自行生成并在本地进行分析。图4展示了GraphPep及其他方法在由ADCP与AlphaFold-Multimer(AFM)生成的ADCP99基准诱饵集上的表现。对于组合诱饵集,采用了与文献中相同的共识评分策略:当肽序列的多序列比对(MSA)具有非零有效序列数(即Neff-pep > 0)时,选取AFM预测结果;否则根据测试评分函数选择Top 1结构。
从图4可以看出,GraphPep在全部三种诱饵集中均表现优于其他方法。具体而言,在ADCP诱饵集上(图4a),GraphPep在中等精度与高精度标准下的成功率分别为56.6%与31.3%,显著高于ADCP的47.5%与23.2%,以及InterPepScore的51.5%与25.3%。在AFM诱饵集中(图4b),各评分函数间的性能差异较小,可能源于结构多样性有限,但GraphPep在中、高精度标准下仍保持最佳表现。对于组合诱饵集(图4c),所有方法的成功率均有所提升,说明共识策略具有显著效果。其中,GraphPep在该策略下表现最优,成功率达到74.7%,超过InterPepScore(68.7%)与原论文中采用的共识能量方法(71.7%)。在高精度预测方面,GraphPep优势更加突出,成功率为58.6%,而InterPepScore与共识能量方法分别为48.5%与51.5%。
图4d–f展示了GraphPep与其他方法在不同诱饵集上的Top 1 DockQ分数比较。研究重点关注了37个肽序列无有效MSA(即Neff-pep = 0)的复合物,因为在这些情况下评分函数直接决定最终排名。结果显示,GraphPep在大多数样本中均取得相当或更高的DockQ分数(见补充表6)。在ADCP诱饵集中,GraphPep在14个案例中优于ADCP,在18个案例中优于InterPepScore(图4d);在AFM诱饵集中,GraphPep在15个案例中优于AFM,在20个案例中优于InterPepScore(图4e);而在组合诱饵集中,GraphPep在25个案例中取得更高的DockQ分数,进一步验证了其在不同诱饵生成策略下的稳健性与泛化能力。
2.6 在由AlphaFold3生成的诱饵数据集上的性能
GraphPep还在nr_epitope_minus基准数据集上进行了评估,该数据集包含121个抗体–肽复合物。研究者利用AlphaFold3并采用强制采样策略(每个复合物使用20个随机种子生成100个模型)生成诱饵结构(见补充表7)。生成模型最初依据肽链的界面预测模板建模分数(ipTM score)进行排序,该分数反映了模型在链间位置预测上的置信度。为了对比性能,研究还在相同诱饵集上测试了InterPepScore。
如图5a所示,GraphPep在所有被评估方法中表现最佳。在Top 1预测下,GraphPep在中等精度与高精度标准下的成功率分别为43.7%与15.6%;相比之下,InterPepScore分别为37.5%与14.1%,而AlphaFold3为42.2%与12.5%。值得注意的是,图中显示评分方法与“最佳采样模型”之间仍存在显著性能差距,这表明在抗体–肽复合物场景下,结合模式评分仍是一项挑战。
图5b展示了不同方法的DockQ分数箱线图,可以看到GraphPep的平均DockQ分数最高,为0.469,而InterPepScore与AlphaFold3分别为0.428与0.441。图5c进一步展示了不同肽长度范围内的Top 1预测表现。从图中可以看出,随着肽链长度增加,三种方法的成功率均逐渐下降,反映出长肽结合模式预测的复杂性与困难度更高。
此外,GraphPep在短肽与中等长度肽预测中均优于InterPepScore,并在短肽情况下超过AlphaFold3。对于长度超过15个氨基酸的长肽,由于构象柔性与结合复杂度的增加,所有方法在中等精度下的成功率均为33.3%。然而,AlphaFold3未能在这些复合物中预测出任何高精度结构,而GraphPep与InterPepScore均实现了11.1%的高精度成功率,进一步展现了GraphPep在处理复杂抗体–肽结合体系中的稳健性与泛化能力。
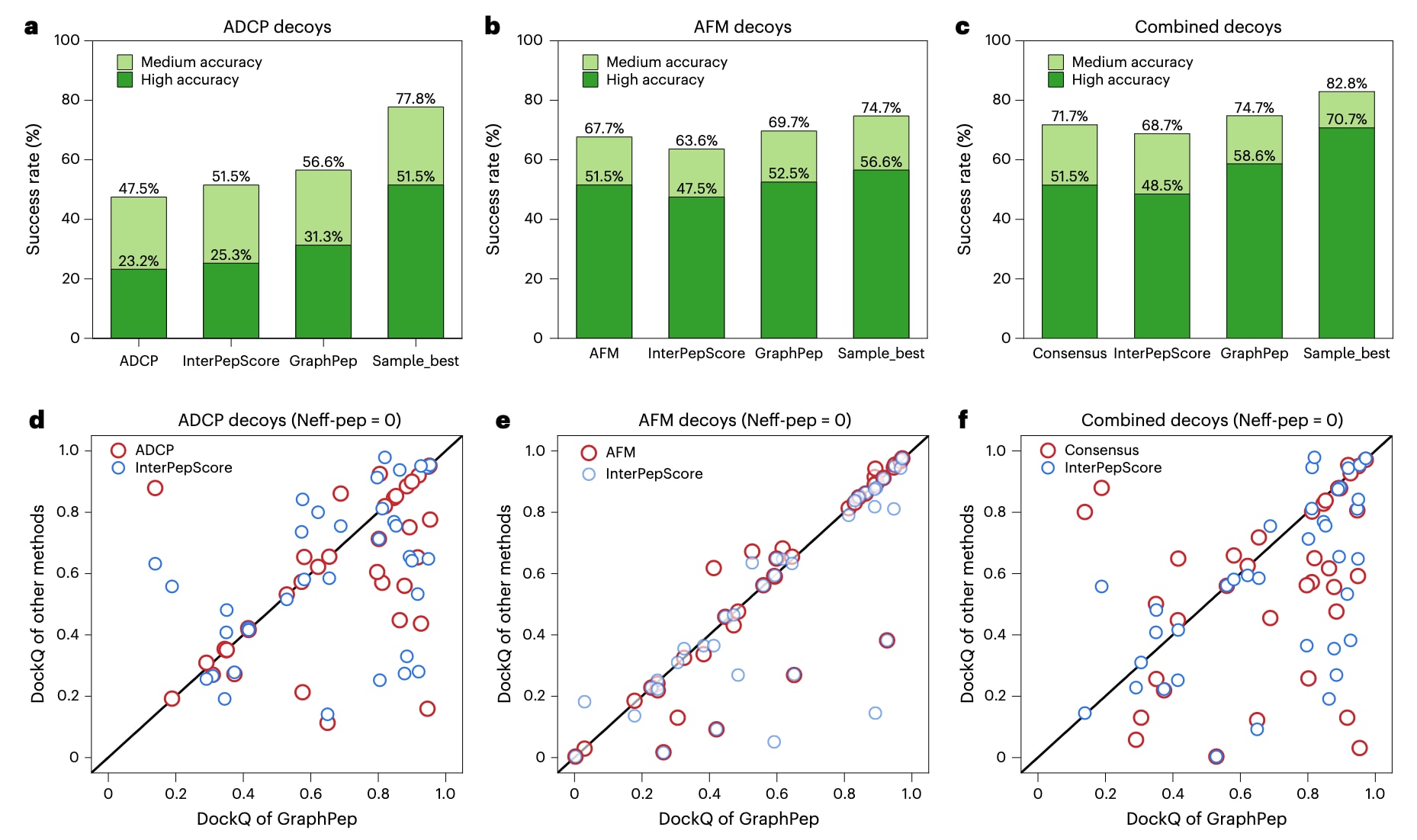
图4 | GraphPep与其他方法在ADCP99测试集上的比较。 a–c,不同方法在由ADCP(a)、AFM(b)及其组合诱饵集(c)生成的结构上的成功率比较。对于c中的组合诱饵排名,当肽的有效序列数Neff > 0时,采用AFM预测结果;否则根据各评分函数在组合诱饵集中的Top 1模型进行选择。作为参考,同时展示了采样效果最佳诱饵的结果。d–f,GraphPep与其他方法在由ADCP(d)、AFM(e)及其组合诱饵集(f)生成的结构上的Top 1 DockQ分数散点图。为便于展示,图中仅显示了肽的有效序列数Neff-pep = 0的复合物结果。
2.7 预测结合模式示例
图6展示了由HPEPDOCK与GraphPep预测的三组典型结合模式,并与实验测定的天然肽结构进行了比较。从图中可以看出,HPEPDOCK预测的结合模式与实验结构存在显著差异,且GraphPep对这些结构给出的置信度得分较低。相反,GraphPep预测的结合构象与天然结构更为接近,且与天然肽重叠良好的残基对应更高的置信度评分。
在LEADS-PEP数据集中,GraphPep在4DS1复合物上取得了高精度预测,DockQ分数达到0.875,而HPEPDOCK仅为0.469。对于Local_62数据集中的2C3I,HPEPDOCK预测的肽方向与天然结构相反,而GraphPep则正确预测了结合方向,DockQ分数为0.771。至于Local_62中的1T7R,实验结构中的肽为α螺旋,但HPEPDOCK预测为无规卷曲构象,导致DockQ分数仅为0.247;相比之下,GraphPep正确预测其为α螺旋,DockQ分数为0.745,达到了中等精度标准。
这三个案例突显了HPEPDOCK在结合模式预测中的局限性,包括结合位姿偏差、肽链取向错误及二级结构预测不准等问题。而GraphPep能够准确恢复与天然结构一致的结合模式,正确捕捉结合方向与二级结构特征。值得注意的是,GraphPep还能为每对相互作用残基或接触提供置信度评分,可用于指导预测结合模式的优化。例如,在4DS1、2C3I与1T7R中,GraphPep预测结果的结合尾部区域均呈红色(图6),提示这些残基区域仍需进行局部精修。
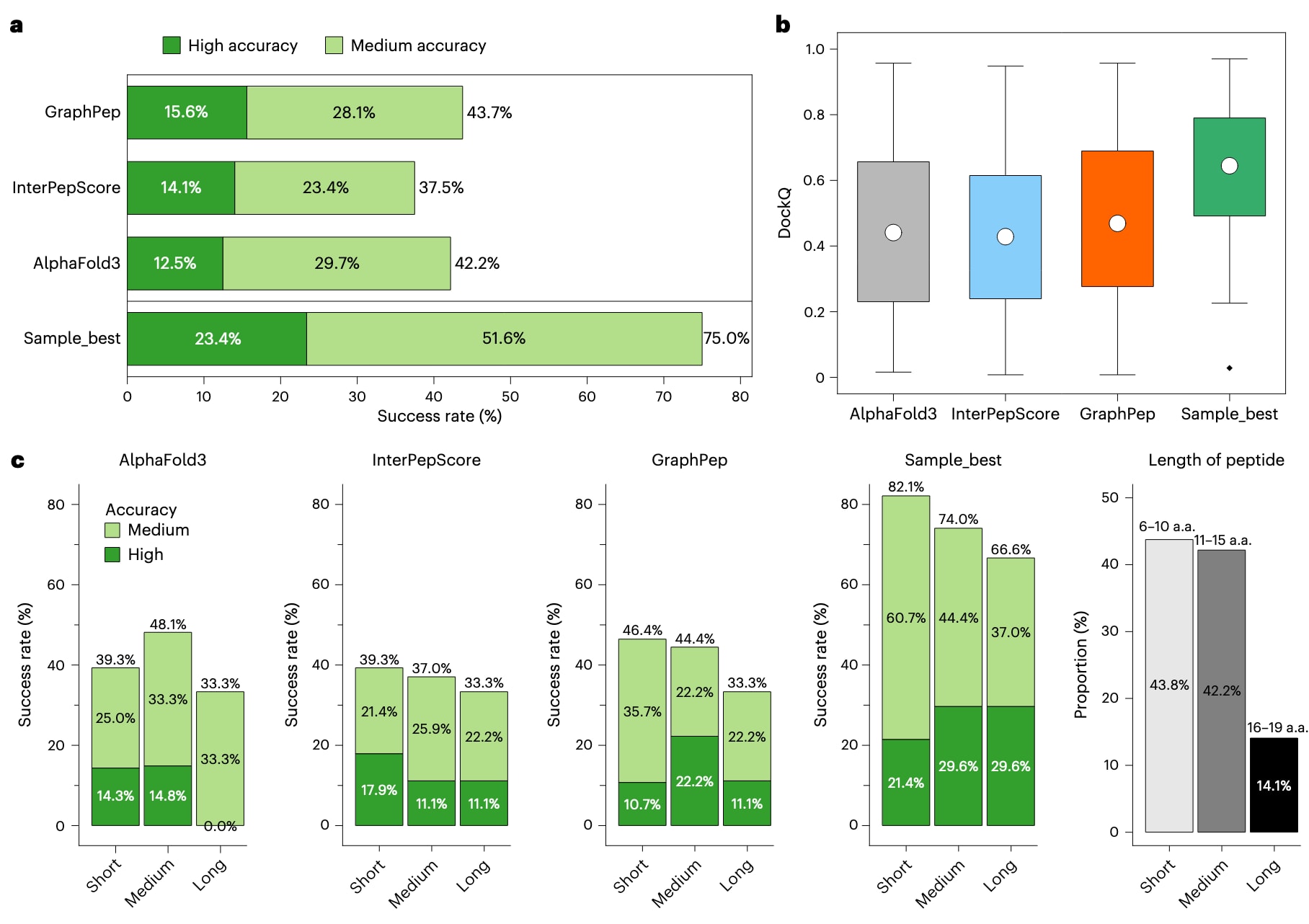
图5 | GraphPep与其他方法在nr_epitope_minus测试集上的比较。 该测试集的诱饵由AlphaFold3通过增强采样生成。a,不同方法的总体成功率比较。b,Top 1预测的DockQ分数箱线图(n = 64),圆点表示平均值,箱体边缘表示四分位区间,须线表示1.5倍四分位距。c,不同肽长度范围内的成功率比较。右侧展示了短肽(6–10个氨基酸)、中等长度肽(11–15个氨基酸)与长肽(16–19个氨基酸)的比例。成功率计算仅考虑每个复合物的Top 1预测结果。作为参考,同时展示了AlphaFold3生成的最佳采样诱饵结果。
2.8 GraphPep评分与结合构象精度的关系
理想的评分函数不仅应能识别接近天然状态的结合构象,还应能引导结合模式向天然结构方向逐步优化。换言之,在结合能量景观中应呈现出典型的“漏斗状”(funnel-like)特征,即越接近天然构象的结合模式应获得越优的评分。为此,研究选取了LEADS-PEP与Local_62数据集中由HPEPDOCK生成的诱饵集合进行评估,因为这两组数据集中每个复合物包含更多诱饵模型,统计结果更具可靠性。
研究分别计算了不同方法在两组诱饵集上评分与配体RMSD(LRMSD)及DockQ之间的皮尔森相关系数(见补充表8与9)。结果显示,GraphPep在两组数据集中均获得了整体更高的相关性(见补充图1)。
在评分–LRMSD关系方面,GraphPep在LEADS-PEP诱饵集中的平均相关系数为0.541,明显高于HPEPDOCK的0.134与InterPepScore的0.305(补充表8);在Local_62诱饵集中,GraphPep的相关系数为0.446,而HPEPDOCK与InterPepScore分别为0.047与0.098(补充表9)。
在评分–DockQ关系方面,GraphPep同样表现出更优的负相关趋势。在LEADS-PEP诱饵集中,其平均相关系数为−0.599,而HPEPDOCK与InterPepScore分别为−0.172与−0.348(补充表8);在Local_62诱饵集中,GraphPep的相关系数为−0.512,而HPEPDOCK与InterPepScore分别为−0.063与−0.118(补充表9)。
这些结果表明,GraphPep不仅能有效区分接近天然构象的结合模式,还具备指导初始采样结果进一步结构精修的潜力,为结合构象优化提供可靠的能量指引。
2.9 消融实验
为探究不同模块对GraphPep性能的影响,研究进行了系统的消融实验,考察了图层级结构、蛋白语言模型嵌入及分子内相互作用等因素(见补充表10与11)。补充图2展示了在由HPEPDOCK生成的LEADS-PEP与Local_62诱饵集上,不同消融模型的Top 1预测结果对应的DockQ区间图。
从结果可以看出,GraphPep的多尺度图学习机制——即结合原子层与残基层的联合表示——相比单一层级模型(ResGraph或AtomGraph)表现更优,说明多层级特征融合对捕捉关键相互作用具有重要作用。此外,引入ESM-2蛋白语言模型嵌入显著提升了模型性能,尤其在Local_62数据集上效果更为明显。
有趣的是,将分子内相互作用(包括分子内接触w/Intra_contact或分子内共价键w/Intra_covalent)显式纳入图结构并未提升性能(见补充图2),这可能是由于分子内信号过强,掩盖了关键的分子间界面相互作用。
此外,研究还通过将GraphPep中的ESM-2替换为最新的ESM-3模型来评估语言模型的影响。然而,此举导致性能下降(见补充图3)。原因可能在于ESM-3在训练中引入了结构信息,使其对构象变化更为敏感;而在蛋白–肽结合过程中,界面常伴随显著构象变化,因此ESM-3在实际相互作用预测中稳健性较低。类似地,当将当前的图卷积网络(GCN)替换为**SE(3)-等变网络(E3NN)**时,也观察到性能下降(见补充图4),进一步说明在蛋白–肽相互作用预测任务中,GraphPep当前架构在精度与稳健性之间达到了更优平衡。
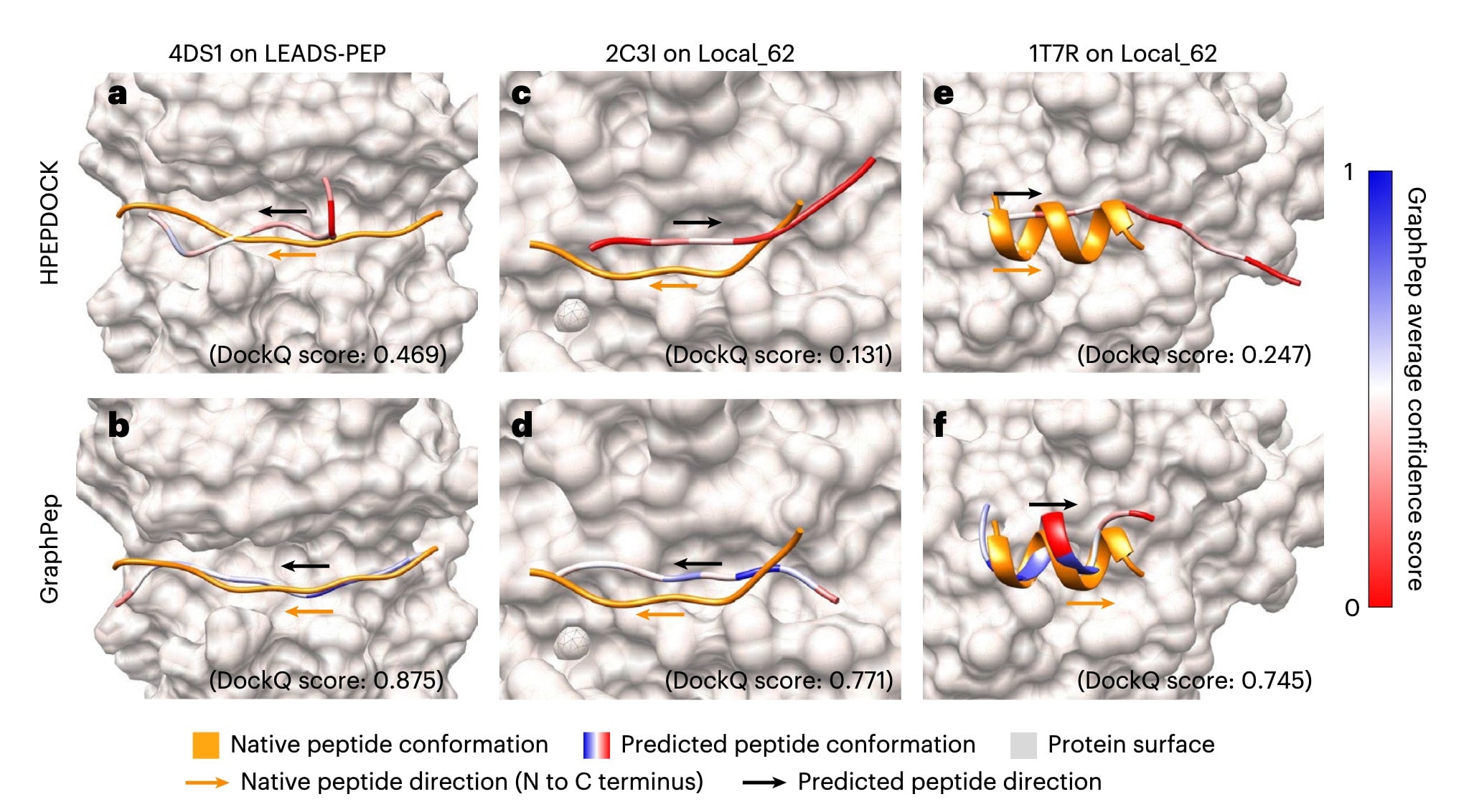
图6 | HPEPDOCK与GraphPep预测的Top结合模式示例。 a–f,第一行分别展示了HPEPDOCK对4DS1(a)、2C3I(c)与1T7R(e)的Top结合模式预测结果;第二行则展示了GraphPep对4DS1(b)、2C3I(d)与1T7R(f)的对应预测结果。
3 讨论
该研究提出了GraphPep,一种基于相互作用衍生图的图神经网络(GNN)评分模型,用于预测蛋白–肽相互作用。由于PDB数据库中可用的蛋白–肽复合物结构数量有限,训练高精度评分函数一直是该领域的挑战。为克服训练数据稀缺问题,GraphPep融合了三项关键策略。首先,GraphPep在图表示中以蛋白–肽相互作用而非传统的原子或残基作为节点,并在两节点共享同一原子或残基时建立边连接,从而更直接地描述界面作用模式。其次,GraphPep的训练目标是复现真实的蛋白–肽残基–残基接触模式,而非仅依赖单一的肽RMSD指标,从而显著降低了训练所需诱饵数量。第三,GraphPep采用多尺度图学习策略,通过将原子层特征汇聚到残基层节点,实现精度与稳健性的平衡。
在多个独立测试集上的广泛评估结果表明,GraphPep在蛋白–肽结合模式预测中表现显著优于现有主流方法,包括FlexPepDock、HADDOCK、HPEPDOCK、ADCP、InterPepScore、AFM及AlphaFold3。此外,GraphPep的评分与LRMSD及DockQ分数呈高度相关,在结合能量景观中呈现出理想的“能量漏斗”特征,进一步证明了其评分机制的物理合理性与预测准确性。
由于对接算法常会生成存在空间排斥的模型,GraphPep引入了PoseBusters工具用于结构合理性检测。结果显示,尽管在仅保留空间合理模型后GraphPep性能略有下降,但仍显著优于其他评分方法(见补充图5)。同时,GraphPep能够识别并降低带有空间冲突的诱饵评分。例如,在某HPEPDOCK诱饵中,原始模型DockQ分数为0.843,但存在原子冲突,GraphPep最初将其排在第44位;经轻微结构精修去除冲突后,该模型提升至第7位(见补充图6),说明GraphPep倾向于优先选择无明显空间冲突的构象。
值得注意的是,GraphPep在计算总评分时会综合所有接触置信度,因此若诱饵仅包含少量局部冲突而整体界面合理,模型仍可能获得较高评分。这一设计在一定程度上增强了GraphPep对受体柔性的容忍性,使其能够优先选择总体结合界面优良的模型。
此外,肽的二级结构类型同样会影响蛋白–肽复合物预测的准确性,尤其是采用螺旋构象的肽常被认为更具挑战性。为评估此影响,研究将肽分为螺旋型与非螺旋型两类,并在三个基准数据集上进行了性能分析(见补充图7)。结果显示,GraphPep在两类肽结构上均表现出优于或可比的预测精度,无论在传统对接诱饵集还是深度学习生成诱饵集中均保持稳健,进一步凸显了其在多样化蛋白–肽相互作用预测中的普适性与可靠性。