NMI 2025 | ITsFlexible: 预测抗体与T细胞受体互补决定区的构象柔性

获取详情及资源:
0 摘要
许多蛋白质具有高度的柔性,其形状适应能力往往是其功能特性的基础。例如,抗体互补决定区(CDR)环的柔性会影响结合亲和力与特异性,因此是理解和设计抗原相互作用的重要因素。借助AlphaFold等方法,可以在计算上高精度地预测单一、静态的蛋白质结构,但可靠地预测结构柔性仍未实现。限制此类预测的主要因素之一是缺乏适合的训练数据。该研究聚焦于功能上重要的抗体与T细胞受体CDR3环的结构柔性预测。为此,研究者通过从蛋白质数据银行中提取所有CDR3及类似CDR3的环状结构模体,构建了名为ALL-conformations的数据集。该数据集包含约120万个环结构,覆盖超过10万个独特序列,囊括了这些模体的所有实验观测构象。基于此,研究者开发了ITsFlexible——一种采用图神经网络架构的深度学习工具。模型通过抗体结构输入,将CDR环二分类为“刚性”或“柔性”。在晶体结构数据集上,ITsFlexible优于所有其他方法,并在分子动力学模拟中展现出良好的泛化性能。研究者还利用ITsFlexible预测了三个尚无解析结构的CDRH3环的柔性,并通过冷冻电子显微镜实验验证了其构象。
1 引言
许多蛋白质是具有柔性的分子,能够呈现多种稳定结构(称为构象),而这些构象之间的转变往往是其功能的关键。抗体与T细胞受体(TCR)主要通过六个环状结构域与靶标结合,这些环被称为互补决定区(CDR)。CDR的结构柔性与多种关键功能特性密切相关。对于某些抗体而言,构象变化是识别抗原所必需的;而抗原受体能够采用多种构象的能力与多特异性相关,因为不同的结构状态允许其识别更广泛的抗原。此外,柔性还会影响结合亲和力,因为它直接决定了抗原结合时的熵代价,而刚性化则被认为是自然界中提高亲和力的一种机制。特异性与亲和力是抗体与TCR治疗分子的两项核心性质。为了最大化靶标结合并减少非特异性相互作用,理想的治疗分子应具备高亲和力与高特异性,这意味着它们应倾向于刚性。然而,也有证据表明,构象柔性有助于更好地识别突变抗原,因此在设计广谱中和抗体时可能是理想特征。不论哪种情形,能够预测CDR柔性的方法都将有助于深入理解抗体功能,并为调控其治疗性质提供新的手段。
从序列出发预测蛋白单一静态结构已成为常规任务,AlphaFold等模型的成功便是例证,且近期出现的工具在蛋白复合物预测中也表现出潜力。然而,预测蛋白多种构象状态仍然极具挑战。限制进展的一个主要因素是缺乏合适的数据。目前,关于构象柔性的证据主要来自多种实验技术。核磁共振光谱与氢–氘交换质谱可用于测量蛋白在溶液中的动态变化,但通常无法提供原子分辨率层面的柔性信息。X射线晶体学是解析高分辨率构象状态的标准方法,可通过在不同条件下求解同一蛋白的多个晶体结构来捕捉不同状态。然而,能以晶体学数据评估柔性的蛋白数量远少于已解析的总体结构。已有研究利用晶体结构探索特定环类型的柔性,或针对完整蛋白进行个案分析,但尚缺乏对蛋白质数据库中相同序列代表的不同构象的系统挖掘。分子动力学(MD)模拟提供了一种计算生成构象集合的途径,但由于计算开销极大,即便是目前最大规模的标准化MD数据库,也不足以支持机器学习模型的训练。
尽管存在数据局限,仍有多种方法尝试预测蛋白的构象集合。近期的一些工作集中于修改AlphaFold2的推理过程,以提升输出结构的多样性。这些方法通常针对AlphaFold2的核心输入——多序列比对(MSA)——进行调整,通过减少MSA深度、随机采样或序列聚类等方式解耦不同构象状态的信号。除此之外,研究者还开发了一系列专门为构象预测任务训练的生成模型,这些模型通常基于蛋白质数据库及少量MD模拟数据进行训练。尽管这些方法在个案中表现出一定效果,但整体来看,目前尚不能可靠地预测蛋白的多构象状态。
过去,对抗体与TCR的CDR构象状态预测仅在少量个案中得到验证。该研究聚焦于功能上最为关键的抗体与TCR的CDR3环。为缓解数据稀缺的问题,研究者考虑了所有蛋白中具有相同二级结构模式的环状结构,即被两条反平行β链界定的环。通过对蛋白质数据库及抗体/TCR专用数据库的系统挖掘,构建了“抗体样环构象”(ALL-conformations)数据集。该数据集包含约120万个环状晶体结构,覆盖10万个独特序列,全面收录了在反平行β链间观测到的所有实验构象,包括抗体与TCR的CDR3环。在ALL-conformations数据集中,研究者分析了环的结构柔性,并根据构象变化能力为两万余个独特序列标注柔性状态。基于此,构建了免疫球蛋白与TCR柔性分类器(ITsFlexible),可判断CDR3环是刚性的(仅呈单一构象)还是柔性的(可在多状态间转变)。ITsFlexible在晶体结构集合中以高精度预测CDR3柔性,表现优于现有方法,并能有效泛化至来自分子动力学模拟的测试集。此外,研究者还利用ITsFlexible预测了三个尚无解析结构的CDRH3环的柔性,并通过冷冻电镜实验验证其构象,结果显示其中两项预测准确。ALL-conformations数据集已在Zenodo平台公开,ITsFlexible模型亦可在GitHub与Zenodo获取。
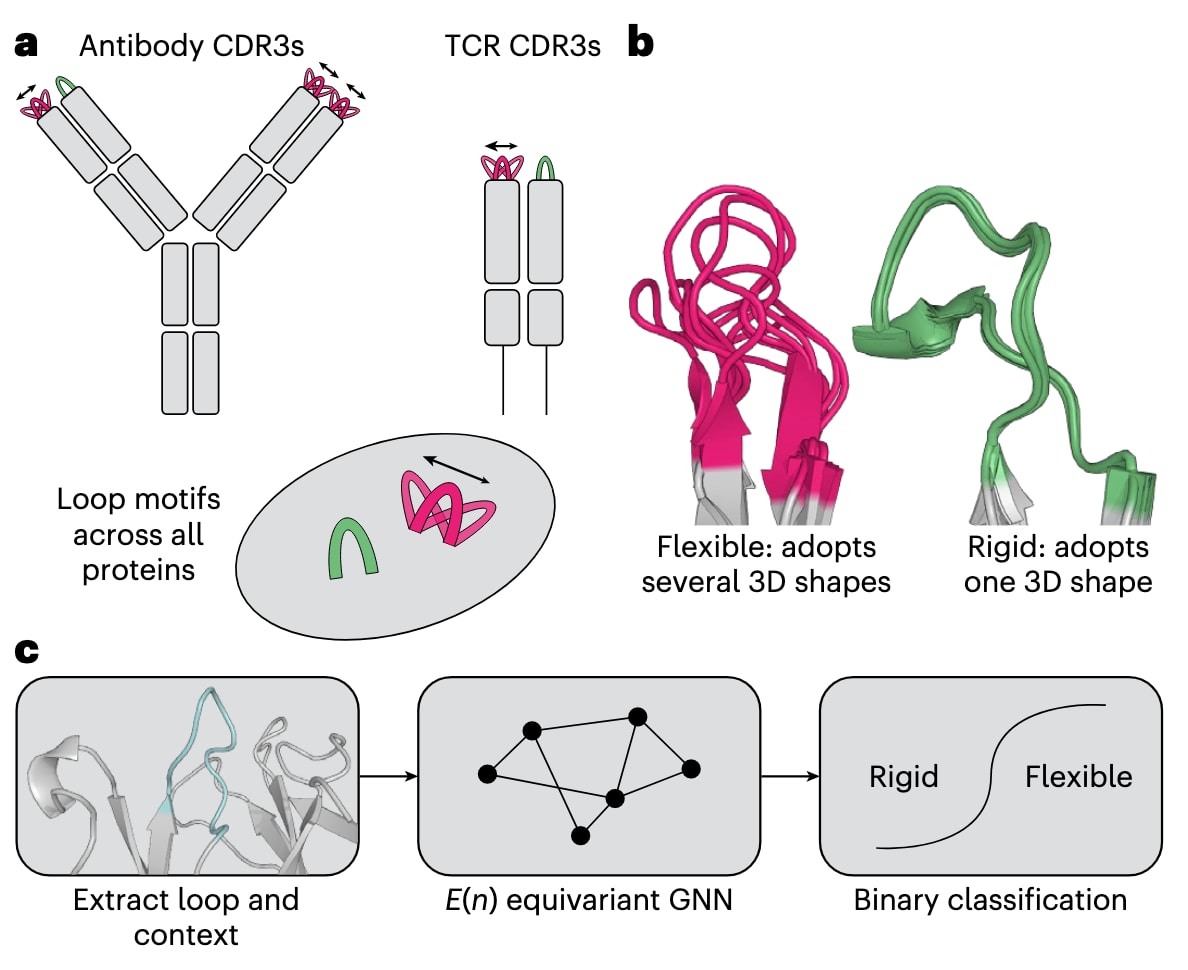
图1 | ALL-conformations与ITsFlexible的概览。 a,ALL-conformations是一个数据集,包含抗体CDR3、TCR CDR3以及所有蛋白中的CDR样环状结构的晶体结构。该数据集收录了此类环结构的全部实验观测构象。 b,环根据其在不同构象中的观测情况被标注为“柔性”或“刚性”:若同一序列被观测到多种构象,则标为柔性;若仅有单一构象证据,则标为刚性。构象依据结构相似性定义,并采用RMSD=1.25 Å作为区分不同状态的阈值。 c,展示ITsFlexible方法的流程图,该方法用于预测CDR环的构象柔性。模型从PDB文件中提取目标环的结构与序列(蓝绿色)及其结构环境(灰色),构建图表示,然后利用图神经网络(GNN)对环进行分类,判断其是构象柔性还是刚性。
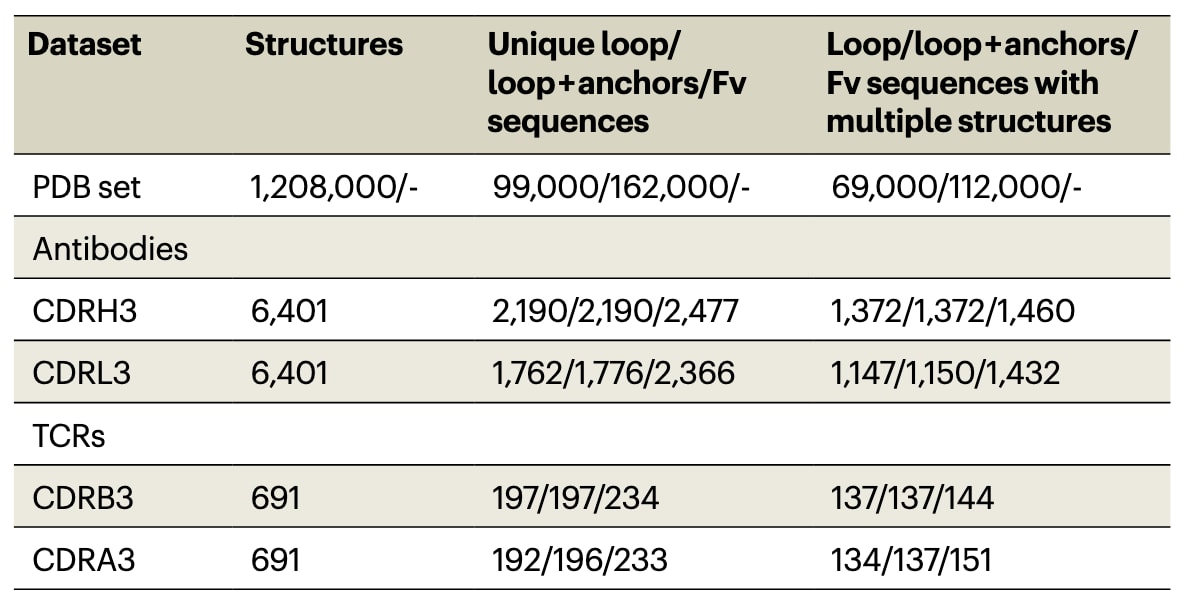
表1|ALL-conformations数据集中的结构与序列数量
2 结果
2.1 ALL-conformations数据集
ALL-conformations是一个用于刻画由两条反平行β链界定的环状结构构象柔性的数据集,包含五个子集:抗体的CDRH3与CDRL3、TCR的CDRB3与CDRA3,以及蛋白质数据库(PDB)中所有蛋白的环状结构模体(见补充图4)。该数据集收集了此类环结构的所有可用晶体结构,覆盖了实验上观测到的全部构象状态。通过从抗体结构数据库(SAbDab)与T细胞受体结构数据库提取CDR3结构,并从PDB中提取环状模体,最终获得超过120万个环实例,涵盖10万余条独特序列(见表1)。PDB中的环长度分布范围为1至87个氨基酸残基,但短环显著富集(见补充图5)。CDR3的长度分布则不同,主要集中在10至15个残基左右,且不包含少于4个氨基酸的环(见补充图6)。大量环序列在数据库中由多个晶体结构表示,说明存在多种实验构象。
所有环根据构象状态被分类为刚性、柔性或未知(详见方法部分)。在晶体结构中观测到多种构象的环被标记为柔性。构象定义为任意两成员的均方根偏差(RMSD)小于1.25 Å的簇结构,这一阈值此前已被证明能较好地区分抗体的功能性构象。因此,构象簇可视为功能上独立的结构状态。由于无法完全排除某些仅观测到单一构象的环仍存在未捕捉的其他状态,研究者仅在相同构象被重复观测超过5次时,才将其标记为刚性,以确保刚性类别富含确为单一构象的环。依据上述标准,共识别出约16,000个刚性环与4,000个柔性环(见补充表5)。关于RMSD阈值变化对分类比例的影响,详见补充图10与补充章节2.3。
2.2 预测类似CDR的蛋白环状结构的柔性
ALL-conformations数据集被用于训练ITsFlexible模型,用以预测蛋白环的构象柔性。ITsFlexible是一种图神经网络(见补充图1),能够根据环及其结构环境的序列与结构编码信息,将环二分类为可呈现多种构象的“柔性”或仅保持单一稳定状态的“刚性”(详见方法部分)。模型的训练与评估基于ALL-conformations中来自所有蛋白的环状模体集合。数据划分依据序列同一性进行,测试集中的环与训练及验证集中长度匹配的环序列同一性不超过80%。
分类器性能与随机分类、三种基线模型以及基于AlphaFold2预测的局部距离差异测试(pLDDT)的零样本柔性预测流程进行了比较(见图2)。随机分类的验证指标来自于对给定标签比例的随机分配结果。基线模型依据生物物理特征(如环长度与溶剂暴露度)预测柔性,这些特征在以往研究中已被证明与环的动力学相关。较长的环具有更多可旋转的键,而溶剂暴露度较高的环受空间位阻限制较小,更易发生构象变化。pLDDT是残基级置信度指标,可用于识别无序区域。由于测试集中包含同一环的多个结构,ITsFlexible对每个实例分别进行预测,并根据平均预测得分计算评估指标。
ITsFlexible能够有效区分环的柔性状态,其精确率–召回曲线下面积(PR AUC)为0.62,ROC曲线下面积(ROC AUC)为0.84,优于所有基线模型与零样本分类器。通过消融实验进一步分析模型输入特征的重要性。ITsFlexible-loop与默认模型相似,但仅使用环本身的序列与结构作为输入(见补充章节1.1);ITsFlexible-sequence则是基于卷积神经网络的模型,仅使用环序列编码训练(见补充章节1.1)。两者性能相近,均优于相关基线模型(仅环长作为对照,因为溶剂暴露需考虑结构环境),但预测能力均不及完整的ITsFlexible模型(见图2)。
这些结果揭示了决定蛋白环构象柔性的关键因素。已知较长且暴露于溶剂的环往往比短而埋藏的环更具柔性。该研究结果与这一趋势一致,同时显示环的氨基酸序列本身对柔性的预测性高于长度,说明序列特征对其多构象能力有重要影响。模型性能的大幅提升来自于引入环的结构环境信息,这与以往分子动力学研究结果一致,进一步强调了环与其蛋白环境之间的相互作用是决定其构象动力学的重要因素。
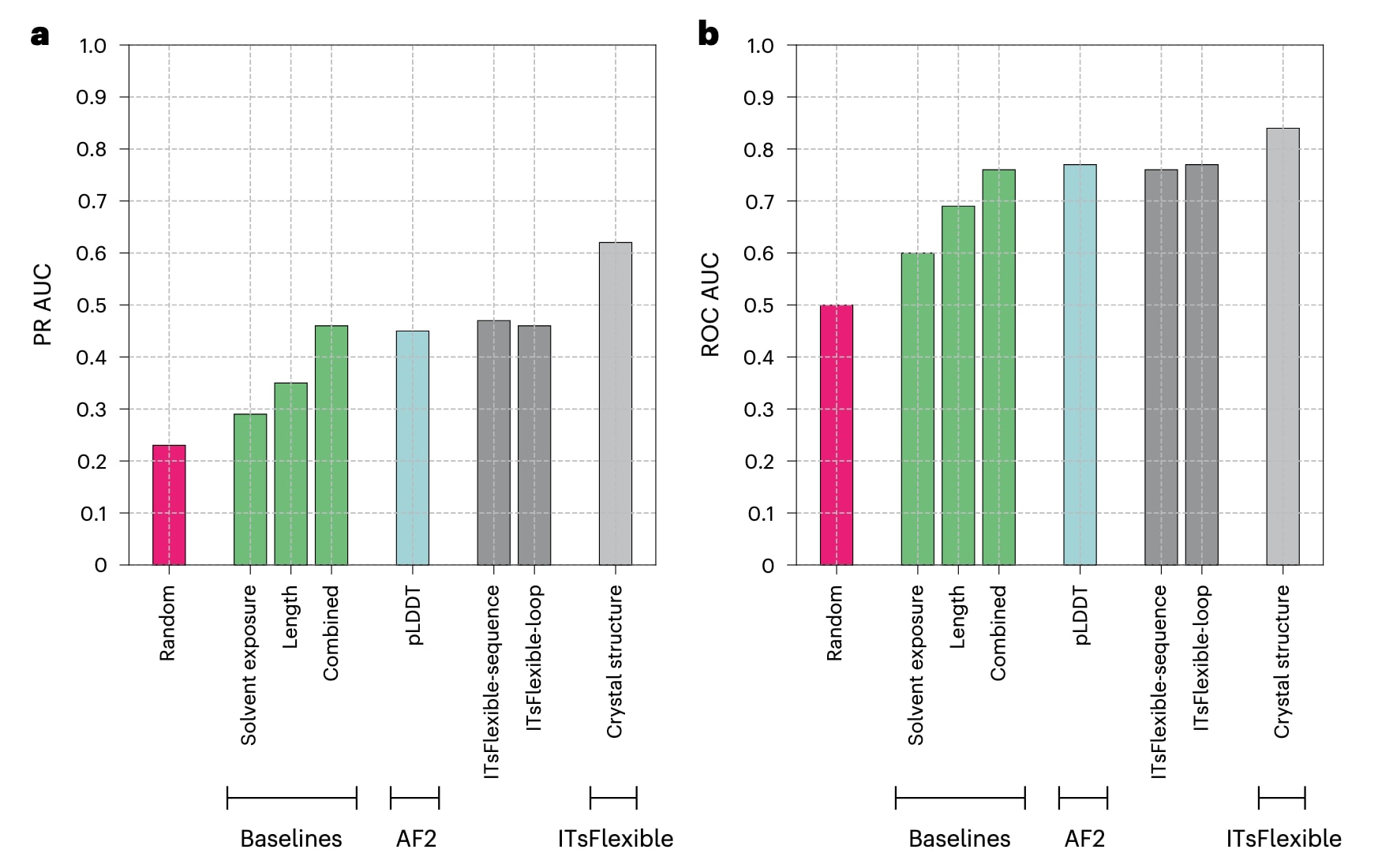
图2|ITsFlexible在PDB测试集上的性能评估。 a、b,基于PDB测试集(包含2,845个环状模体)的分类结果,分别以PR AUC(a)与ROC AUC(b)为评估指标。浅灰色柱表示ITsFlexible模型性能,并与随机分类(红色)、三种基于生物物理特征的基线模型(绿色)、基于AF2的pLDDT模型(蓝色)以及两种输入特征消融版本的ITsFlexible模型(深灰色)进行比较。具体的PR AUC与ROC AUC数值见补充表9与表10。
2.3 ITsFlexible在预测CDR3柔性方面表现出高准确性
研究进一步评估了ITsFlexible(在通用蛋白环上训练)对抗体与TCR CDR3柔性的预测能力。模型在ALL-conformations数据集中针对CDR3子集进行测试,这些测试集与训练和验证集严格无重叠,即序列同一性不超过80%。初步评估以晶体结构作为输入。除前文提及的基线模型与基于pLDDT的零样本预测器外,还引入了两种额外的零样本柔性预测流程:其一通过多次AlphaFold2推理、对多序列比对(MSA)进行子采样,依据所得构象多样性推断柔性;其二则使用抗体特异结构预测器ABodyBuilder2(ABB2)的残基级置信度指标——预测均方误差(RMSPE)。
结果显示,ITsFlexible对抗体与TCR的CDR3柔性具有极高预测能力(见图3)。该方法在几乎所有CDR测试集中均优于生物物理基线模型及零样本预测器。唯一例外是CDRA3集合,在此情况下MSA子采样方法略微超过ITsFlexible。然而,ITsFlexible在CDRH3(规模最大且最具代表性的数据集)上显著超越所有其他方法,并且是唯一在四个测试集上均能稳定保持高预测准确度的模型。值得注意的是,ITsFlexible采用了比零样本模型更严格的数据划分标准:其训练集的CDR与测试集间序列同一性低于80%,而ABB2使用的是100%同一性过滤,且AF2训练集中可能存在测试集重叠。
进一步分析ABB2的置信度评分表明,RMSPE更可能反映特定抗体在训练数据中出现的次数,而非真正的柔性特征(见补充章节2.4)。比较四个CDR子集的分类性能(以ROC AUC衡量),L3与B3的结果最佳,H3与A3略低。抗体H3的序列多样性高于L3,因而分类更具挑战;A3性能略差可能与其基因多样性模式不同于B3有关,但A3数据集样本量较小,也可能导致性能估计偏差。
此外,研究者评估了当输入为不同晶体结构(但环序列相同)时,ITsFlexible预测结果的一致性。结果显示预测得分差异普遍较小,表明模型具有良好的稳定性(见补充图7)。鉴于已解析结构仅覆盖部分已知抗原受体序列,ITsFlexible还以ImmuneBuilder(IB)与AlphaFold2生成的预测结构作为输入进行了验证。在抗体CDR数据集上,IB模型的预测性能与晶体结构几乎相当,而AF2模型略逊一筹。虽然模型质量与预测误差间未呈明显线性关系,但总体上质量较低的模型倾向于产生更大的预测误差(见补充图8),这可能解释了AF2模型性能略低于IB模型的原因(见补充表7)。在TCR数据集中,AF2模型在PR AUC上略优于IB模型,但在ROC AUC上差距更大。与抗体类似,AF2模型在TCR数据中预测误差更大,模型精度较低(见补充表7),但总体分类指标未受显著影响。
在整个研究中,CDR柔性的判定遵循以下定义:CDR3区定义为国际免疫基因组信息系统(IMGT)编号107–116(见方法与补充图4);结构多样性以环残基对齐后的RMSD衡量;构象定义为任意两成员间RMSD低于1.25 Å的结构簇。研究还在若干变体数据集上进行了补充分析,包括以二级结构精确定义CDR3(见补充表8)、以框架残基对齐计算柔性(见补充图9)以及使用不同RMSD阈值(见补充图10)。结果表明,ITsFlexible在前两种实验中保持了相似的高预测准确性,而RMSD阈值的变化仅对分类结果产生轻微影响。
2.4 ITsFlexible在分子动力学模拟中准确复现了CDR柔性
虽然晶体学数据能够揭示CDR的构象状态,但并不能直接测量其柔性,仍有可能存在尚未捕捉到的其他构象状态。因此,ALL-conformations数据集的一个局限在于:相较于柔性标签,其刚性标签的置信度更低。尽管研究中通过额外约束进行了修正(见方法部分),但真实的柔性范围仍可能被低估。
为进一步验证模型的可靠性,研究者利用分子动力学(MD)模拟对CDR柔性进行了分类,选取19个抗体进行模拟并标注其CDR3的柔性状态(见补充表2)。结果显示,在MD模拟中观测到的柔性CDR3比例显著高于晶体结构数据:CDRH3中柔性比例为84%,CDRL3为37%,而晶体数据中分别为64%与18%。ITsFlexible在CDRH3的分类中实现了近乎完美的区分,对于CDRL3的预测性能略低(见图4)。这一结果表明,ITsFlexible不仅能从实验结构中识别柔性特征,也能捕捉到反映分子动力学中物理驱动柔性变化的信号。
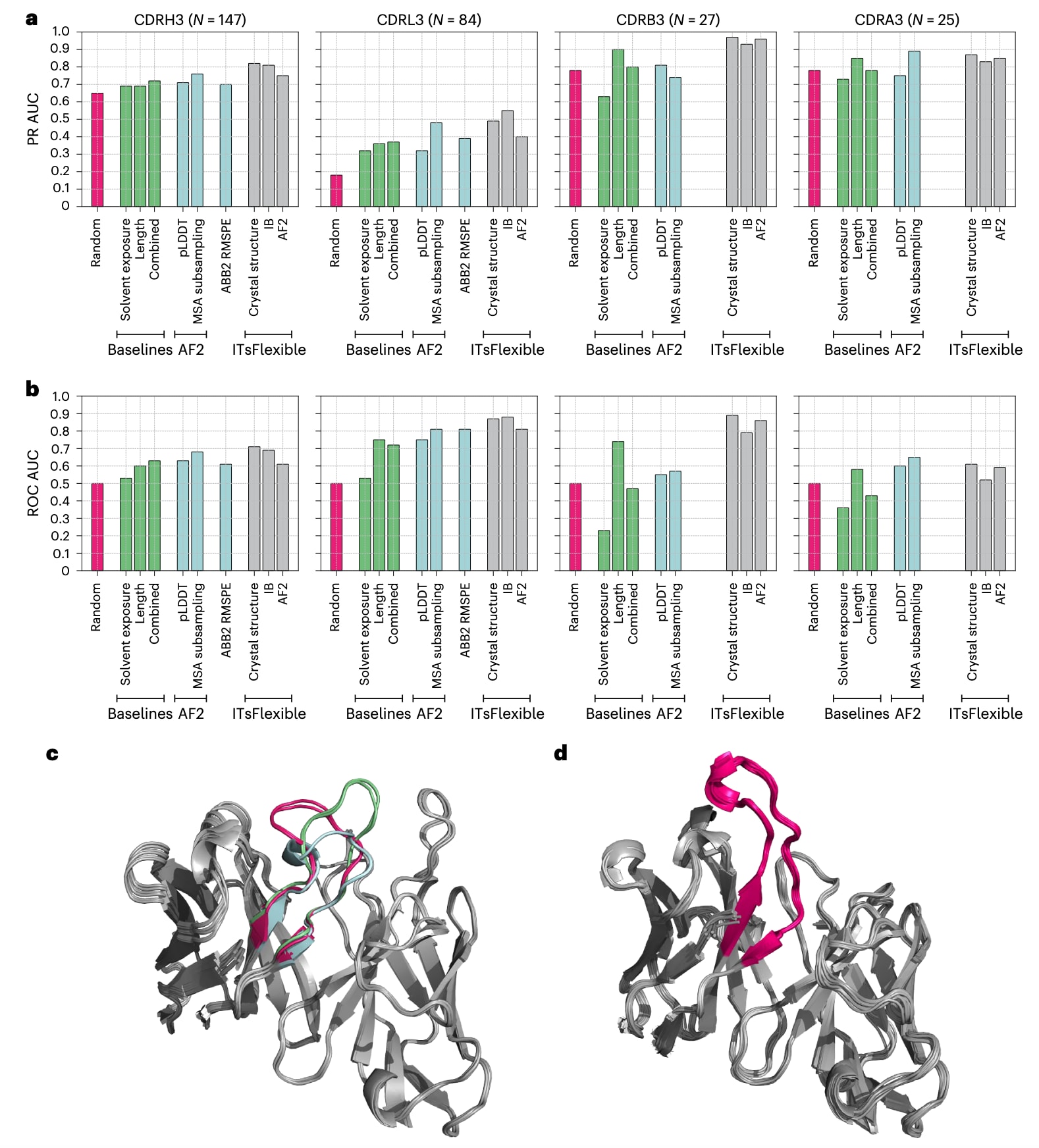
图3|ITsFlexible在CDR测试集上的性能评估。 a、b,对四个测试集的分类结果进行评估,指标为PR AUC(a)与ROC AUC(b)。浅灰色柱表示ITsFlexible模型在晶体结构、IB模型及AF2模型输入下的性能,并与随机分类(红色)、三种基于生物物理特征的基线模型(绿色)以及三种基于蛋白结构预测工具输出的零样本模型(蓝色)进行比较。具体PR AUC与ROC AUC数值见补充表9与表10。
c、d,展示ITsFlexible预测为柔性与刚性的抗体实例。c,叠加显示同一抗体Fv的六个结构,CDRH3被预测为柔性,晶体结构显示其CDRH3(彩色部分)呈现三种不同构象(红、蓝、绿)。d,叠加显示同一抗体Fv的22个结构,CDRH3被预测为刚性,在所有结构中均保持相同构象(红色)。
2.5 冷冻电镜实验验证了ITsFlexible对柔性的预测
研究者选取了三种抗体作为具有挑战性的实验验证对象,利用冷冻电子显微镜(cryo-EM)进行柔性检测。候选抗体来自针对流感病毒H1N1血凝素(HA)的专利与文献抗体数据库,筛选标准为ITsFlexible预测得分低于0.1(刚性)或高于0.5(柔性)。最终选择了三种与训练集序列相似性较低的抗体,并特意挑选了在ALL-conformations数据集中呈现相反趋势的环长度——刚性抗体具有较长环,而柔性抗体具有较短环。三种抗体均以与抗原复合物的形式成像,通过分析密度图中的异质性来反映构象多样性。
抗体1的CDRH3长度为19个氨基酸,比ALL-conformations中86%的CDRH3更长,ITsFlexible预测其为高置信度刚性(得分0.02)。冷冻电镜结果显示,经二维分类筛选出的高质量粒子大多属于单一、均一的三维结构类别,可用于建立高分辨率共识结构(见图5)。该结果与模型预测一致,表明CDR仅呈现单一构象。
抗体2的CDRH3较短(16个残基),被预测为高置信度柔性(得分0.76)。初步图像处理显示其抗体–抗原结合界面存在明显柔性特征,但分辨率较低。通过进一步三维分类与数据采集,获得了其中一种结合状态的高分辨率结构(见图5)。尽管分辨率不足以构建不同构象的精确模型,因此无法明确柔性位置,但密度图清晰显示结合界面存在构象异质性,这种多样性由抗体诱导,极可能源于CDRH3的柔性(见补充视频1与视频2)。
抗体3的CDRH3更短(13个残基),预测得分为0.60,置信度较低。冷冻电镜结果与预测不符,未观察到显著构象异质性(见图5)。这可能与实验设计有关——由于cryo-EM对粒子尺寸的要求,实验中成像对象为抗原结合状态的抗体,而结合可通过额外分子相互作用引入约束,从而使部分残基刚化。研究者推测,ITsFlexible预测的柔性更符合抗体游离状态下的真实情况(详见讨论部分)。在抗体3中,CDRH3尖端的谷氨酸残基与抗原形成了三个氢键,这种约束可能导致柔性在实验条件下未被观测到。相反,抗体1的游离状态可能比实验所示更为柔性。总体而言,数据清楚表明,在相同实验条件下,抗体1的柔性显著低于抗体2,与模型预测高度一致。
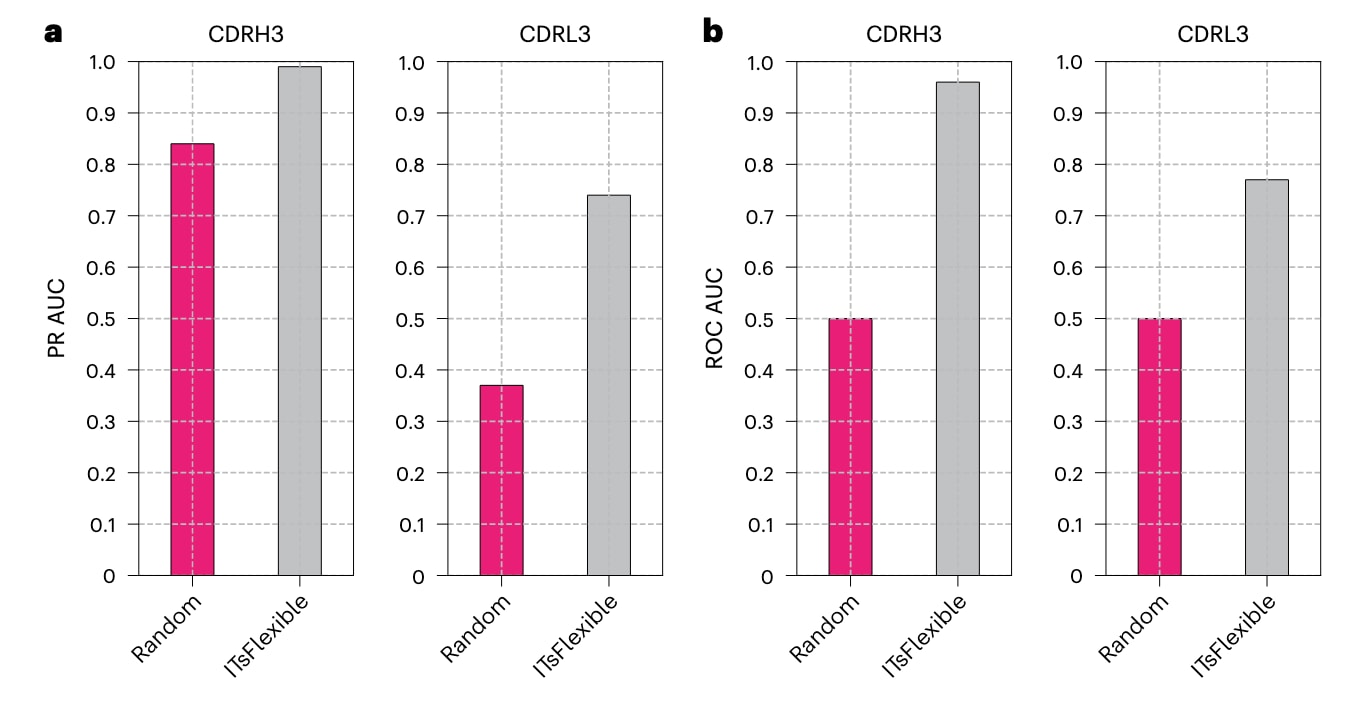
图4|ITsFlexible在包含19种抗体的MD测试集上的性能评估。 a、b,对CDRH3与CDRL3的分类结果进行评估,指标为PR AUC(a)与ROC AUC(b)。每个代表性结构均取自分子动力学(MD)模拟集合,分类依据该集合中观测到的最高ITsFlexible得分进行。补充表17中给出了精确的PR AUC与ROC AUC数值,以及基于集合平均ITsFlexible得分计算的性能结果。
3 讨论
构象变化是许多蛋白质功能特性的核心基础。对于抗体与T细胞受体(TCR),尤其是功能最关键的CDR3,其柔性直接影响亲和力与特异性等关键性质。然而,目前的结构预测工具仍难以准确捕捉蛋白的多构象状态,特别是在抗原受体CDR的情形中。限制这一领域进展的主要障碍之一,是缺乏足够大规模的数据集来支持机器学习模型的训练与评估。
该研究聚焦于抗原受体CDR3柔性的计算预测。为弥补数据不足的问题,研究者收集了所有蛋白中具有相同二级结构模式(由两条连续反平行β链界定的环结构)的结构模体。通过系统挖掘PDB数据库,构建了ALL-conformations数据集,包含超过120万个此类环的晶体结构,覆盖10万余条独特序列。对该数据集的柔性分析标注了2万个高置信度的环结构,其中柔性环可呈多种构象,而刚性环仅呈单一状态。
基于ALL-conformations,研究者开发了ITsFlexible模型,用于将CDR环分类为柔性或刚性。模型在PDB中通用蛋白的环模体上训练,并在分布外测试集中评估其对CDR柔性的预测能力。ITsFlexible在晶体结构测试集上达到了目前最优性能,并能有效泛化至分子动力学(MD)模拟数据。此外,当输入为预测结构(而非实验晶体结构)时,模型仍能保持相似表现,显示其在无解析结构抗体上的潜在适用性。输入特征消融实验揭示了影响CDR柔性的主要生物物理因素,与先前MD研究结果一致,证实CDR周围残基的结构环境对柔性具有关键作用。
进一步分析表明,现有蛋白结构预测工具的不确定性评分及基于结构集合的多样性指标,并不能可靠反映CDR柔性。AlphaFold2的pLDDT虽被视为无序区域的良好指标,但在CDR柔性预测中表现不佳;抗体特异模型ABodyBuilder2的预测误差亦更多地反映序列在训练集中出现频次,而非真正的柔性特征。MSA子采样生成多构象的方法虽然较上述置信度指标更具相关性,但依旧难以准确刻画CDR的柔性。
在实验验证中,研究者选取了三种序列与训练集低相似度、且环长度与数据趋势相反的抗体,通过冷冻电镜(cryo-EM)成像其与抗原结合的复合物并分析密度图异质性。三者中有两例实验结果与模型预测一致——一个刚性、一个柔性,其中刚性抗体的CDRH3更长,这进一步说明模型捕捉到了除环长度以外与柔性相关的特征。第三个抗体的CDRH3更短,预测为柔性但置信度较低,实验中未发现显著构象异质性。总体而言,cryo-EM实验提供了有力证据,表明ITsFlexible能够准确反映CDR3在溶液中的真实构象动态。
该研究的局限之一在于柔性的定义方式。CDR被标记为柔性仅取决于是否观测到多个构象,而不区分抗体是结合状态还是游离状态。既往研究显示,结合态的构象常包含于游离态的构象集合中,这说明抗体结合更常通过“构象选择”而非“诱导契合”机制完成。因此,ITsFlexible的预测结果应能代表游离态CDR的柔性,这一假设也得到游离抗原受体MD模拟结果的支持。由于某些功能特性(如亲和力)取决于结合态与游离态柔性的平衡,未来可以进一步探索结合后柔性变化的预测。然而,目前受限于抗原条件下CDR构象数据的稀缺,该方向仍具挑战性。
该研究公开了ALL-conformations数据集与ITsFlexible模型。ALL-conformations全面捕捉了反平行β链间环状结构的实验构象多样性,为深入研究蛋白环动力学与开发更鲁棒的构象预测模型提供了基础。ITsFlexible则实现了对CDR构象柔性的高精度预测,可为药物设计提供重要参考。CDR柔性通常与亲和力下降及多特异性增加相关,而这两种特征在治疗性抗体设计中往往是不利的。ITsFlexible能够实现候选分子的快速筛选,从而帮助识别具有更优治疗性质的受体。此外,柔性预测还可用于确定哪些抗体最值得进行高计算代价的MD模拟,从而优化计算资源的使用。既有研究表明,将柔性分子的结构集合纳入后续任务(如抗体–抗原对接)中,可显著提高结果精度。总体来看,ALL-conformations与ITsFlexible的建立为未来更复杂的任务——如多构象状态采样——奠定了基础。
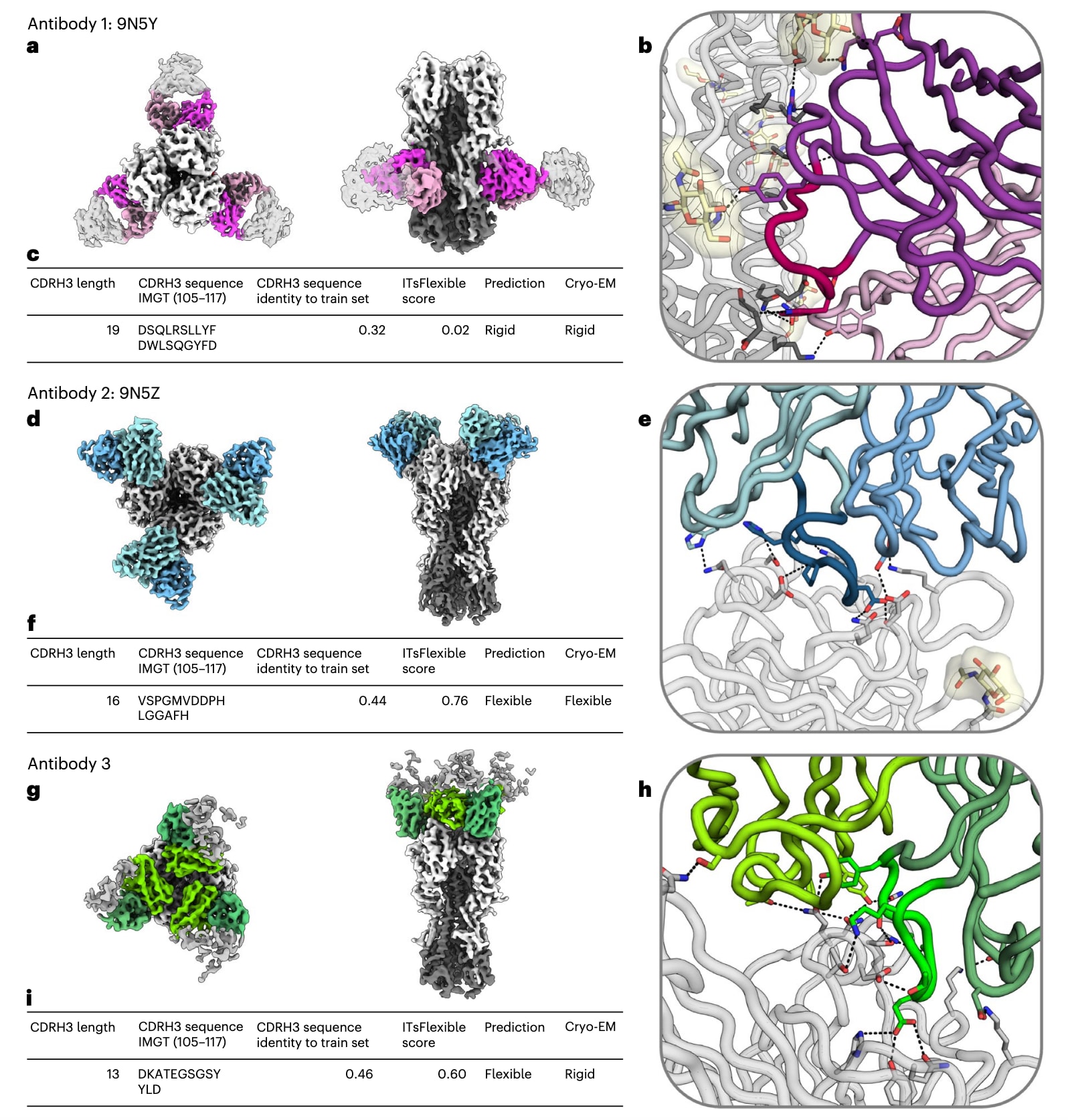
图5|用于冷冻电镜实验的抗体案例研究。 解析了三种与流感病毒H1N1血凝素(HA)结合的抗体复合物的高分辨率结构。 a、d、g,展示抗体与抗原复合物的冷冻电镜密度图的顶视图与侧视图(抗体重链为深色,轻链为浅色,抗原为灰色)。每个结构中均捕获到三个对称排列的抗体分子。抗体1(9N5Y,a)结合于HA茎部,而抗体2(9N5Z,d)与抗体3(g)结合于HA头部。 b、e、h,显示抗体–抗原结合界面的卡通表示图,分别对应抗体9N5Y(b)、9N5Z(e)与抗体3(h)。CDRH3区域以不同色调标示,关键结合相互作用以棒状表示。 c、f、i,总结表展示了抗体9N5Y(c)、9N5Z(f)与抗体3(i)的相关信息,包括CDRH3的长度与序列、与ITsFlexible训练集中最相似样本的序列同一性、ITsFlexible预测得分(数值越高表示柔性可能性越大)、基于预测得分的二元柔性判断结果以及通过冷冻电镜实验确定的柔性状态。三种抗体的补充元数据详见补充表21。