AS 2024 | GeminiMol: 构象空间表征提升了基于人工智能的配体驱动药物发现中的通用分子表示能力
今天介绍的研究成果发表于 Advanced Science ,题为**“GeminiMol: 构象空间表征提升了基于人工智能的配体驱动药物发现中的通用分子表示能力”**。该工作聚焦于分子表示学习的核心问题——如何让AI不仅理解分子的化学结构,更能捕捉其动态构象特征。现有分子表示模型大多停留在二维结构层面,忽视了分子在生理环境中的三维构象多样性与动态变化,而这些恰是决定药物作用机制、靶点识别及细胞行为的重要因素。
GeminiMol通过引入构象空间相似性(Conformational Space Similarity, CSS)描述符,建立了一种基于对比学习的分子表征框架,能够学习分子结构与构象空间间的复杂映射关系。尽管仅使用约3.9万条分子数据进行预训练,该模型在虚拟筛选、靶点识别、QSAR建模与ADMET预测等多项任务中均取得了优于传统分子指纹和主流模型的性能。GeminiMol展示了构象空间信息在提升模型泛化性与发现新活性分子方面的巨大潜力,为未来基于AI的分子设计和药物发现提供了新的研究方向。

获取详情及资源:
0 摘要
分子表示模型是一种神经网络,用于将分子表示形式(如SMILES或分子图)转化为特征向量,是广泛应用于人工智能驱动药物发现场景中的核心模块。然而,现有的分子表示模型很少考虑分子的三维构象空间,从而忽略了小分子的动态特性,以及构象空间在体现分子性质异质性方面的关键作用,例如多靶点作用机制、对不同生物分子的识别能力、以及在细胞质和膜环境中的动态行为。
该研究提出了一种新的模型——GeminiMol,将构象空间表征融入分子表示学习之中,能够提取反映分子结构与构象空间复杂相互作用的特征。尽管GeminiMol仅在相对小规模的数据集(39290个分子)上进行预训练,但其在67项分子性质预测、73项细胞活性预测以及171项零样该任务(包括虚拟筛选与靶点识别)中均表现出均衡且优异的性能。通过有效捕捉分子的构象空间特征,这一策略为快速探索化学空间提供了新途径,并推动了药物设计范式的革新。
1 引言
如果说表示学习是人工智能在面对复杂实体时的“感知受体”,即将文本、图像和声音等人类可读信息转化为智能体可理解的数字表征的调节器,那么分子表示学习便是负责将人类可读的分子表示(如SMILES或分子图)翻译为特征向量的“解释器”,使AI模型能够据此开展药物发现任务。分子表示学习通过预训练任务让模型理解分子表示形式,从而提升其在各类下游任务中的表现,这一思路也被称为元学习(meta-learning)。
近年来,科研界主要形成了三种分子表示学习的训练策略:自监督学习、监督学习与复合策略。自监督学习是一种广泛应用的训练方式,旨在让模型理解不同格式的分子表示,包括SMILES、IUPAC命名、InChI、分子图、三维结构、分子指纹甚至分子图像等。代表性模型有SMILES-Bert、ChemBERTa、KPGT、NYAN、GraphMVP、GEM、UniMol、3DGCL、ImageMol等。通过对多种分子表示的对比学习或自生成任务,自监督预训练模型能够在不依赖结构标签的情况下有效提取分子结构特征。然而,这类方法需要在预训练阶段学习上百万个未标注的分子结构,因此其在远离训练数据的化学空间中的泛化能力仍难以评估。有研究指出,部分自监督策略在下游任务中的表现并不一定优于未经过预训练的方法。
监督预训练策略则通过结构–性质投射或结构–标签语义匹配来捕捉分子的有机化学或药理特征,如ChemBERTa-2、MolT5、CLOOME等。此类方法需要利用不同类型的分子性质构建预训练任务,既可以是分类或回归任务,也可以是与分子描述符或生物医学数据相关的检索与生成任务。其主要缺点在于对标注数据的依赖,同时在预训练中引入分子特征信息也可能影响模型的泛化能力。
为综合两类方法的优缺点,一些研究提出了复合策略,如HelixADMET。这类策略采用分阶段预训练流程:先进行自监督预训练(包括自生成、分子指纹生成、三维几何预测等任务),再执行监督预训练(包括分子性质预测等任务)。这种组合式方法在多数下游任务中展现出更优性能。然而,复合策略也存在局限,如自监督阶段化学空间表征不足、监督阶段依赖高质量大规模数据,以及在泛化性方面的相对欠缺。
针对化学空间表征与泛化能力不足的问题,该研究提出了一种混合对比学习框架,通过分子间相似性作为投影头进行分子间对比学习,从而在不依赖实验性质数据的前提下,训练出可靠的分子表示模型。基于此策略,构建了新的分子表示模型——GeminiMol,该模型在学习药物分子的构象空间相似性(Conformational Space Similarity, CSS)的同时,整合了完整的分子构象空间与药效团特征。研究总体流程如图S1所示:首先定义了构象空间相似性描述符;然后基于39290个分子的较小数据集生成CSS描述符用于训练GeminiMol;最后在四个关键药物发现任务上验证模型性能,包括虚拟筛选、靶点识别、定量构效关系(QSAR)以及吸收、分布、代谢、排泄与毒性(ADMET)性质预测。广泛的实验结果表明,GeminiMol在多种药物发现场景中展现出优异的泛化能力,拓展并深化了分子表示学习领域的研究范式。
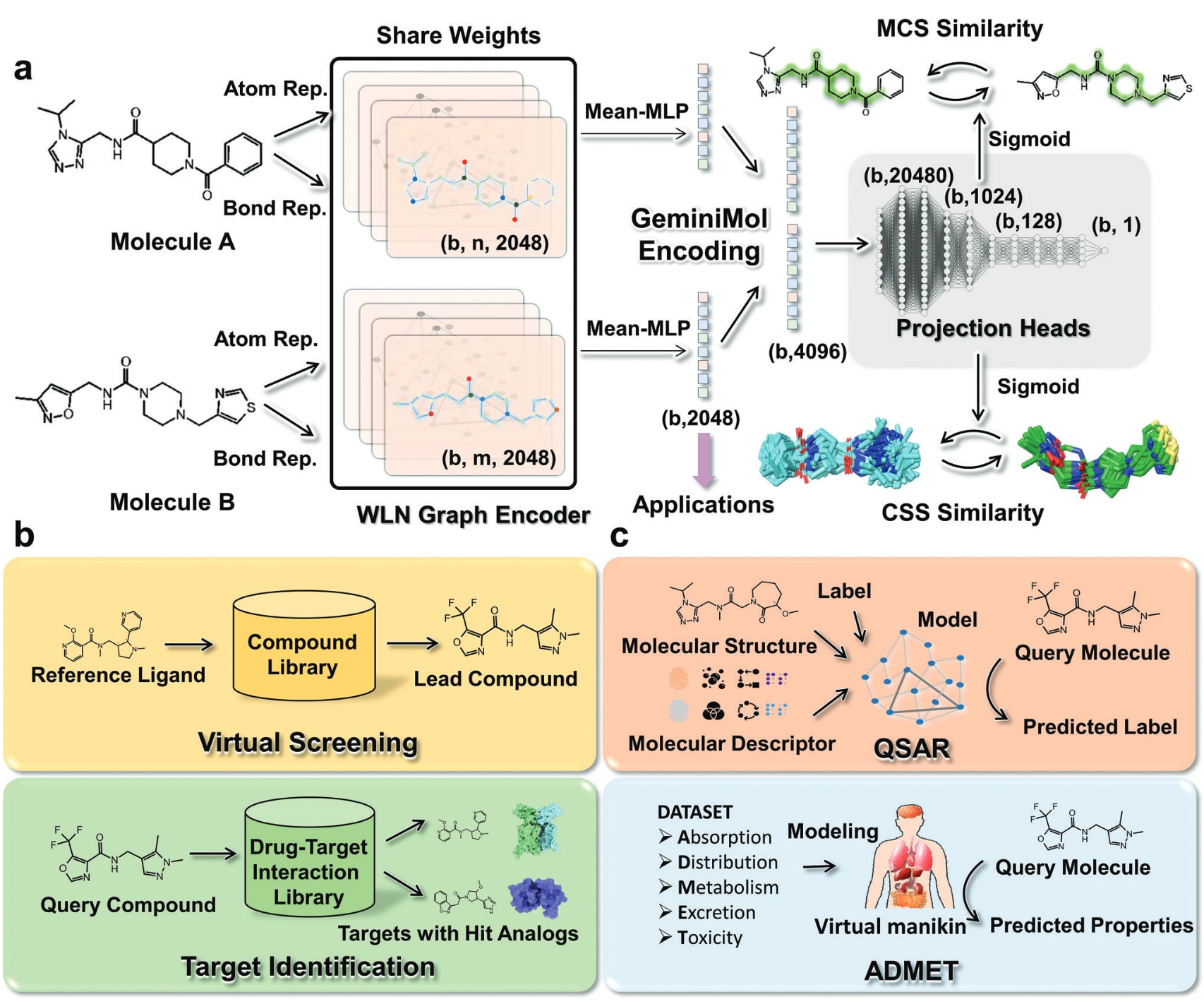
图1 | 对比学习框架的构象空间表征与应用任务 a) GeminiMol的预训练与应用框架。在预训练阶段,模型同时考虑了最大公共子结构(MCS)相似性与构象空间相似性(CSS)。其中,CSS描述符通过PhaseShape计算(详见方法部分),MCS相似性则通过最大公共子结构比对获得。分子以图结构的形式输入网络,其中原子被表示为节点,化学键被表示为边。图中直箭头表示不同网络组件之间的信息流向。高亮的二维分子结构展示了分子间的最大拓扑公共子结构(忽略元素差异)。各网络层之间的特征向量维度标注在括号中。b) GeminiMol的零样本应用任务。通过比较表示向量之间的相似性,或通过投影头预测分子间的MCS或CSS相似性,GeminiMol可用于分子相似性预测任务。c) GeminiMol的分子性质建模任务。通过利用表示向量训练解码器或对GeminiMol模型进行微调,可构建基于GeminiMol的预测器,用于分子性质预测等下游任务。
2 结果
2.1 构象空间相似性描述符
分子间的相似性为对比学习提供了重要契机。然而,以往研究往往通过对分子结构进行修改生成分子对(如MolCLR和iMolCLR),而非依据具有药理或物理意义的分子相似性进行学习。为了进一步增强对比学习在分子表示学习中的作用,有必要将具有药理学或物理学意义的分子相似性评价指标引入分子表示学习体系中,如三维形状相似性与药效团相似性。近期,MolCLaSS模型已将三维形状相似性纳入对比学习框架。然而,药物分子的生物活性构象空间在药物与靶标结合中发挥关键作用,而先前工作(如GEM、Uni-Mol、MolCLaSS)所采用的少量最小构象并不能充分代表与靶标结合相关的生物活性构象空间。
为克服现有方法未能完整表征分子生物活性构象空间的局限,该研究采用系统构象搜索方法,为每个分子枚举其所有可能的三维构象,以全面表征分子的构象空间。随后,通过比较两种分子构象空间中三维形状的相似性来评估它们之间的整体相似度。大多数小分子包含多个可旋转键,不同的旋转组合会产生不同的构象。从理论上讲,通过对每个可旋转键进行小角度分区旋转而生成的系统性构象搜索结果,几乎可以覆盖整个分子的构象空间。小分子在与不同蛋白结合口袋相互作用时,可能采用不同的构象。例如,伊马替尼可与四种不同靶蛋白结合,呈现三种不同的结合构象(如图S2a所示),这一现象说明构象信息对于准确描述药物作用性质至关重要。
随着可旋转键数量的增加,小分子的构象空间变得更加复杂,形成更多低能构象簇(如图S2b所示),这意味着其具有更多可供识别不同靶点的可能性。图S3显示了从蛋白质数据银行(PDB)中获得的4518个分子的6670个实验测定本征构象的应变能分布,结果表明可旋转键的数量是决定分子构象空间复杂度的关键因素。因此,需要开发能够表征构象空间相似性(CSS)的描述符,以便通过对比学习更恰当地刻画分子的性质。
GeminiMol编码器的核心原理是基于二维分子结构提取构象空间特征。为实现这一目标,研究者通过不同的CSS描述符监测构象空间特征的获取过程(具体方法见方法部分)。研究构建了一个包含39290个分子的分子数据集,并对其构象空间进行了全面扫描。随后,为每个分子生成了两个构象集合:一个为近本征构象集合,其应变能低于定义阈值(0.5060 kcal·mol⁻¹·每可旋转键);另一个为更大范围的构象集合,其应变能阈值为1.4804 kcal·mol⁻¹·每可旋转键。
通过使用PhaseShape软件对分子对进行药效团和几何形状比对,计算并生成了不同能量阈值下的原始CSS描述符(最大与最小相似性得分)。此外,还设计了用于数据增强的高级CSS描述符,通过对原始CSS描述符进行数值变换获得,包括用于表征最大相似性的MaxSim、最大差异性的MaxDistance、构象空间接近程度的MaxAggregation以及构象空间重叠程度的MaxOverlap(具体细节见方法部分)。
2.2 GeminiMol框架的训练与解释
作为一种分子间对比学习框架,GeminiMol在训练阶段使用相同的编码器独立地对分子对进行编码(如图1a所示)。原始分子图首先通过Weisfeiler–Lehman网络(WLN)的信息传递机制进行更新。随后,从更新后的分子图中读取分子表示,并将两者连接后输入到多个投影头中,以预测不同的构象空间相似性(CSS)描述符和最大公共子结构(MCS)相似度(如图1a所示)。这些投影头的主要作用是指导编码器学习到具有丰富信息的分子表示(即GeminiMol编码)。
与其他常见对比学习框架中仅含有一到两层隐藏层的投影结构不同,GeminiMol的投影头采用由多层线性层和激活函数构成的整流结构(如图1a所示),以压缩输入特征并提炼高维信息。除以分子间相似性描述符作为对比学习目标来训练独立的分子编码器外,研究还考虑到分子相似性问题与自然语言处理中句子相似性问题具有一定相似性。因此,研究者构建了一个基于化学语言的端到端分子相似性预测框架作为基线模型,命名为CrossEncoder。CrossEncoder允许参考分子与查询分子之间的信息交互,通过在ELECTRA模型中添加化学分词器并借助AutoGluon工具实现。
为评估GeminiMol、CrossEncoder、传统分子指纹方法以及其他第三方基线模型的性能,研究将它们应用于多种下游基准任务(如图1b、1c所示)。这些任务的数据集及对应基线包括:1)用于虚拟筛选的DUD-E与LIT-PCBA;2)用于靶点识别的Target Identification Benchmark Dataset(TIBD);3)用于定量构效关系(QSAR)分析的靶点级与细胞级化合物活性数据;4)用于ADMET建模的吸收、分布、代谢、排泄与毒性相关化合物性质数据。方法部分提供了更详细的说明。以上四类任务涵盖了配体驱动药物设计的主要应用场景,可全面评估不同分子表示方法在药物设计任务中的整体表现。与以往研究相比,该研究在零样本学习任务中引入了靶点识别(如图1b所示),并将细胞水平的活性预测纳入分子性质建模任务之中。
CrossEncoder与GeminiMol均基于从37336个分子中随机采样所得的800万个分子相似性描述符进行训练,并在交叉集与测试集上进行评估。需要注意的是,交叉集中的分子对中至少含有一个来自训练集的分子,而测试集中的分子对则完全不包含训练集样本。正如图2a所示,两种模型在测试集与交叉集上的Spearman相关系数高度一致,表明模型具有良好的泛化能力。进一步分析了包含60万个样本的子集(见图S4),发现二维结构差异显著但三维结构相似度较高的分子对极为罕见,可视为高质量的分布外(OOD)样本。OOD测试被广泛用于评估机器学习系统在超越训练集偏差时的泛化能力。因此,基于这些数据,研究进行了消融实验,以探讨GeminiMol模型中各组成部分的学习贡献。如图2b所示,减少层数、降低节点特征维度或移除投影头中的整流结构均会导致性能下降;同样,在高级CSS描述符中也观察到类似趋势(如图2c所示)。
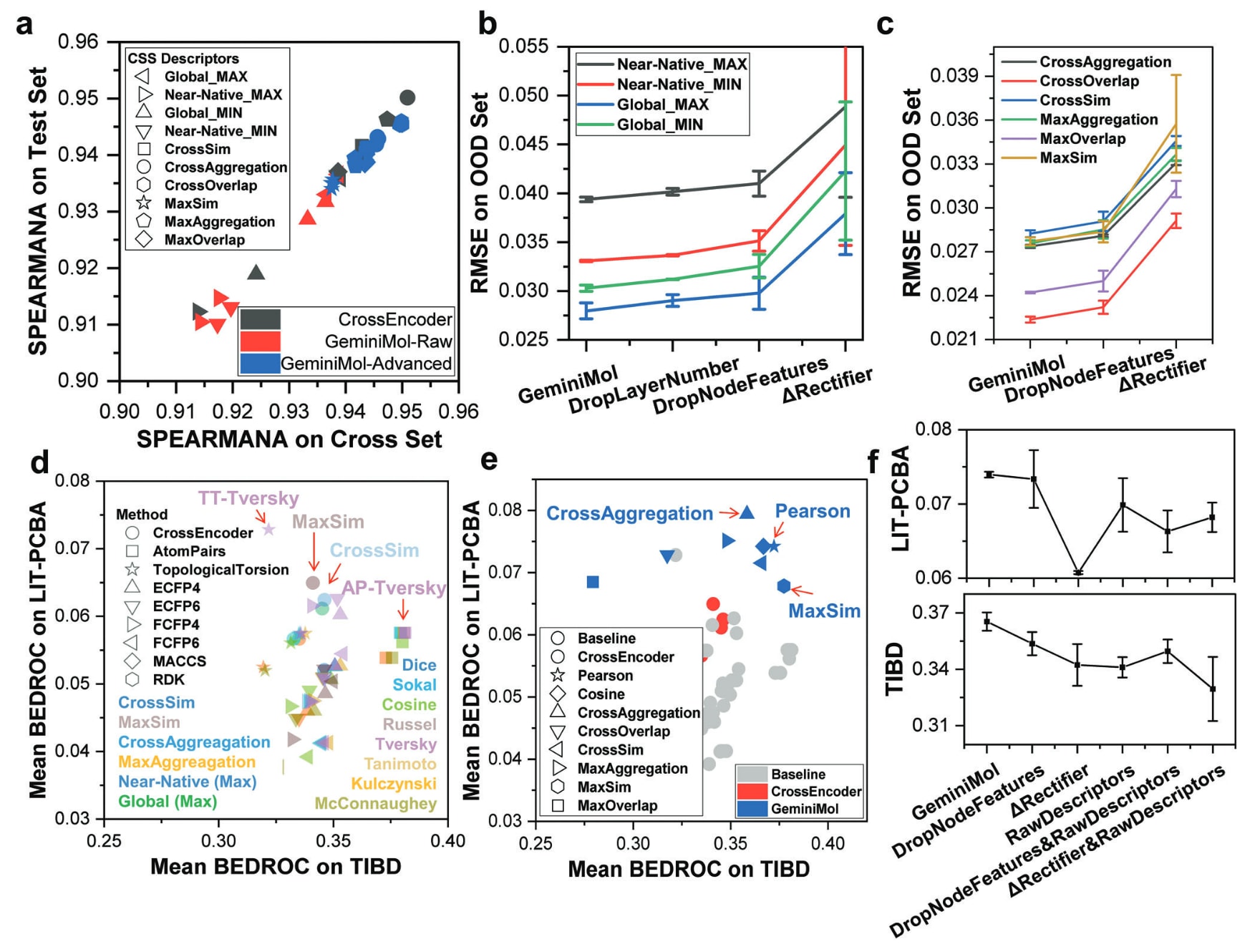
图2 | GeminiMol模型在CSS预测与零样本药物发现任务中的测试与解释 a) 交叉集与测试集性能相关性结果显示,模型具有较强的泛化能力。不同形状代表不同的CSS描述符。例如,Global_MAX表示在全局构象空间中的最大相似性;MaxSim表示所有相似性中的最大值;MaxDistance表示最小相似性;MaxOverlap表示MaxSim与MaxDistance之和的一半;MaxAggregation表示在两种应变能阈值下最大相似性的平均值。当相似性名称中的“Max”被替换为“Cross”时,表示A对B与B对A相似性之和的一半。b) 原始CSS标签的消融实验结果。c) 经数值变换后的CSS描述符的消融实验结果。GeminiMol模型在每组实验中进行了四次独立的训练与测试,而其他模型至少进行了两次重复实验。“DropLayerNumber”表示将WLN网络的层数减少至2层;“DropNodeFeatures”表示将WLN网络中节点特征维度降至1024;“ΔRectifier”表示将投影头替换为简化的深度神经网络。d) CrossEncoder与分子指纹方法的性能比较。虚拟筛选性能以LIT-PCBA基准测试中的Boltzmann增强受试者工作特征曲线判别指标(BEDROC)衡量,靶点识别性能以TIBD基准测试中的BEDROC指标衡量。选择LIT-PCBA数据集的原因在于其中的分子来源于真实的高通量筛选实验。红色箭头标示性能最均衡的模型。不同的分子表示方法与指纹类型以不同形状表示,而相似性度量指标以不同颜色区分。e) 不同分子指纹、CrossEncoder与GeminiMol的性能比较。各类CSS描述符以不同形状表示,基线方法计算的相似性以灰色圆点表示。GeminiMol编码向量的皮尔逊或余弦相似性均显示出优越的平均性能。f) GeminiMol模型及CSS描述符的消融实验结果。在零样本药物发现实验中,采用皮尔逊相似性作为度量指标。GeminiMol模型在每组实验中均进行了四次训练与测试,而其他模型至少进行了两次重复实验。“DropLayerNumber”表示将WLN层数减少至2层;“DropNodeFeatures”表示将节点特征维度降至1024;“ΔRectifier”表示将投影头替换为简化的深度神经网络;“RawDescriptors”表示在训练过程中使用原始CSS描述符替代高级CSS描述符进行对比学习。
2.3 基于GeminiMol编码的零样本药物发现
在基于配体的药物设计领域中,一个核心假设是“结构相似的分子往往具有相似的生物活性”。这一原则使得研究者能够通过比较配体间的相似性来预测其生物活性的相似程度,即与参考分子在结构上相似的分子可能具有相似的活性或作用靶点。由此,配体相似性分析被广泛应用于虚拟筛选与靶点识别等任务。在机器学习领域,这种方法通常被称为零样本学习(zero-shot learning)。因此,分子相似性搜索成为实现零样本药物发现的主要途径。
在GeminiMol中,可通过两种方式进行分子相似性预测:一种是利用投影头直接预测分子间的构象空间相似性(CSS)描述符;另一种是比较GeminiMol编码向量之间的相似性,例如计算余弦相似度或皮尔逊相关系数。值得注意的是,该研究所用的分子数据集(39290个分子)与DUD-E(1463336个分子)和LIT-PCBA(2808885个分子)重叠极少,因此模型必须在学习二维结构与CSS之间关系的同时,具备较强的泛化能力,才能在下游任务中取得优异表现。
PhaseShape是一种基于三维药效团与形状比对的相似性打分方法,该研究以其作为参考,评估了GeminiMol和CrossEncoder在虚拟筛选任务中的表现,以探究引入构象空间表征是否有助于性能提升。如图S5所示,GeminiMol的多种CSS描述符以及编码向量间的相似性均在DUD-E和LIT-PCBA基准数据集中显著优于PhaseShape。其中,GeminiMol编码向量间的皮尔逊相关与余弦相似度在两个虚拟筛选基准上均表现出最优性能。
为进一步比较不同模型与主流基线方法的差异,研究评估了8种常用分子指纹、CrossEncoder以及GeminiMol在LIT-PCBA和TIBD数据集上的表现。分子指纹作为主流二维结构相似性搜索方法被用作基线(如图2d、2e所示)。CrossEncoder的表现与分子指纹相当,这主要得益于MaxSim与CrossSim预测器的贡献,但整体未能超越分子指纹方法。令人意外的是,GeminiMol编码的余弦相似度与皮尔逊相关在虚拟筛选与靶点识别任务中均取得了平衡且优异的表现。其中,皮尔逊相关系数在两个任务上整体效果最佳。
进一步的消融实验(如图2f所示)表明,去除投影头中的整流结构会显著削弱GeminiMol的性能与稳定性,这与前文的实验结果一致。同时,减少节点特征维度或将高级CSS描述符替换为原始CSS描述符均会导致模型在虚拟筛选与靶点识别中的性能下降。
2.4 利用GeminiMol编码识别新的生物活性分子
在药物发现过程中,为满足化合物新颖性的要求,分子表示模型必须能够有效捕捉分子间潜在的相似性,即便它们在二维结构上存在显著差异。这一能力在实践中尤为重要,尤其是在识别具有不同骨架结构的新活性分子时。与多种基线方法相比,GeminiMol在零样本学习任务(如虚拟筛选与靶点识别)中表现出卓越性能(见图2e)。为了深入理解其优异表现背后的原因,研究进一步探讨了GeminiMol对分子结构变化及药物骨架跃迁(scaffold hopping)的敏感性,并以三个不同的靶点为例进行了分析。
首先,为评估常见环闭合操作对先导化合物优化的影响,研究对比了GeminiMol编码、ECFP4指纹与TopologicalTorsion指纹的表现(如图3a所示)。选取了两种非甾体类雌激素受体调节剂——拉索福西芬(lasofoxifene)与阿非莫西芬(afimoxifene)。二者作用于相同的雌激素受体,且在结构上高度相似,具有相近的结合模式(见图3a),其相似性通过视觉比对即可辨识。然而,分子指纹法显示二者在二维结构上存在显著差异。而GeminiMol则能够识别这类微妙的结构变化,将它们判断为具有高度相关性的类似物,预测的MaxSim值为0.942,对应编码相似度超过0.7。
人类免疫缺陷病毒(HIV)蛋白酶是药物研发中的经典靶点之一,相关研究历史悠久。早期研究主要集中于拟对称肽类抑制剂,随后发展出多样的化学骨架。尽管这些新型抑制剂在结构上与原始肽类抑制剂差异明显,但其作用机制相同。研究以经典的拟对称抑制剂A79285为起点,对比了其与四种非对称抑制剂的结构差异(见图3b)。分子指纹显示这些分子的二维结构差异较大,但在GeminiMol编码空间中,它们彼此距离较近,表明GeminiMol能够捕捉不同结构分子在构象空间中的内在相似性。
同样地,研究还分析了三种凝血酶(thrombin)抑制剂的分子结构及其与靶点的相互作用模式(见图3c)。结果显示,即使这些分子的二维结构差异显著,GeminiMol模型仍能识别出它们之间的基础相似性。更为重要的是,研究发现抑制剂的活性与其相似度呈正相关——与最强抑制剂的相似性越低,其活性也相应降低(见图3c)。这些结果表明,GeminiMol不仅能够超越二维结构的局限,更能通过构象空间特征识别不同骨架化合物间的生物学相关性,为新活性分子的发现提供了强有力的工具。
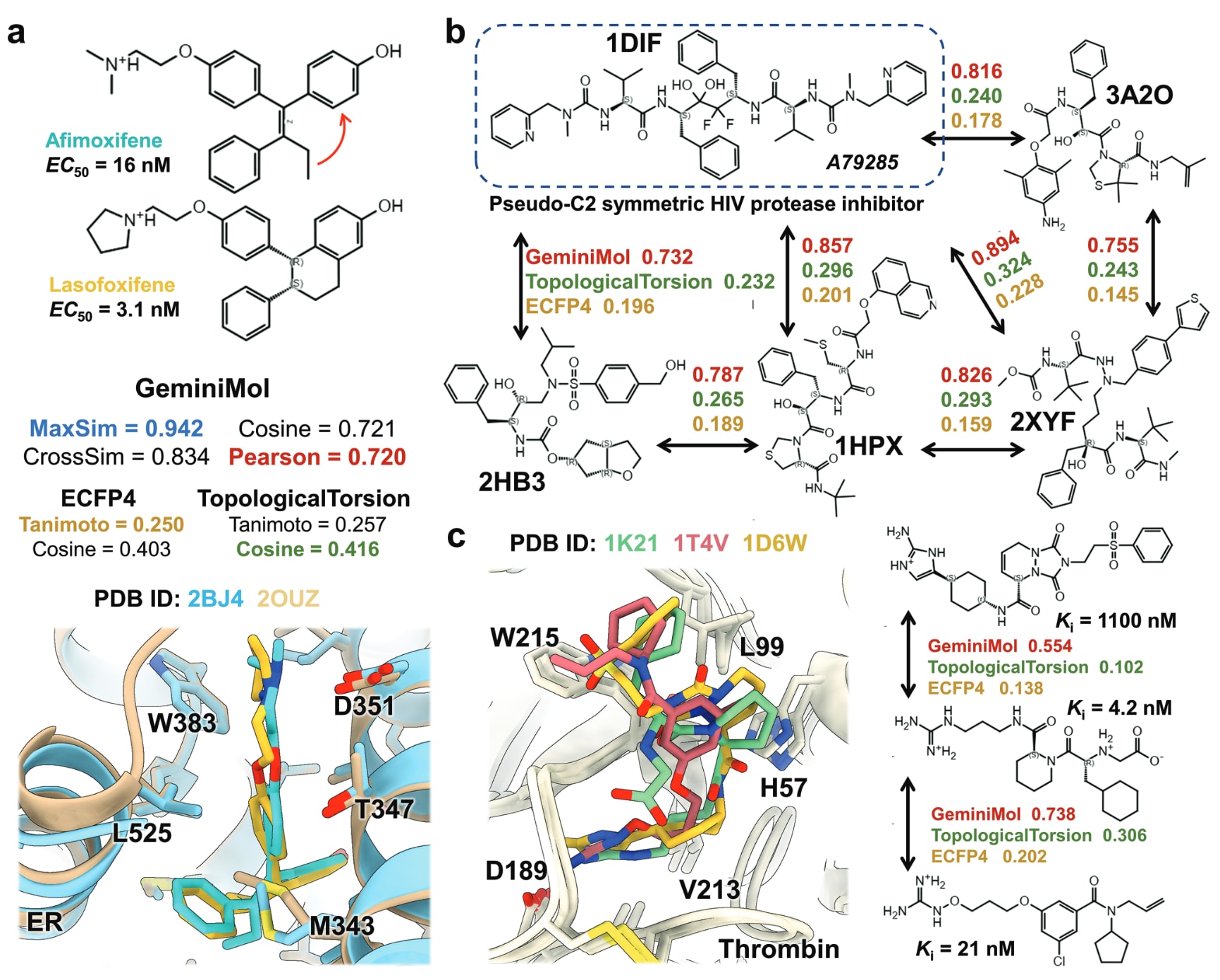
图3 | 基于GeminiMol编码的分子相似性评估 a) 环闭合类化学修饰引起的分子相似性变化。在结构发生变化后,分子指纹的相似性指标显著下降,所有指标均低于0.5;然而,GeminiMol的相似性仍保持在0.7以上,表明其对结构修饰具有较强的鲁棒性。b) 对称与非对称HIV蛋白酶抑制剂的内在相似性。分子指纹无法识别这些具有不同骨架的分子间相似性(得分低于0.4),而GeminiMol编码基于皮尔逊相似性度量,认为这些分子的相似性均高于0.7,能够揭示不同结构分子在构象空间中的共性特征。c) 三种二维结构差异显著但在蛋白结合口袋中表现出相似结合模式的分子。GeminiMol编码的皮尔逊相似性能够准确识别其内在相似性,例如较弱结合分子的相似性得分为0.554,而较强结合分子的得分为0.738。
2.5 GeminiMol编码在分子性质建模中的评估
分子性质建模是评估分子表示模型在下游任务中性能的常用方法。以往研究常使用包含ADMET性质信息与靶点层级生物活性数据的数据集,如MoleculeNet、Therapeutics Data Commons(TDC)、LIT-PCBA及PubChem BioAssays,用以评估不同分子表示方法的表征能力。
该研究为全面考察多种分子表示方法的性能,从LIT-PCBA与PubChem BioAssays中收集了21个靶点层级生物活性数据集,从美国国家癌症研究所药物开发项目(NCI/DTP)中收集了73个细胞层级生物活性数据集,并从TDC中获得了46个ADMET性质数据集,此外还包含一个药物成瘾相关数据集(具体见方法部分)。其中,定量构效关系(QSAR)任务分为两类:靶点层级与细胞层级。前者与特定靶点相关,而后者可能涉及多个未知靶点及细胞毒性机制。
研究首先比较了GeminiMol与多种分子指纹方法在两类QSAR任务及ADMET分类与回归任务中的表现(见图S6)。结果显示,在所有分子指纹方法中,由ECFP4、FCFP6、AtomPairs和TopologicalTorsion组合而成的CombineFP表现最佳。由于细胞层级活性建模在基于表型的药物发现中具有重要意义,研究进一步重点评估了GeminiMol、分子指纹及第三方基线模型(包括FP-GNN与Uni-Mol)在73种癌细胞系上的QSAR任务表现(如图4a所示)。在这些分子表示方法中,GeminiMol模型表现最为优异,显著优于CombineFP与其他基线方法(见图4b)。
为揭示GeminiMol在细胞层级QSAR任务中性能优越的原因,研究进一步比较了不同解码器的设计及其对性能的影响(见图4c)。结果显示,模拟GeminiMol框架中投影头结构的PropDecoder表现最佳。结合图S2可见,伊马替尼能够以多种构象结合不同靶点,这说明构象空间特征在建模多靶点相互作用与复杂药物–靶点网络中发挥重要作用,从而解释了GeminiMol在细胞层级QSAR任务中的卓越性能。
此外,GeminiMol在靶点层级QSAR与ADMET任务中也展现出与其他先进方法相当的性能(见图4d–f)。研究进一步测试了仅使用原始GeminiMol编码作为下游任务输入、且固定编码器的模型表现。在此固定编码器配置下,GeminiMol的性能略有下降,但仍优于大多数基线模型,在QSAR任务中优于FP-GNN,并在ADMET任务中超越部分分子指纹方法(见图S7)。值得注意的是,该固定编码器仅基于37336个分子结构进行训练,这表明GeminiMol的训练策略赋予了模型较强的泛化能力。未来若引入更丰富多样的分子结构,预计将进一步提升其表现。
总体而言,GeminiMol不仅在性能上可与传统分子指纹相媲美,还能与专为QSAR任务设计的FP-GNN以及在预训练中使用1900万个分子的Uni-Mol相抗衡。这表明,该研究提出的基于分子构象空间相似性描述符的对比学习框架是一种高效的分子表示模型训练策略。
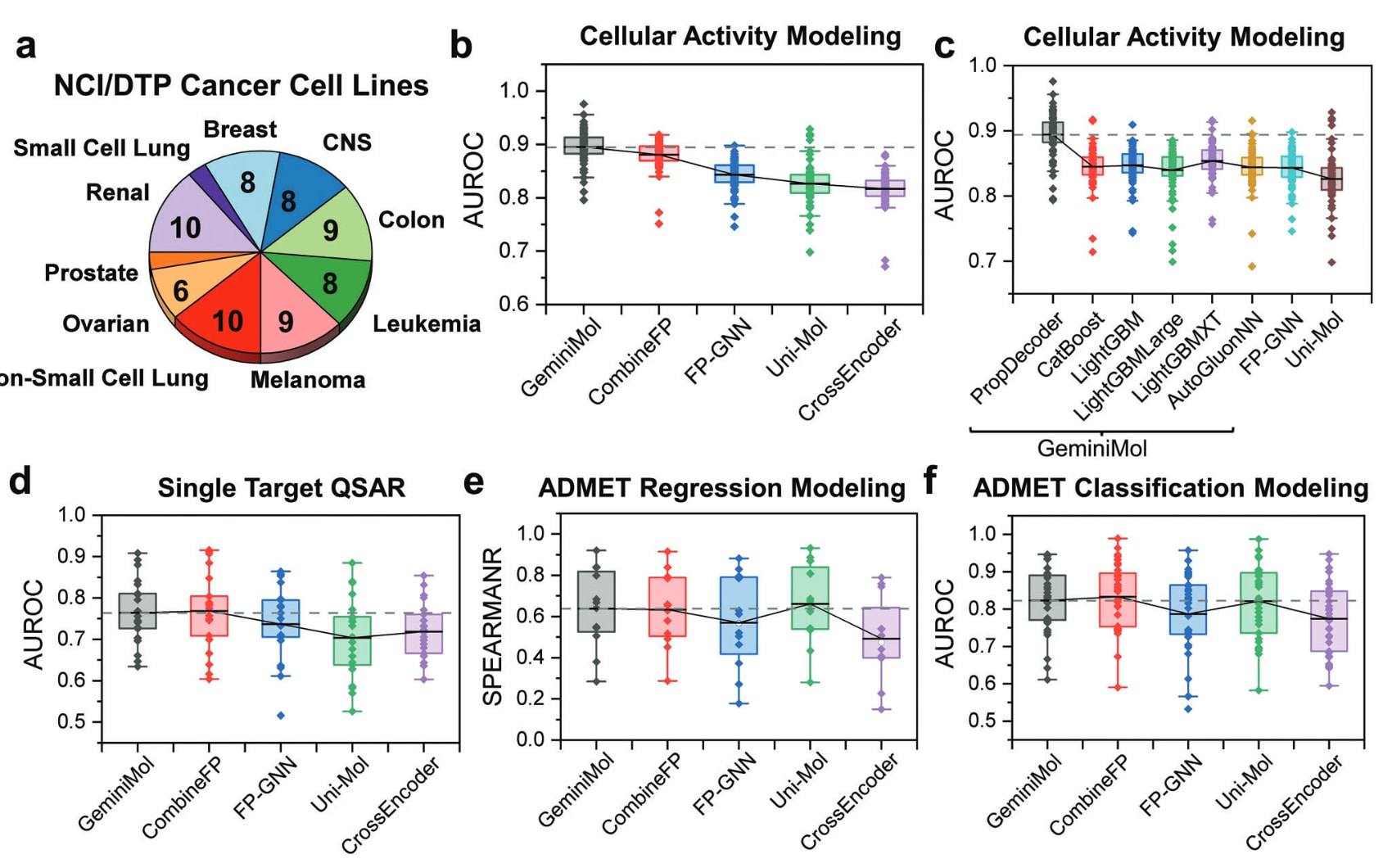
图4 | 基于GeminiMol编码的分子性质建模应用 a) NCI/DTP中73种癌细胞系的统计分布。b) GeminiMol与其他基线方法在73个细胞层级生物活性建模任务中的性能比较。虚线表示GeminiMol模型的表现。结果显示,GeminiMol在大多数细胞系任务中均优于其他模型,展现出更强的表征与预测能力。c) 不同解码器类型对GeminiMol在细胞层级生物活性建模任务中性能的影响。其中,PropDecoder为用于微调GeminiMol模型的多层感知机(MLP);CatBoost、LightGBM与AutoGluonNN则是基于固定编码器构建的预测模型。虚线表示经过微调的GeminiMol-PropDecoder模型性能,其优于其他解码结构。d–f) GeminiMol与其他基线方法在靶点层级QSAR任务与ADMET性质建模任务中的性能比较。包含21个靶点层级QSAR任务、35个分类ADMET任务及12个回归ADMET任务。虚线同样表示GeminiMol模型的表现。整体结果表明,GeminiMol在多种任务类型中均保持稳定且优异的性能,验证了其作为通用分子表示模型在药物发现中的广泛适用性。
3 结论
数据的数量与质量长期以来都是人工智能科学研究中的关键挑战。该研究提出了一种创新性解决方案,即利用高精度分子模拟技术生成高质量训练数据,以缓解深度学习中高质量数据稀缺的问题。通过这种方式,不仅能够独立训练分子表示模型,还可为其他学习策略提供补充性数据来源。研究发现,将构象空间相似性描述符引入分子表示学习,能够显著提升模型的表征能力与在药物发现任务中的有效性。与以往研究不同,该研究在框架设计中避免引入已存在于其他表示方法(如分子指纹)中的信息,而是探索新的分子表征途径,为未来模型的改进奠定基础。
近年来,靶点导向的药物设计一直是药物研发的主流范式。然而,最新研究表明,大多数药物实际上来源于基于细胞水平表型的发现策略,且部分靶点导向药物的效应也涉及非靶点相互作用。这一发现提示,基于配体的药物设计仍具有重要潜力,特别是在骨架跃迁与配体相似性筛选等方向。该研究揭示了当前分子表示模型的改进空间,并推动药物发现范式的潜在转变。随着以Target 2035为代表的全球药物靶点研究计划的推进,基于配体相似性的靶点识别正变得愈发重要。配合GeminiMol等先进的分子表示技术,未来的药物设计有望通过小分子相似性网络实现生物活性预测与靶向选择性优化,从而推动理性分子设计的新范式形成。该研究的成果也将对药物–靶点相互作用预测及药物副作用预测等领域产生积极影响。
需要指出的是,构象空间表征并非药物发现的“万能钥匙”。对于部分任务,化合物的二维结构信息已足以建立可靠的构效关系,此时引入构象空间信息未必带来显著收益。近年来,借助于PDB-Bind等代表性药物–靶点三维相互作用数据集,基于三维相互作用建模的人工智能方法(如PLANET)已被广泛应用于药物–靶点相互作用预测。此外,药物–靶点结合的性质还与靶点信息密切相关,包括结合亲和力与结合动力学等;同时,药物的吸收、分布与代谢过程也常受到其他生物大分子的影响。因此,单纯依赖小分子构象空间信息并不能有效解决所有问题。GeminiMol的目标是改进现有小分子表示方法,并为其他下游任务(如蛋白–配体亲和力预测与条件分子生成)提供实用工具,从而为人工智能驱动的药物设计提供新的技术支撑。