SA 2025 | AMP-Designer: 基于大型语言模型(LLM)基础模型发现具有显著抗菌活性的抗菌肽
今天介绍的这项工作来自 Science Advances。
细菌耐药性(AMR)正成为全球公共卫生的重大威胁,而新型抗生素的研发在过去半个世纪几乎停滞。抗菌肽(AMPs)因其独特的膜破坏机制和低耐药性潜能,被认为是替代传统抗生素的希望。然而,AMP设计面临巨大挑战:序列空间极其庞大、实验筛选代价高昂、活性与毒性难以兼顾。
研究团队提出的 AMP-Designer 利用大型语言模型(LLM)作为蛋白质序列的基础生成模型,通过整合提示调优(prompt tuning)、对比学习、知识蒸馏与强化学习,实现了高效的抗菌肽从头设计。仅用11天,该系统便生成18种具有广谱抗革兰氏阴性菌活性的AMP,其中两种(KW13与AI18)在体内外均展现出优异抗菌效力、低溶血性及高血浆稳定性。更令人瞩目的是,AMP-Designer在数据稀缺条件下仍能精准设计出针对特定菌株(如P. acnes)的高效AMP,为应对全球抗生素耐药危机开辟了全新思路。

获取详情及资源:
0 摘要
大型语言模型(Large Language Models,LLMs)在化学与生物医学研究领域展现出卓越的进步,已成为可广泛应用于多种任务的通用基础模型。研究者提出了一种名为AMP-Designer的基于LLM的方法,用于快速设计具备特定性质的抗菌肽(Antimicrobial Peptides,AMPs)。在仅11天内,AMP-Designer成功实现了18种具有广谱抗革兰氏阴性菌活性的抗菌肽的从头设计。体外实验验证显示,其成功率高达94.4%,其中两种候选肽表现出优异的抗菌效力、极低的溶血毒性、在人体血浆中的高稳定性及较低的耐药性诱导风险,并在小鼠肺部感染模型中显著降低了细菌负荷。整个从设计到实验验证的过程仅耗时48天。
此外,AMP-Designer在针对特定菌株进行AMP设计时依然具备出色表现,即便可用数据有限,其中一条最优候选肽对痤疮丙酸杆菌(Propionibacterium acnes)的最低抑菌浓度仅为每毫升2.0微克。通过整合先进的机器学习技术,AMP-Designer展现出卓越的效率与生成能力,为抗生素耐药问题提供了全新的解决思路。
1 引言
细菌抗菌耐药性(Antimicrobial Resistance, AMR)已成为威胁全球公共健康的重大问题。据估计,2019年全球约有495万人死亡与细菌耐药性相关,其中127万人直接死于细菌耐药感染,且这一数字预计到2050年将上升至每年1000万人。尤其是革兰氏阴性菌,其对现有抗生素的普遍耐药性更为严重。令人担忧的是,自1968年喹诺酮类抗生素问世以来,尚无针对革兰氏阴性菌的新型抗生素类别成功进入临床阶段。
抗菌肽(Antimicrobial Peptides, AMPs)是一类由生物体天然产生、通常由10至50个氨基酸组成的抗菌分子。由于其结构与功能的多样性、良好的抗菌效果以及较低的耐药风险,AMPs被认为是传统小分子抗生素的潜在替代物。然而,相较于小分子药物,AMPs普遍存在活性不足、毒性不确定及在生产与运输过程中易失活等问题,从而限制了其临床应用。研究者因此致力于开发具备高抗菌活性、低溶血性及高稳定性的AMP候选物,以提高其作为抗生素的竞争力。
近年来,计算辅助设计方法逐渐被引入AMP研究。传统设计策略主要依赖于对现有AMPs的结构优化或基于预测模型的大规模筛选,但由于肽序列空间极其庞大(长度不超过32个氨基酸的可能序列约为4.5×10⁴¹种),新型AMP的发现仍极具挑战。目前仅约10种AMP获得监管机构批准,显示出功能性抗菌肽在庞大序列空间中的稀疏分布。为解决这一问题,研究者尝试利用深度生成模型(如RNN、GAN、VAE)探索AMP设计的新方法,但在多属性约束下(如高活性与低毒性并存)的设计依旧困难。传统监督学习依赖有限标注数据,导致生成序列新颖性不足;而条件生成模型(Conditional GAN/VAE)虽能控制特征,但其对标注数据的依赖会引入偏差与预测误差。
随着ChatGPT等大型语言模型(Large Language Models, LLMs)的出现,蛋白质语言建模取得了突破。借鉴自然语言处理的成功经验,大规模序列生成模型被认为可同样适用于AMP设计。相比传统微调(fine-tuning),提示调优(prompt tuning)在计算效率与灵活性上更具优势,能缓解小样本过拟合并在多条件生成任务中平衡多样性与有效性。
基于此,研究者提出了AMP-Designer框架——一种融合GPT、提示调优、对比学习、知识蒸馏与强化学习(Reinforcement Learning, RL)的综合性AMP设计平台。AMP-Designer首先基于UniProt提取的肽序列数据训练AMP专用语言模型AMP-GPT;随后通过对比提示学习(AMP-Prompt)实现特征迁移,并利用知识蒸馏将模型压缩为三层门控循环单元(GRU)网络以降低计算成本。此外,研究者基于AMP-GPT建立最小抑菌浓度(MIC)预测模型AMP-MIC,为不同菌种提供反馈并辅助RL优化筛选。
通过AMP-Designer,研究者在仅48天内实现了18种广谱抗菌肽的从头设计与实验验证,其中两种候选物(KW13与AI18)表现出卓越的体外抗菌活性、低溶血毒性、血浆稳定性以及极低的耐药诱导性,并在小鼠肺部感染模型中显著降低了细菌负荷。AMP-Designer框架具有可即插即用的特性,针对新任务仅需约3天即可完成设计。尤其值得注意的是,即便在标注数据极度稀缺的情况下(如针对痤疮丙酸杆菌,仅有20条数据),AMP-Designer仍能高效生成高活性AMPs,其中三条候选肽经实验验证表现出显著活性。这一成果充分展示了LLM基础模型在蛋白质设计与抗菌肽发现中的巨大潜力。
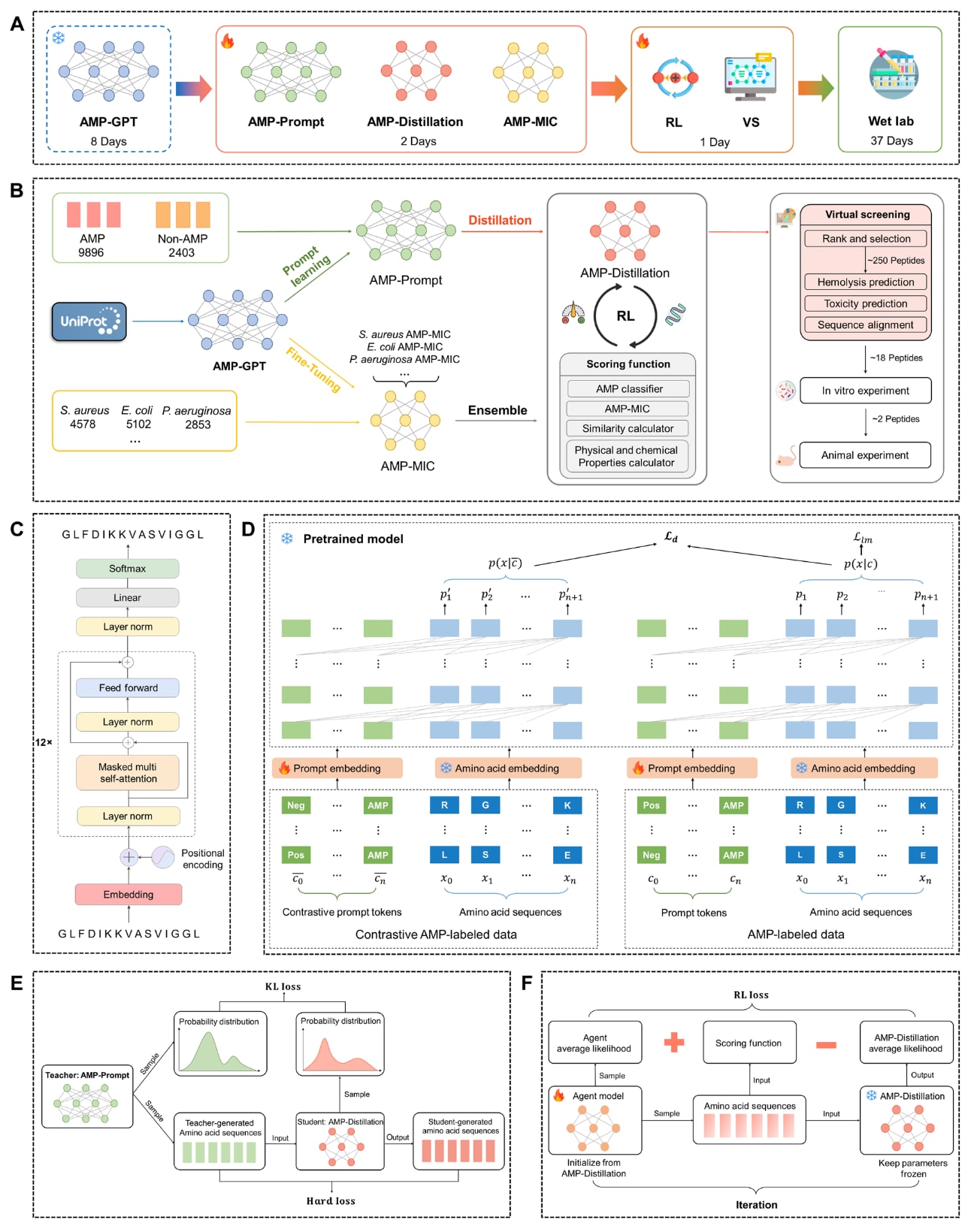
图1 | 展示了AMP-Designer的总体概览。 (A) 展示了利用AMP-Designer进行抗菌肽(AMP)筛选的时间线,包括计算设计、体外筛选及体内验证的完整流程。(B) 展示了AMP-Designer的工作流程(workflow),其中vS表示虚拟筛选(virtual screening)。流程依次包括序列数据准备、AMP-GPT基础模型训练、对比提示微调、模型蒸馏与强化学习优化等步骤。(C) 展示了AMP-GPT的模型结构,基于GPT框架构建的蛋白质语言模型,用于学习肽序列的分布与语义表示。(D) 展示了对比提示微调(contrastive prompt fine-tuning)过程,通过固定AMP-GPT参数,仅训练提示嵌入层以增强抗菌活性特征的可区分性。(E) 展示了模型蒸馏(model distillation)过程,将AMP-Prompt的知识压缩至更轻量的神经网络结构,以提高运算效率并保持生成能力。(F) 展示了强化学习(reinforcement learning, RL)优化过程,通过AMP-MIC预测模型反馈信号,对生成的序列进行活性打分与筛选,从而优化模型生成高活性AMP候选物的能力。
2 结果
2.1 AMP-Designer总体概述
图1B展示了AMP-Designer的整体工作流程及模型训练细节。首先,从UniProt数据库中收集长度不超过32个氨基酸的肽序列,共计630,683条,用于AMP-GPT模型的无监督训练。与基于小规模标注数据的训练相比,利用更大且多样化的数据集可使AMP-GPT学习到更全面的序列表示,这一现象已在大规模语言模型与生物建模研究中得到验证。
接下来,研究目标是在AMP-Designer学习到的肽序列空间中识别具有抗菌活性的序列。为此,从多个AMP数据库中整理出9,896个阳性样本与2,403个阴性样本。利用这些带标签的数据,通过对比提示调优(contrastive prompt tuning)微调模型。在该过程中,首先根据AMP-GPT的词嵌入初始化提示层的嵌入表示,然后在保持AMP-GPT参数冻结的情况下,仅训练提示层参数。经此过程得到的模型称为AMP-Prompt。
在生成阶段,结合top-k采样策略(见“活性分析”部分),AMP-Prompt能够以较高成功率生成AMP序列,并由三种独立的AMP预测器进行性能评估。然而,由于AMP-Prompt仅在二分类数据集上进行调优,其生成能力局限于区分抗菌与非抗菌序列,无法进一步控制其他性质(如特定活性或靶向性)。在实际应用中,治疗性AMP往往需要同时满足多项要求,例如具有更高活性或针对特定细菌的选择性,因此需在AMP-Prompt基础上进一步优化。
借鉴ChatGPT中基于人类反馈强化学习(RLHF)的思想,AMP-Designer采用强化学习框架,通过专家知识或模型打分机制对生成序列进行评价并反馈,从而引导模型优化。然而,对AMPs进行人工打分极具挑战性,因为其活性受多种因素影响。AMP通常兼具一定程度的疏水性与亲水性,这种平衡影响其与细菌膜的相互作用;带正电荷的氨基酸残基可增强其与细菌膜中阴离子分子的结合,从而扰乱膜结构;此外,α-螺旋二级结构的形成也对AMP插入细胞膜并造成破坏起重要作用。
因此,在AMP-GPT的基础上,研究者针对不同细菌训练了AMP-MIC预测模型,综合考虑电荷等理化性质,为强化学习提供反馈(详见“高活性抗革兰氏阴性菌肽设计”部分)。但直接在基于GPT的AMP-Prompt上执行强化学习优化需要极高的计算成本。为此,研究者通过知识蒸馏(knowledge distillation)将AMP-Prompt压缩为更轻量的模型AMP-Distillation,该模型由三层门控循环单元(GRU)构成,既保持了与AMP-GPT相当的生成能力,又大幅降低了计算负担。随后,在AMP-Distillation上实施强化学习优化,从而高效筛选并确定最具潜力的候选抗菌肽以供后续合成与实验验证。
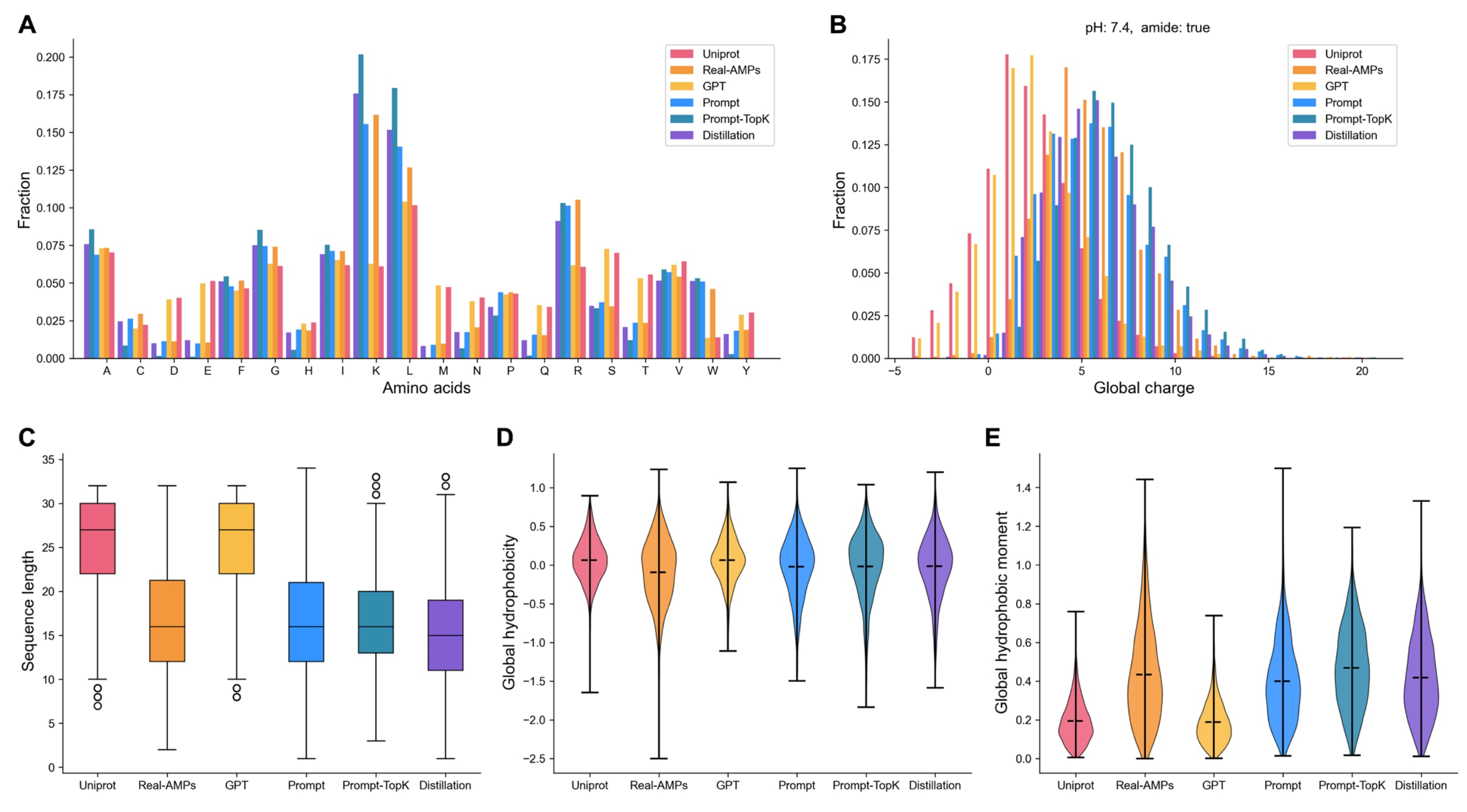
图2 | 展示了不同数据来源与模型生成肽序列在理化性质上的分布对比。 红色表示UniProt数据集,橙色表示真实抗菌肽(AMPs),黄色表示基础模型AMP-GPT,蓝色表示经对比提示微调后的AMP-Prompt,绿色表示采用top-k采样的AMP-Prompt-topK,紫色表示蒸馏模型AMP-Distillation。(A) 氨基酸组成分布;(B) 全局电荷(global charge)分布;(C) 序列长度(sequence length)分布;(D) Eisenberg疏水性(hydrophobicity)分布;(E) Eisenberg疏水矩(hydrophobic moment)分布。从结果可以看出,AMP-GPT生成序列的理化性质分布与UniProt数据集接近,说明模型成功学习了通用肽的统计特征;经对比提示学习与top-k采样后,AMP-Prompt与AMP-Prompt-topK生成的序列在电荷、疏水性与疏水矩等关键特征上更接近真实AMPs。蒸馏模型AMP-Distillation的分布与教师模型几乎一致,表明其充分继承了生成模型的理化属性分布与生成能力。
2.2 理化性质分析
为评估AMP-Designer的生成性能,研究者从生成模型与真实AMP数据集中各随机选取2000条肽序列进行分析。利用modlAMP工具对这些序列的氨基酸组成频率、总体电荷、Eisenberg疏水性、Eisenberg疏水矩及序列长度等理化属性进行了可视化统计。这些基础特征用于考察模型的学习动态,即基础模型是否有效捕捉到小肽序列空间的整体分布,以及后续微调是否成功学习到真实AMP的特征分布。
如图2所示,AMP-GPT生成的肽序列在理化性质上的分布与UniProt训练集高度一致,说明模型成功学习了训练集中肽的理化特征。经过对比提示学习(contrastive prompt learning)后,生成肽的多项理化性质分布发生明显变化,其属性更接近真实AMP数据,表明提示调优有效地引导模型捕获了抗菌肽的关键特征。
进一步采用top-k采样生成序列,结果显示,与传统的温度采样法相比,top-k采样生成的肽序列整体带有更高的正电荷,并在Eisenberg疏水性与疏水矩等特征上表现出更接近真实AMPs的分布特征。由于多数已报道的AMP呈阳离子性与两亲性,这些特征被认为对其插入并破坏细菌外膜至关重要,而外膜正是许多抗生素失效与耐药产生的关键因素。
最后,蒸馏模型生成的肽序列在理化性质分布上与教师模型几乎一致,表明AMP-Distillation成功学习了教师模型的概率分布,充分保留了生成性能与特征表达能力。

图3 | 展示了生成肽的活性概率分布及匹配得分分布。 (A–C) 为三种抗菌肽(AMP)预测器对生成序列的活性概率分布结果,分别基于CAMP、AMP Scanner与Macrel模型进行预测。结果显示,经对比提示调优与top-k采样生成的Prompt-TopK模型,其预测活性概率分布显著偏向高值区间,整体趋势更接近甚至优于真实AMPs。(D–E) 展示了匹配得分(match scores)的分布情况。(D) 为真实AMPs之间的匹配得分分布,(E) 为生成序列之间的匹配得分分布。两者对比表明,AMP-Designer生成的序列在序列多样性与相似性分布上与真实AMP集合高度一致,说明模型在生成过程中有效保持了序列空间的合理结构与生物多样性特征。
2.3 活性分析
为评估生成肽的生物活性,研究者采用了三种独立的抗菌肽分类器进行预测分析,分别为CAMP(Collection of Anti-Microbial Peptides)、AMP Scanner与Macrel。同时,引入两种当前最先进的对照方法——CLaSS与无细胞蛋白合成(CFPS)——作为比较基线。根据AMP Scanner的建议,长度少于10个氨基酸的序列预测结果可靠性较低,因此该研究仅选取长度不小于10个氨基酸的多肽序列进行评估。
图3(A–C)展示了基于提示调优模型在三种预测器上的显著性能提升。尤其是通过top-k采样生成的Prompt-TopK模型,其生成肽的活性分布最接近真实AMPs。在预测过程中,当分类器输出值≥0.5时,该序列被认为具有抗菌活性。结果显示,各模型生成的候选AMP在三种预测器上的表现与预期一致。AMP-GPT从UniProt学习的肽序列总体活性概率低于0.5,说明基础模型仅捕捉到一般肽特征而非抗菌特征。而经过提示调优(Prompt tuning)后,生成的候选AMP活性概率显著提高,与真实AMPs接近,尤其是Prompt-TopK模型的预测概率分布甚至超过真实AMPs,大多集中在接近1的高活性区域。
此外,与CLaSS和CFPS相比,AMP-Designer在三种预测器中均表现出更高的活性预测概率。为进一步量化结果,研究者计算了被三种预测器同时判定为活性AMP(预测值≥0.5)的序列比例。结果如表1所示,CLaSS为50.7%,CFPS为57.2%,而AMP-Designer方法的比例高达83.4%,相较CFPS提升26.2%,显示出显著优势。
研究结果还表明,Prompt-TopK生成肽的预测活性概率整体高于真实AMPs,说明模型在抗菌特征捕获方面具有更强的生成能力。同时,蒸馏模型AMP-Distillation在性能上与AMP-Prompt几乎一致(见表S1),结合前述理化性质分析结果可知,AMP-Distillation已成功继承并再现教师模型AMP-Prompt的概率分布与生物活性特征。
2.4 多样性与新颖性分析
为进一步评估AMP-Designer生成肽序列的有效性、多样性与新颖性,研究者将其与两种基线方法进行了比较:条件标记(Conditional Token, CT)模型与全模型微调(Full Fine-Tuning, FT)模型。CT方法通过在标注数据上重新训练模型,在训练与生成阶段均输入属性标签,以自回归方式生成完整肽序列;传统FT方法则直接在真实AMP数据集上对AMP-GPT模型进行全参数微调。相关细节见方法部分“CT与FT模型”章节。
其中,有效性(Validity)衡量生成序列是否为合法氨基酸序列;唯一性(Uniqueness)表示生成序列中不重复肽的比例;新颖性(Novelty)则指生成序列与真实AMPs不同的比例。分析结果显示,CT模型生成部分无效序列(如出现“[mask]”等标记),表明其未能充分学习AMP序列的氨基酸分布特征。表2的比较结果进一步揭示,FT生成序列的唯一性与新颖性均低于基于提示调优的方法。这表明FT模型存在较严重的过拟合倾向,生成结果中重复序列较多,且与真实AMPs相似度较高。这种现象的根本原因在于CT与FT方法在真实AMP数据上重新训练整个模型,易导致模型记忆原始数据分布。
相反,提示调优方法仅冻结预训练模型参数,并在真实AMP数据上训练提示嵌入层,从而在保持高抗菌活性概率的同时,显著提升了生成序列的多样性与新颖性。
为进一步定量分析生成序列的多样性,研究者使用biopython库中的pairwise2.align.globalxx方法进行序列比对,计算生成序列的匹配得分(match scores)。该方法通过两两比对生成的AMP序列与真实AMPs(或生成序列自身)以获得最高匹配得分。图3D显示了生成序列与真实AMPs之间的匹配得分分布,结果表明AMP-Designer生成的序列与真实AMPs相似度较低,明显优于对照模型;图3E则展示了不同模型生成序列间的内部相似度分布,AMP-Designer生成的序列内部相似度更低,说明模型具备更强的多样性。
综合来看,AMP-Designer不仅能够生成具有较高抗菌活性的肽序列,同时在多样性与新颖性上表现突出,成功平衡了模型生成的有效性与创新性,从而显著提升了新型抗菌肽的发现潜力。
表1 | 显示了由三种预测器(CAMP、AMP Scanner 和 Macrel)同时判定为抗菌肽(AMP)的序列比例。 结果表明,CLaSS模型的比例为50.7%,CFPS为57.2%,而AMP-Designer方法显著更高,达到83.4%,相较CFPS提升了26.2%,体现出其在生成高活性抗菌肽方面的显著优势。
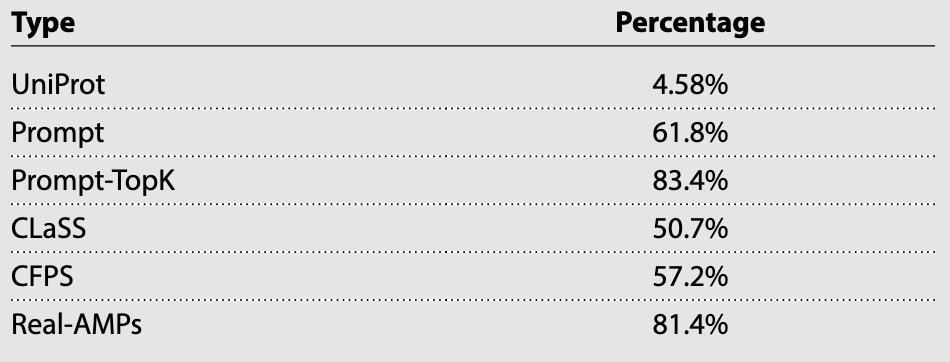
2.5 面向革兰氏阴性菌的高活性肽设计
上述方法虽能生成具生物活性的候选AMP,但对候选序列固有性质(如活性强度与净电荷)的可量化调控仍不充分。为获得更高活性及其他目标属性,引入强化学习进行进一步优化。强化学习的回报函数综合了5个部分:分类概率(CP)、针对三种菌株的MIC、序列长度(SL)、记忆相似度(MS)与电荷,细节见表S4。CP由SOTA预测器Macrel给出;三种菌的MIC由基于AMP-GPT训练的AMP-MIC模型预测(涵盖金黄色葡萄球菌、铜绿假单胞菌与大肠杆菌)。与Huang等提出的RNN及自零开始训练的GPT相比,AMP-MIC在表S5中表现出更高准确度。为提升多样性,未直接将连续数值的CP与MIC作为强化学习回报。为量化RL贡献,在调参过程中逐步采样并统计平均得分,分析RL步数与生成质量的关系。图S3(A–C)显示,随RL步数增加,AMP预测器的平均活性概率由0.5提升至>0.7;对大肠杆菌与铜绿假单胞菌的预测MIC由>500降至<5。进一步在第10、30与50步可视化分布,图S3D表明AMP预测得分逐步提高;对两种细菌的活性分布在图S3(E、F)中愈发集中,多数值低于5(为便于展示,将>20截断为20)。
为兼顾质量与多样性,设置记忆队列保存得分最高的256条AMP;RL过程中每次采样128条序列评估当前状态,将高分序列加入队列并替换低分者。记忆相似度以biopython的pairwise2.align.globalxx比对所得匹配分数除以序列长度计算。综合上述因子的最终回报见
表2 | 展示了五种模型在生成序列的有效性(Validity)、唯一性(Uniqueness)与新颖性(Novelty)方面的比较结果。 传统的条件标记(CT)方法生成了部分无效序列(如出现“[mask]”等符号),说明其对氨基酸分布的学习不足;全模型微调(FT)虽然能生成有效序列,但其唯一性与新颖性均低于基于提示调优的方法,表明其存在明显的过拟合现象。而AMP-Designer的提示调优方法在保持高活性概率的同时,实现了更高的序列多样性与创新性,生成的肽序列既合法又具有独特性与原创性,性能优于CT与FT模型。

2.6 候选AMP对ESKAPE成员的体外抗菌活性
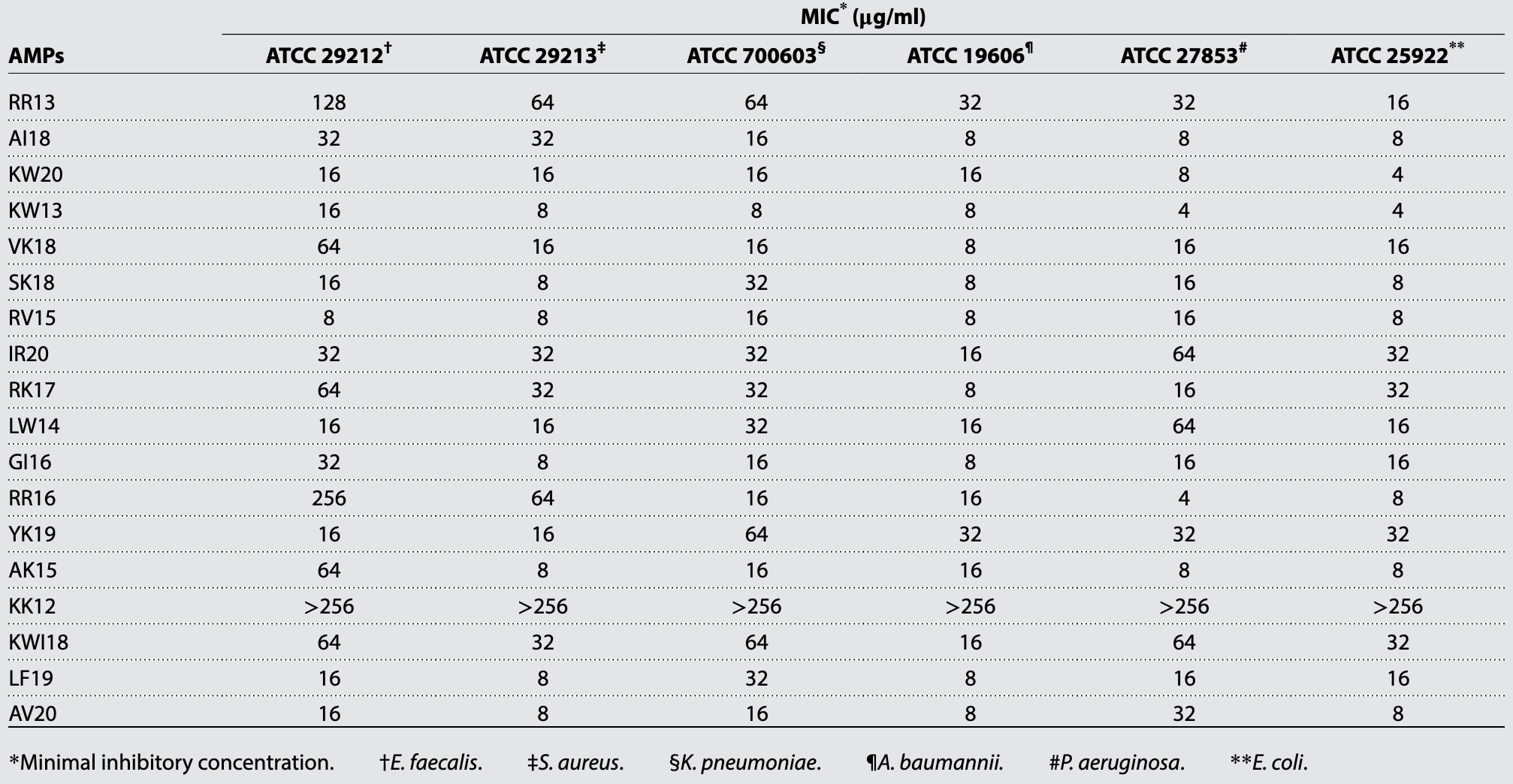
研究者从强化学习与筛选阶段选出的前20个预测AMP中进行了化学合成与验证,其中2条肽在三次合成尝试后仍未成功。最终获得18条候选肽,并测试其对ESKAPE六种标准菌株的体外抗菌活性,包括肠球菌(Enterococcus faecalis ATCC 29212)、金黄色葡萄球菌(Staphylococcus aureus ATCC 29213)、肺炎克雷伯菌(Klebsiella pneumoniae ATCC 700603)、鲍曼不动杆菌(Acinetobacter baumannii ATCC 19606)、铜绿假单胞菌(Pseudomonas aeruginosa ATCC 27853)及大肠杆菌(Escherichia coli ATCC 25922)。如表3所示,18条候选肽中有17条对至少一种菌株表现出显著抗菌活性。总体来看,大多数AMP对革兰氏阴性菌的抑菌效果优于对阳性菌的作用,这与模型针对阴性菌的定向设计目标一致。此外,这些原本为P. aeruginosa与E. coli设计的AMP对K. pneumoniae与A. baumannii同样具有显著活性,提示其可能具有普适的抗革兰氏阴性菌杀菌机制。
其中AI18、KW13、KW20、RV15与GI16表现出最优广谱抗菌性能,对阴性菌的MIC值介于4–16 μg/ml,对阳性菌为8–32 μg/ml。随后评估了这5条肽对临床耐药阴性菌株的抑菌效果,包括全耐药(PDR) K. pneumoniae 418015、广耐药(XDR) K. pneumoniae 325016、多重耐药(MDR) K. pneumoniae 327004、MDR E. coli 103231、MDR P. aeruginosa 304238与MDR A. baumannii 316039(结果见表4)。五条AMP均对五种“超级细菌”表现出显著活性,MIC为4–16 μg/ml;但KW13与AI18对PDR型K. pneumoniae 418015的活性略低(该菌为黏菌素耐药株)。进一步结合文献对比,研究团队使用LENS.ORG平台的patseqfinder工具分析AMP序列的多样性与专利序列相似度(结果见表S7),结果显示该研究所设计的AI肽在MIC、序列创新性等多项指标上均优于以往人工智能方法。
表3 | 前18条候选抗菌肽(AMP)对六种标准ESKAPE菌株的体外抗菌活性。
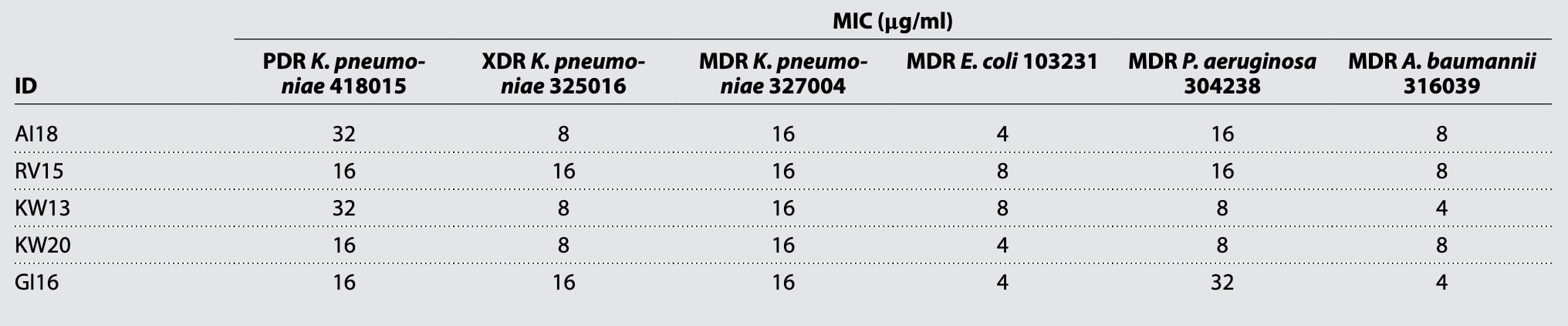
针对阳离子AMP常见的非靶向毒性(尤其溶血性),评估了AI18、KW13、KW20、RV15与GI16对人红细胞的溶血作用。如图4A所示,KW13与AI18几乎不引起溶血。进一步测定其半溶血浓度(HC50)均高于1000 μg/ml(见图S4),定量证明其血液相容性优异。为更全面评估其杀菌动力学,研究者对E. coli ATCC 25922进行时间杀灭实验(图4B)。结果显示,KW13与AI18在2小时内即可显著降低菌落存活量,杀菌效应可持续至少24小时;相比之下,黏菌素仅维持约8小时。
由于AMP主要通过破坏细菌膜结构发挥作用,其耐药性诱导潜能较低。研究者在亚MIC水平下连续传代培养E. coli ATCC 25922,比较KW13、AI18与小分子抗生素诺氟沙星(norfloxacin)的耐药诱导情况。结果如图4C所示,E. coli在接触KW13与AI18后30代内未出现明显耐药性变化,与黏菌素表现一致;而对诺氟沙星在第17代后即出现耐药性,并在30代时敏感性下降128倍。
进一步分析了KW13与AI18在血浆中的稳定性。4小时人血浆降解实验(图4D)显示二者降解缓慢,表明其具有良好的血浆稳定性。随后选取体外活性最强的KW13,对其膜破坏机制进行研究。AMP的常见作用机制为通过形成膜孔导致细菌裂解。采用透射电子显微镜(TEM)观察KW13处理后的E. coli ATCC 25922细胞形态(浓度分别为1×与10× MIC,作用5小时)。如图4E所示,细胞内容物外泄并发生裂解,且呈剂量依赖关系,说明其可有效破坏细胞膜完整性。
综上,KW13与AI18在体外表现出优异的杀菌活性、低溶血性与高血浆稳定性,通过初步安全性筛查,具备进一步体内治疗潜力,因此进入随后的动物模型验证阶段。
表4 | 四种筛选出的抗菌肽(AMP)对六种临床耐药革兰氏阴性菌株的体外抗菌活性。
2.7 KW13与AI18在小鼠肺炎模型中的体内抗菌活性
研究者进一步在由广耐药(XDR)Klebsiella pneumoniae 325016感染构建的小鼠肺炎模型中验证KW13与AI18的治疗效果。由碳青霉烯耐药K. pneumoniae引起的医院获得性肺炎是临床严重问题,治疗困难且死亡率高。抗菌肽(AMP)被认为是治疗肺部感染的潜在抗生素替代方案。
实验设计如图5A所示。首先使用环磷酰胺抑制小鼠免疫系统,再经气管内接种K. pneumoniae 325016构建肺炎模型。随后给予两种剂量的AMP治疗,处理结束后取肺组织测定细菌负荷。如图5B所示,两组AMP处理小鼠的肺部细菌负荷减少约99%,显著低于空白对照组(PBS处理),其治疗效果与阳性对照抗菌肽indolicidin相当。组织病理切片分析显示,接受KW13、AI18或indolicidin治疗的小鼠肺组织结构完整,肺泡形态清晰,而PBS组则出现严重的组织损伤和炎症反应(图5C)。
上述结果表明,KW13与AI18在体内对耐药性K. pneumoniae感染性肺炎具有显著的抗菌效果,且未观察到明显的不良反应,显示出良好的治疗潜力和进一步开发的价值。
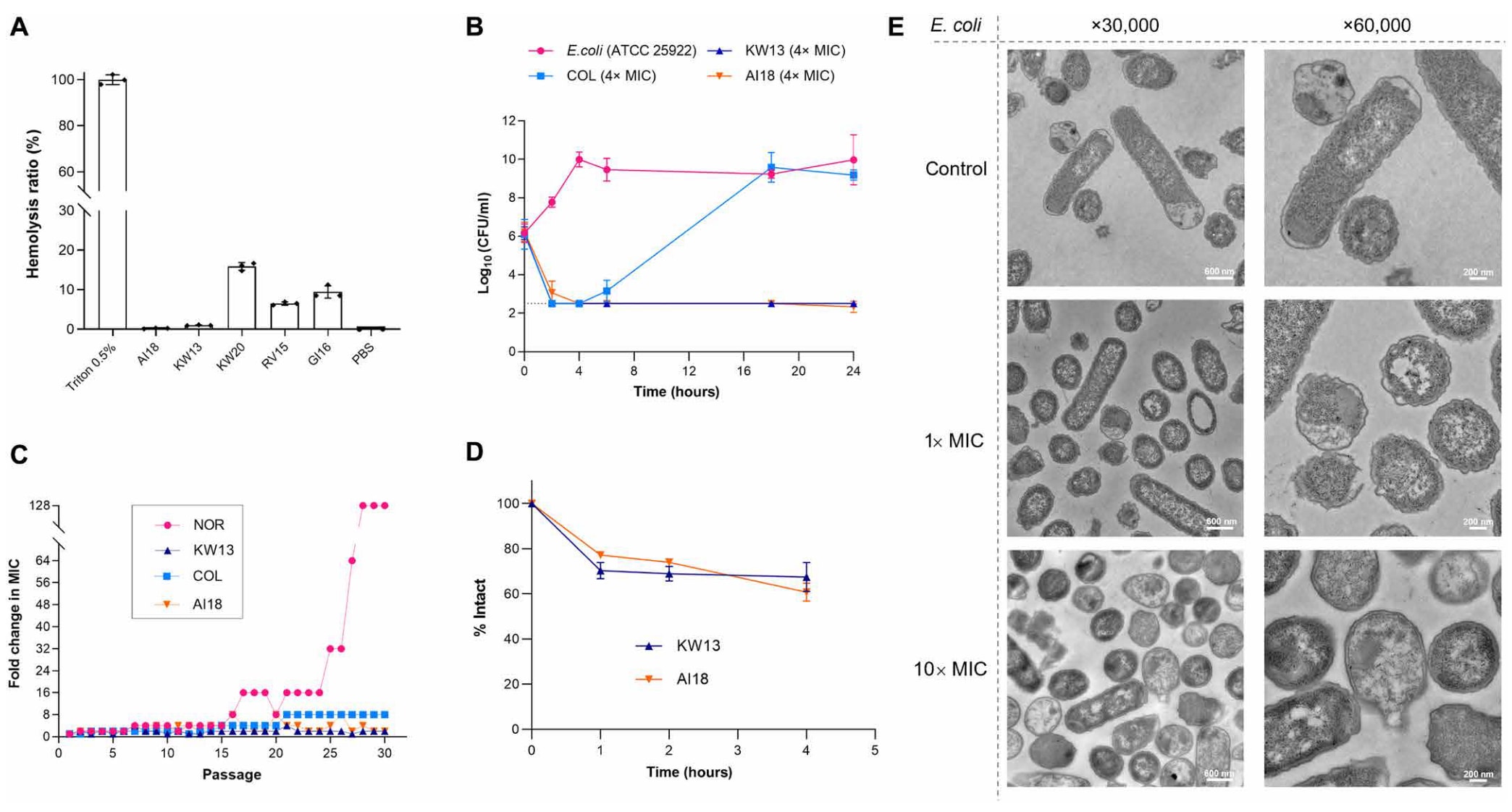
图4 | 展示了筛选出的抗菌肽(AMP)在体外的生物学性质。 (A) KW13、AI18、KW20、RV15与GI16在100 μg/ml浓度下的人红细胞溶血活性测试结果(n = 3个生物学独立重复,数据以平均值 ± 标准差表示)。(B) KW13与AI18的时间杀菌曲线,以黏菌素(COL)作为阳性对照(n = 3个生物学独立重复,平均值 ± 标准差)。(C) E. coli ATCC 25922在亚MIC浓度(1/2×)下与KW13、AI18共培养的耐药性诱导实验结果,以诺氟沙星(NOR)与黏菌素(COL)为阳性对照。(D) KW13与AI18的人血浆稳定性测试结果,采用高效液相色谱(HPLC)分析,数据以平均值 ± 标准差表示(n = 3个生物学独立重复)。(E) E. coli细胞经KW13与PBS(对照)处理后的透射电子显微镜(TEM)图像。放大倍数分别为×30,000(比例尺600 nm)与×60,000(比例尺200 nm)。
2.8 针对P. acnes的少样本抗菌肽设计
寻常痤疮(Acne vulgaris)是一种慢性皮肤疾病,全球患者超过6.3亿例,被列为全球第八常见疾病。厌氧性革兰氏阳性菌P. acnes在皮脂腺毛囊中的定植与痤疮严重程度密切相关。传统治疗多依赖局部或全身应用的小分子抗生素,但近年来抗药性问题日益严重。此任务的挑战在于针对P. acnes的标注数据极为稀缺。通过分析整个微调数据集,仅发现47条带有明确抗P. acnes活性标注的AMP,其中17条具有C端酰胺化修饰且序列长度≤32个氨基酸。分析结果表明,对P. acnes的MIC值与其对S. aureus、S. epidermidis及P. aeruginosa的抗菌活性呈显著正相关。基于此,研究利用这三种菌的活性预测器作为代理模型,推测其对P. acnes的抑菌能力。设计流程与“面向革兰氏阴性菌的高活性肽设计”部分基本一致,最终筛选出五条候选AMP进行体外实验验证。
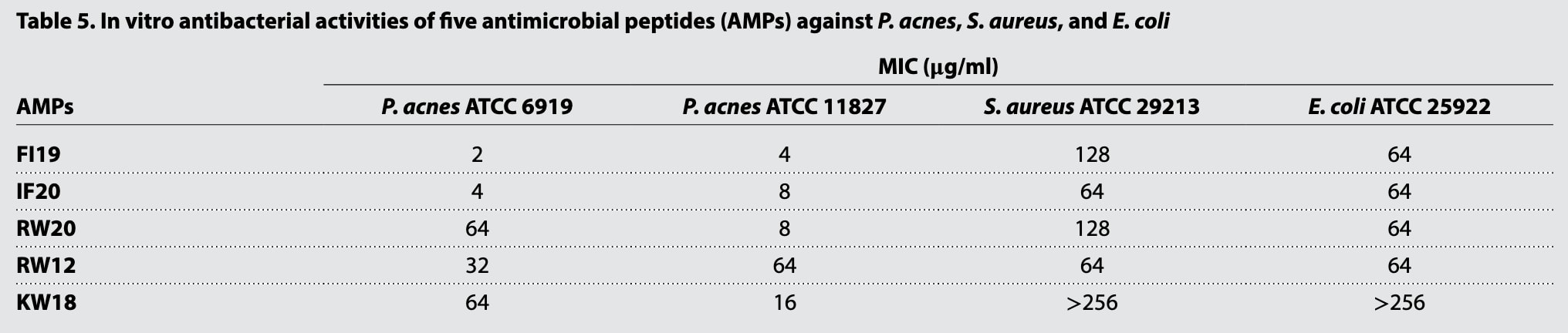
如表5所示,其中三条肽对P. acnes表现出极强活性,MIC介于2–8 μg/ml,明显低于其对阳性菌S. aureus与阴性菌E. coli的MIC值,体现出对P. acnes的选择性靶向特征。其中FI19的活性最强,MIC为2.0 μg/ml。性质–活性相关性分析显示,较高的疏水性与较低的净电荷可能有助于增强抗菌能力。FI19、IF20与RW20作为专门针对P. acnes的高效窄谱抗菌肽,具备进一步改造与开发为系统性抗痤疮药物的潜力。
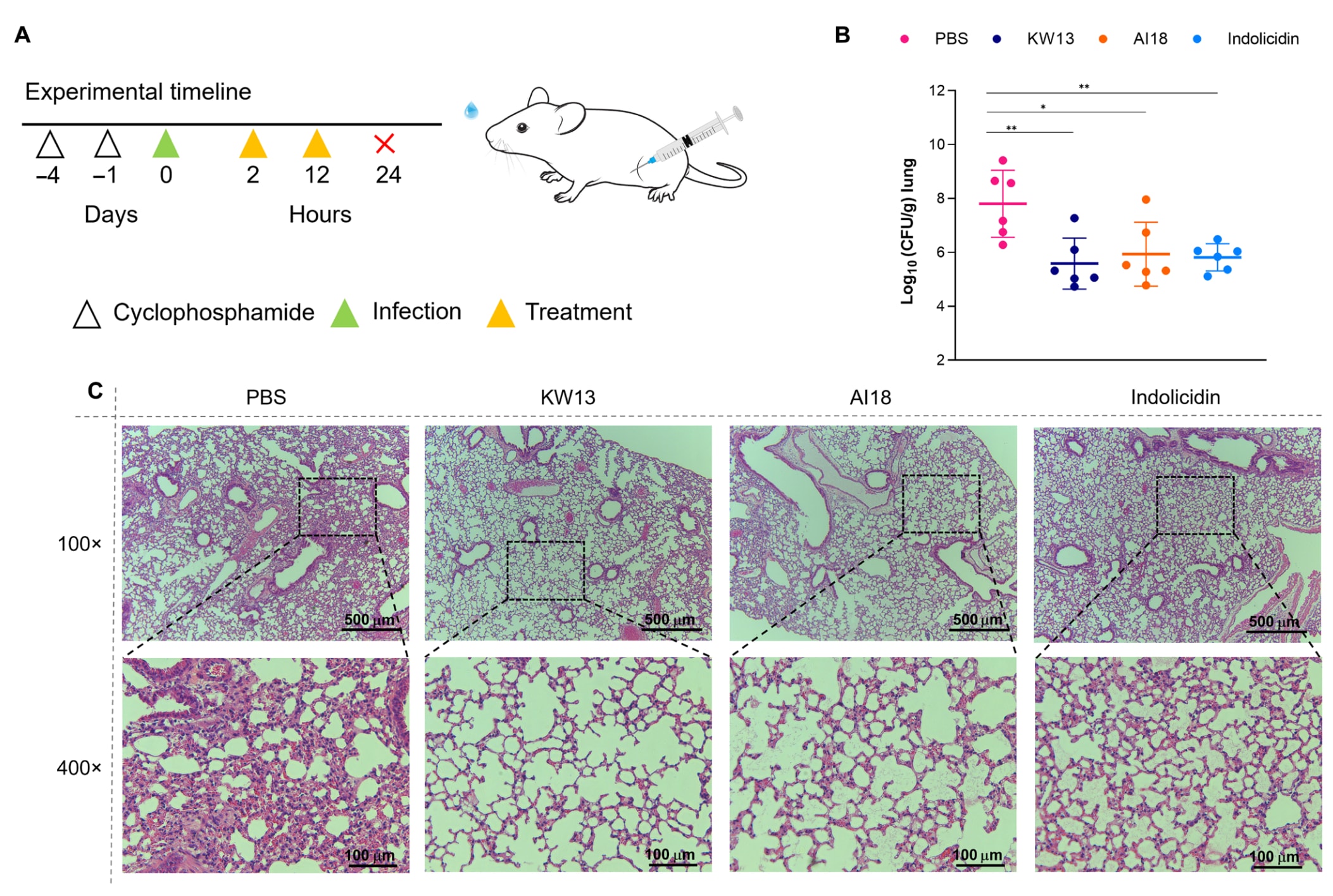
图5 | 展示了KW13与AI18在小鼠肺炎模型中的体内治疗效果。 (A) 小鼠感染模型的构建流程。小鼠先腹腔注射环磷酰胺两次以抑制免疫系统,随后经气管滴注广耐药(XDR)Klebsiella pneumoniae 325016以建立肺炎模型。抗菌肽(AMPs,10 mg/kg)经腹腔注射两次,在感染后24小时处死小鼠取肺组织进行分析。(B) 经PBS、KW13、AI18与indolicidin处理后感染小鼠肺部的细菌负荷结果(n = 6个生物学独立重复,数据以平均值 ± 标准差表示)。统计分析采用单因素方差分析(ANOVA),*P < 0.05,**P < 0.01。(C) 肺组织切片的组织学图像。第一行显示经苏木精-伊红(H&E)染色后的肺组织整体形态(比例尺500 μm);第二行显示黑框区域的放大组织学细节图(比例尺100 μm)。所有实验均重复两次并得到一致结果,图中展示一组代表性结果。
表5 | 五种抗菌肽(AMP)对P. acnes、S. aureus和E. coli的体外抗菌活性。
3 讨论
肽类设计是一项极具挑战性的任务。与小分子不同,肽由氨基酸序列组成,结构柔性高,使得基于专家经验的设计难度极大。近年来,人工智能(AI)的快速发展使得AI驱动的药物与材料设计方法不断涌现。鉴于肽序列与自然语言在模式学习上的相似性,基于GPT等大型语言模型的策略被认为可加速肽类发现进程。
该研究提出了一个以抗菌肽(AMP)为核心的基础语言模型及创新设计框架——AMP-Designer。该框架构建了首个大规模AMP预训练模型AMP-GPT,并结合提示调优、对比学习、知识蒸馏与强化学习等技术,实现了从模型训练、虚拟筛选到实验验证的闭环体系。体外实验确认了所生成AMP的广谱抗菌性能,其中活性最高的前五条肽对多种临床相关的革兰氏阴性菌表现出强效抗菌活性。KW13与AI18作为领先候选者,在小鼠肺炎模型中展现出与阳性对照indolicidin相当的治疗效果,同时具备极低溶血性与耐药诱导倾向,凸显了AMP-Designer在应对全球耐药性(AMR)危机与抗生素研发瓶颈方面的潜力。KW13与AI18可作为后续结构优化的起点,通过肽环化、主链锁定或脂肪酸修饰等手段进一步提高效力与血浆稳定性。此外,其与传统抗生素联合应用的研究同样具有重要意义。
进一步地,该研究还展示了少样本(few-shot)AMP设计能力。利用P. acnes与S. aureus活性相关性,AMP-Designer成功生成了对P. acnes具有极高体外活性的AMP。尽管用于训练的P. acnes特异标注数据不足20条,整体真实AMP数据集约1万条,且未专门针对P. acnes进行微调或强化学习优化,模型仍能生成高活性AMP,表明其基于肽语言模型的设计框架已成功捕获AMP空间的分布特征,并能在数据稀缺情境下生成具有潜在药效的新序列。
展望未来,结合实验验证与深度学习的跨模态研究将发挥更大作用。传统实验依赖“试错—优化”过程,往往造成资源浪费;而深度学习可高效指导实验方向并生成新数据反哺算法性能,形成良性循环,加速AMP发现与优化。
此外,将大型语言模型与优化策略结合,可进一步扩展多功能肽(multiplexed peptide)设计。AMP-Designer基于通用AMP-GPT模型,具备良好的可扩展性,可无缝迁移至其他下游肽类设计任务。整个项目从计算设计到体内验证共耗时48天,其中计算阶段仅用11天。若基于已有AMP-GPT模型进行迁移学习,可将设计周期缩短至3天内,从而快速生成具有免疫调节、细胞运输、信号传导或代谢功能的多类型功能肽。