NC 2025 | DILI: 用于药物性肝损伤预测的大规模人类毒理基因组学资源
今天介绍的是发表于 《Nature Communications》 的一项重要研究,聚焦于药物性肝损伤预测这一长期困扰药物研发的关键难题。研究者构建了DILImap,这是目前规模最大的面向肝毒性研究的人类毒理基因组学资源,基于300种化合物在原代人肝细胞中的RNA-seq数据,系统覆盖多类肝损伤机制。在此基础上开发的机器学习模型ToxPredictor不仅能够进行剂量分辨的肝毒性预测,还可计算安全边界,用于指导早期去风险决策。在独立盲测中,该模型实现88%敏感性与100%特异性,并成功识别多项被动物实验忽略、但在Ⅲ期失败的药物,如Evobrutinib与TAK-875。此外,转录组学提供的系统层面信息使模型能够捕捉线粒体损伤、氧化应激、免疫激活等传统检测难以发现的机制。该研究展示了毒理基因组学在提升药物安全性评估中的巨大潜力,为未来AI/ML驱动的药物发现提供了重要方向。

获取详情及资源:
- 📄 论文: https://doi.org/10.1038/s41467-025-65690-3
- 💻 代码: https://www.github.com/Cellarity/DILImap_reproducibility
0 摘要
药物性肝损伤是药物研发中最关键的挑战之一,常导致患者安全风险、临床试验失败以及药物退市。研究者提出ToxPredictor,这是一种将原代人肝细胞的RNA-seq数据与药代动力学数据结合起来的毒理基因组学框架,能够预测不同剂量下的肝损伤风险与安全边界。其中的核心资源是DILImap,这是一套专为肝损伤研究构建的RNA-seq库,涵盖300种化合物及多种浓度设定。在盲测验证中,ToxPredictor在保持100%特异性的条件下达到88%的敏感性,性能优于现有方法。该框架成功识别出近期多项在动物实验中未被察觉但在Ⅲ期临床失败的药物,包括Evobrutinib、TAK-875和BMS-986142。除预测能力之外,ToxPredictor还能提供肝毒性通路的机制性洞见,为早期风险控制与安全决策提供依据。与依赖单一终点读数的传统检测方式不同,即使是三维模型,也难以达到转录组学在系统层面呈现肝细胞多维响应的能力,从而能够捕捉常规检测未覆盖的多种肝损伤机制。作为可拓展、可应用并可整合进更广泛AI/ML药物发现平台的工具,这项工作展示了毒理基因组学在开发更安全治疗方案、应对毒理学重大难题方面的潜力。
1 引言
药物性肝损伤在药物研发后期仍是一项理解不足的关键难题,平均每家制药公司每年因此损失约3.5亿美元。其在临床人群中的发生率极低,往往低于万分之一,使得临床试验难以捕捉风险,真实危害常在上市后才逐渐显现。动物模型大约会漏掉一半在临床中表现出肝毒性的药物,使其成为候选药物失败与退市的主要原因之一。肝损伤由复杂且多因素驱动的机制引发,包括剂量依赖的内在毒性以及受遗传背景、环境与健康状态影响的难以预测的特异性反应。相关细胞损伤形式涉及线粒体功能障碍、氧化应激、胆汁酸失衡、特定酶或转运蛋白的抑制以及反应性代谢物的形成。然而,具体的致损机制仍未完全解析,缺乏用于早期检测的生物标志物进一步限制了风险识别。现有的定量构效关系模型由于特异性较低且只能提供二元预测,难以揭示机制层面的信息。体外模型虽然来源多样,终点指标却多为LDH、ATP等低维读数,即使三维肝模型改善了生理相关性,其可测信号仍不足以覆盖肝损伤的分子全貌,常导致机制性原因被遗漏,最终仍造成后期退市与临床失败。
受“将细胞视作复杂系统”这一理念启发,研究者采用机器学习驱动的毒理基因组学策略,通过分析药物对基因表达的影响来寻找肝损伤的早期特征,从而更系统地解析其分子机制。基于DILIrank与LiverTox的药物分类,研究者首先利用TG-GATES微阵列数据库验证可行性,在盲测中获得62%敏感性与92%特异性。基于这一初步成果,研究者构建了DILImap,这是一套规模更大且专为肝损伤研究设计的转录组资源,包含300种化合物在不同浓度下的原代人肝细胞RNA-seq数据,成为目前规模最大的肝损伤毒理基因组学数据集。在此基础上训练的ToxPredictor模型在盲测中表现出88%敏感性与100%特异性,优于二十余种现有前临床模型,包括多类机制检测、细胞毒性标志物、理化性质分析、生物活化、BSEP相关检测以及最新的计算模型。值得注意的是,这是首个能够预警Evobrutinib、TAK-875与BMS-986142等Ⅲ期临床失败药物肝损伤风险的前临床模型,这些药物在传统模型中均显示出良好安全性。
ToxPredictor不仅能准确预测未知化合物,还在机制广度上具备显著优势。其利用全转录组信息识别多类肝毒性机制,如线粒体损伤、氧化应激、免疫激活及代谢紊乱,且通常能在细胞死亡前捕捉到早期信号。与依赖低维终点的三维肝模型相比,该方法能够识别后者难以检测的非细胞毒性风险,从而在不同化合物类型中实现更全面与更公平的毒性识别。作为更广泛AI/ML药物发现平台的重要组成部分,这项工作展示了从单一指标向系统层面转变的价值,并凸显机器学习在毒理基因组学中的潜力。研究者已将模型与验证数据开源至dilimap.org,为推动领域发展提供了强有力的工具,并为安全性评估的范式转变奠定基础。
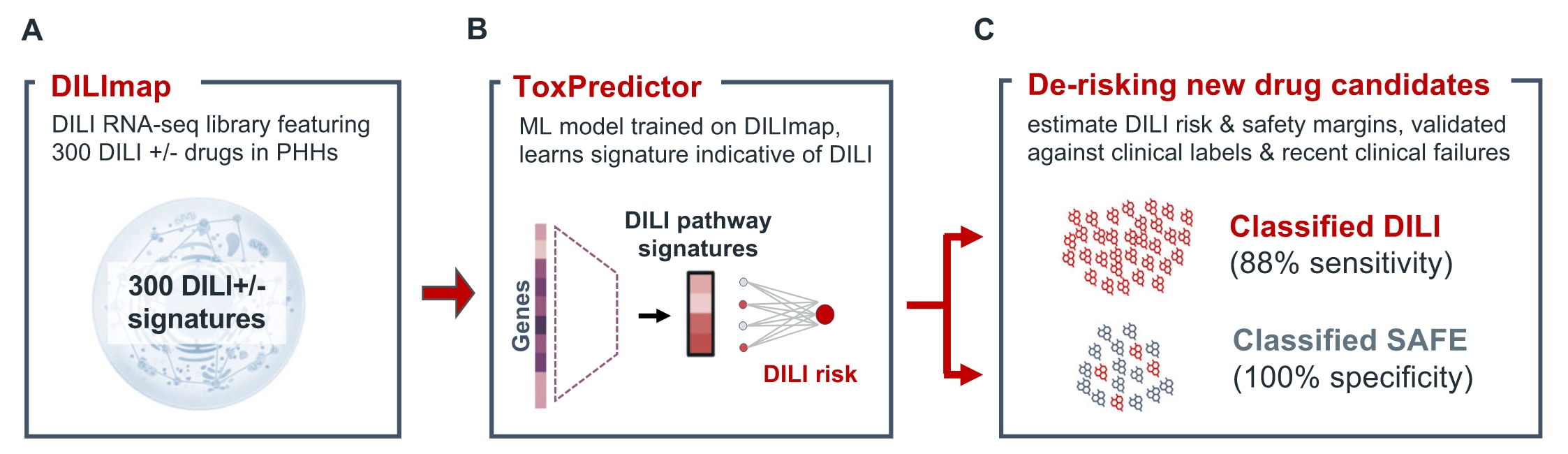
Fig. 1 | DILImap支持对药物性肝损伤的精准预测。 A 构建的DILImap是目前规模最大的毒理基因组学数据集,涵盖300种化合物在不同浓度下处理原代人肝细胞所得的RNA-seq表达谱,化合物类型覆盖多种肝损伤机制、特异性反应以及非肝毒性对照,从而确保机制层面的广泛覆盖,并为模型训练提供基础。B 基于DILImap训练的机器学习模型ToxPredictor能够学习与肝损伤风险相关的通路特征。C ToxPredictor可通过估计肝毒性风险与安全边界来辅助候选药物的早期去风险化。在盲测验证中,该模型实现88%敏感性(成功识别29/33肝毒性药物)与100%特异性(准确分类全部14种无肝毒性药物),为安全性评估带来潜在的范式转变。
2 结果
2.1 DILImap——用于肝损伤建模的人类毒理基因组学数据库
研究者构建了DILImap,这是一套专为药物性肝损伤建模而设计的全面RNA-seq资源,涵盖300种化合物在四个浓度下的转录组数据。作为目前规模最大的毒理基因组学数据集,DILImap收录的化合物既包括明确的肝毒性药物,也包括缺乏典型特征的特异性化合物,从而覆盖已知的多种肝损伤机制。所有化合物均在三明治培养的原代人肝细胞中检测,这一体系在体外模型中最具生理相关性,能长期维持肝细胞代谢活性与胆小管结构。每种化合物以六个浓度进行LDH与ATP活性检测,并在四个剂量下进行RNA-seq测序,覆盖从临床Cmax到略低于IC10阈值的非细胞毒性剂量区间。测序时间选择在处理后24小时,以在信号强度与细胞状态之间取得平衡,既避免过早时间点信号不足,也防止过长培养导致去分化与RNA降解。标志物分析确认24小时样本仍保持肝细胞身份,同时仅保留总RNA量充足且线粒体RNA比例较低的样本,以确保数据质量。
通过自动化溶解度检测、活性筛查与基于IC10的剂量选择,这套流程在四个月内完成300种化合物的系统性测序,其中包括110种首次在前临床体系中测试的药物。为支持全面基准分析,研究者为每种化合物提供详尽注释,包括临床肝损伤标签、机制分类、分子信息、来源于多项研究的共识Cmax以及来自二十余项前临床研究的肝毒性评估结果。化合物依据DILIrank与LiverTox划分为多类:因肝损伤而撤市或试验失败的Withdrawn DILI、具有明确风险的Known DILI、具备病例证据的Likely DILI、罕见且缺乏剂量依赖性的Idiosyncratic DILI、证据不足的Unlikely DILI以及无肝毒性记录的No DILI。Withdrawn、Known与Likely DILI作为阳性对照,No DILI作为阴性对照,而Idiosyncratic与Unlikely DILI因标签不确定性不参与训练,用于后续评估。
训练数据集中包含249种化合物(111种DILI阳性、52种阴性、17种不太可能DILI、69种特异性DILI),并用于交叉验证。盲测验证则基于一套独立的51种化合物(33种DILI阳性、14种阴性及4种标签未知药物,其中包括真实临床失败案例)。这一精心整理的数据集为后续构建预测模型与解析肝损伤机制提供了坚实基础。
2.2 ToxPredictor——基于通路层面的毒理基因组学模型用于预测肝损伤风险与治疗安全边界
ToxPredictor是在DILImap基础上训练的机器学习模型,通过通路层面的转录组特征来预测药物性肝损伤风险。相关特征来源于利用DESeq2对化合物处理组与DMSO对照组进行差异表达分析,并基于WikiPathways的富集分析结果(FDR校正p值)提取的通路信号,每种化合物的每个剂量均单独计算。在模型训练阶段,仅选用DILI标签明确的化合物,从而构建由111种DILI阳性(Withdrawn、Known、Likely)与52种DILI阴性(No DILI)构成的高可信训练集,其余数据用于测试模型稳健性。为了避免将实际有肝损伤风险但低剂量下未表现毒性的化合物错误标记为阴性,训练时进一步限制剂量需超过临床Cmax的20倍以上。
交叉验证采用分层的化合物级拆分方式,确保同一化合物的所有剂量与重复均在同一折中被整体保留,以模拟模型在完全未知化合物上的泛化能力。研究者在八类模型的193个配置中进行比较,最终选择随机森林模型,其验证AUC高、过拟合最小且跨折表现稳定。基于其可解释性与稳定性,研究者进一步构建包含30个随机森林成员的集成模型,以提升最终预测的鲁棒性。
ToxPredictor可在不同剂量下输出肝损伤概率,据此计算安全边界,即首次预测DILI的剂量(预测概率>0.7的最低剂量)与临床治疗水平Cmax的比值。该比值可视作一种基于转录组的临床治疗窗替代指标。安全边界的阈值设为80,以区分高风险与低风险化合物。概率阈值0.7与安全边界80均在独立留出的训练数据上优化确定,旨在在性能趋于平台期时尽量减少假阳性。由于转录组变化可在远低于细胞毒性水平的剂量下出现,该方法相较于细胞毒性终点更敏感,因此需要更高的阈值才能保持特异性。
在暴露量衡量中,研究者采用总Cmax而非游离Cmax,因为其跨化合物覆盖度更高,且预测表现略优,可能源于更多研究提供的更一致的共识值。所有模型选择、超参数调优与阈值设定均仅基于训练数据完成。
最终评价使用一套完全独立的51种化合物作为盲测集(33种DILI阳性、14种阴性以及4种标签未知化合物),这些化合物在独立实验(不同板面、不同测序批次)中测试。盲测集的选择在训练开始前即已敲定,并特意富集因肝毒性撤市或临床失败的药物。四种未知化合物均为当前临床使用或试验中的药物,尚无明确肝毒性结论。
在盲测验证中,模型的敏感性达88%,准确识别33种DILI阳性中的29种;特异性达100%,全部14种DILI阴性化合物均被正确判定为安全。
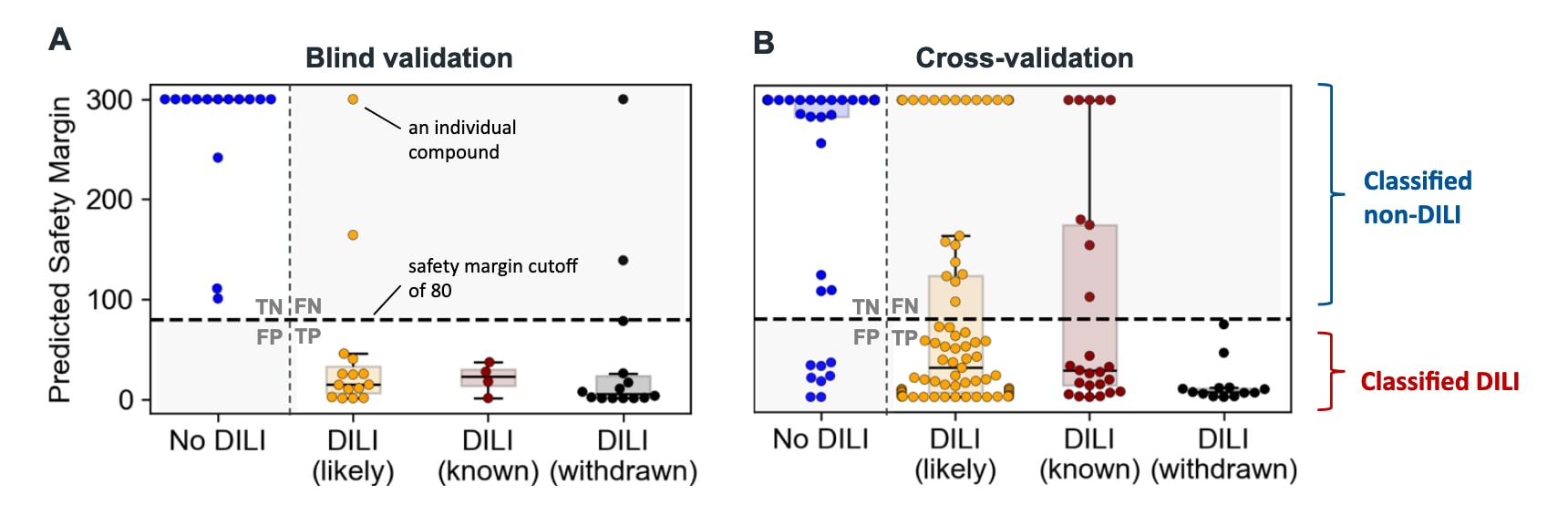
Fig. 2 | 基于转录组的安全边界能够准确识别肝损伤风险,包括已撤市药物。 A 化合物按照潜在肝毒性分为四类:无肝毒性(No DILI, n=14),可能肝毒性(likely DILI, n=15),已知肝毒性(known DILI, n=4),以及因肝毒性撤市的药物(withdrawn, n=14)。图中每个点表示单个化合物的安全边界;箱线图展示中位数、四分位范围及1.5倍四分位距以内的数据。安全边界以首次出现毒性剂量与治疗水平Cmax的比值计算,在盲测验证中利用阈值80(虚线)能够有效区分肝毒性阳性与阴性化合物。模型最终实现88%的敏感性与100%的特异性。B 对整个DILImap数据集进行交叉验证进一步确认模型在更大范围内的稳健性(No DILI: n=52;likely DILI: n=73;known DILI: n=25;withdrawn: n=13)。这些结果表明,基于转录组的建模方法在肝损伤检测方面优于传统策略,不仅能更敏感地识别撤市药物与已知高风险药物,同时保持对无肝毒性对照的高特异性。
2.3 识别先前在动物与临床研究中被忽略的撤市药物与特异性肝损伤
这些结果相较于基于TG-GATES微阵列数据的初始概念验证模型有显著提升。早期模型的敏感性与特异性分别为62%与92%,而借助DILImap,ToxPredictor在同一验证集上将表现提高至88%敏感性与100%特异性(Fig. 2A)。在对整个数据集的交叉验证中,模型成功识别144种肝毒性化合物中的110种,远超此前TG-GATES模型的62种,同时仅误判66种无肝毒性化合物中的8种(Fig. 2B)。这一显著进步得益于DILImap更大规模且机制覆盖更广的数据基础,以及RNA-seq相比微阵列更高的解析度,使其能够检测更多基因、并在表达变化的定量范围上具有更高灵敏性。
值得强调的是,那些上市后因肝损伤而撤市、且在前临床模型与临床试验中均未被识别出的药物,是模型最有信心预测为高风险的一类。ToxPredictor的肝毒性预测依据三个关键参数:血浆Cmax(基线暴露水平)、细胞活性检测(反映细胞死亡)以及基于差异通路的转录组特征。为了评估各参数的贡献,研究者单独测试了它们对肝损伤分类的能力。在144种肝毒性药物中,有29种可仅凭Cmax(>25 μM)检测到,另有42种可通过LDH细胞毒性指标(安全边界<80)检测到,二者的特异性均≥90%。而当将转录组学、Cmax与LDH结合时,检测能力最强,共识别到142种中的110种肝毒性化合物,说明毒理基因组学能够捕捉到超越细胞死亡的更早期信号(Supplementary Fig. S8)。
在交叉验证中,ToxPredictor的ROC AUC为0.82,而单独依赖细胞活性仅为0.66;在盲测验证中,ToxPredictor达到0.96,而活性指标仅为0.65(Supplementary Fig. S9)。这些结果强调了转录组学模型在早期肝损伤检测中的独特价值。
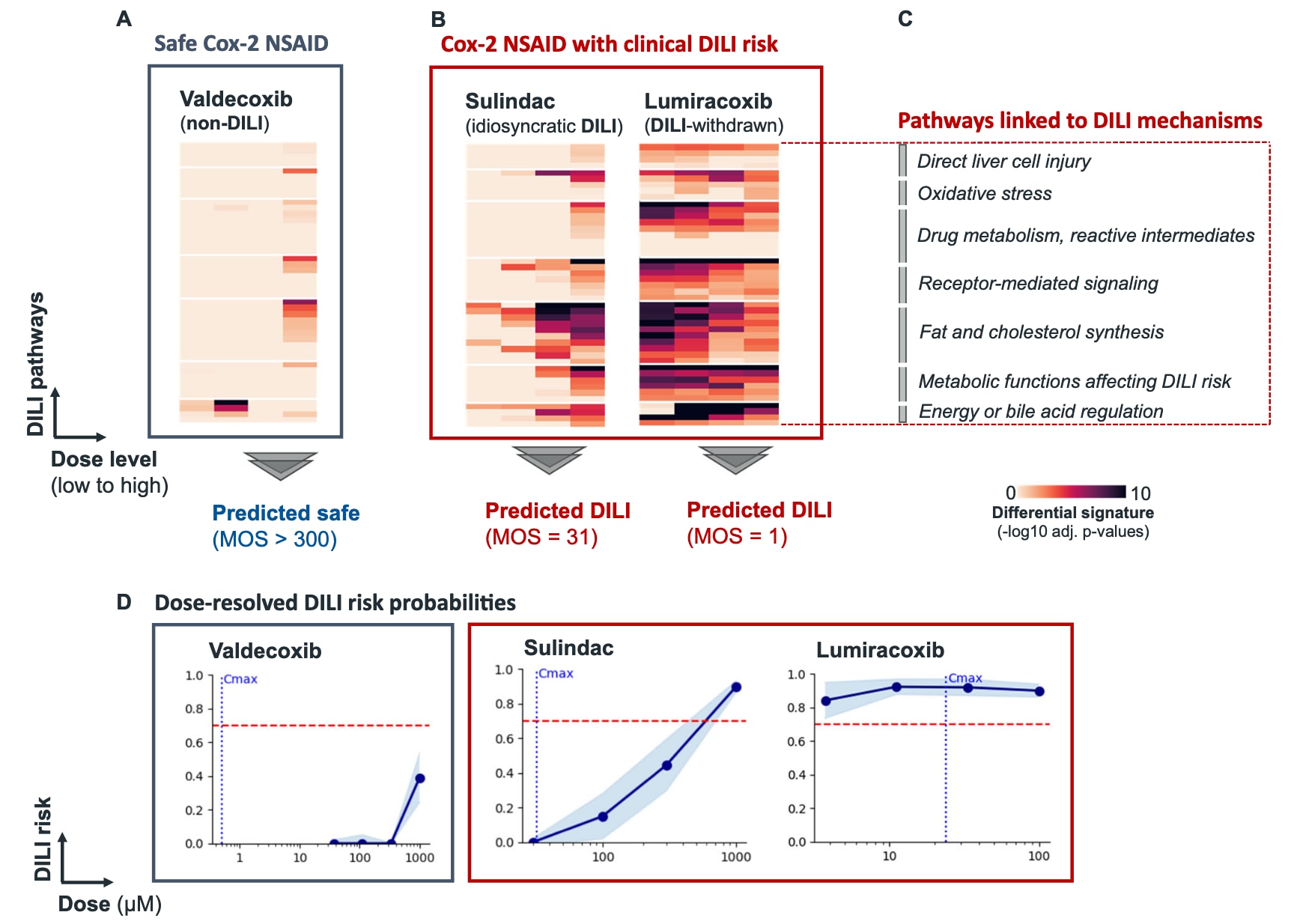
Fig. 3 | 三种靶向Cox2的抑制剂具有不同的肝损伤风险表现。 A Valdecoxib被认为安全,在所有剂量下均无肝损伤风险预测,安全边界(MOS)均高于300。B Sulindac因存在特异性肝损伤报道,以及Lumiracoxib因严重肝衰竭而撤市,均被模型判定为高风险,其MOS分别为31与1。C 这些预测由与肝损伤相关的通路所驱动,差异通路特征突出多类关键机制,包括氧化应激、药物代谢、受体介导信号传导、脂肪与胆固醇合成以及胆汁酸调控。通路富集基于Enrichr(gseapy),采用Fisher精确检验与Benjamini–Hochberg校正,颜色深浅反映–log10调整后p值。D 剂量分辨的肝损伤概率显示剂量升高如何影响肝损伤发生的可能性。散点表示模型的平均预测值,蓝色阴影为模型集成的标准差。这些预测与临床结果高度一致,展示了其在肝损伤风险分层、基于通路的机制理解以及指导更安全药物开发方面的价值。
2.4 解析具有共同靶点但肝毒性表现不同的NSAIDs机制
模型揭示了多种结构与靶点高度相似的COX2抑制类非甾体抗炎药在肝毒性上的差异性,并提供将预测结果与机制层面相联系的线索,包括肝细胞损伤、氧化应激与线粒体功能障碍等核心途径。例如,用于癌痛治疗的Valdecoxib未显示肝损伤风险(Fig. 3A);而用于关节炎治疗、虽罕见但已有特异性肝损伤报道的Sulindac,以及因严重肝衰竭而撤市的Lumiracoxib,则被模型判定为存在肝毒性风险,其安全边界均低于阈值80(Fig. 3B)。值得注意的是,模型还能指出潜在的肝损伤相关通路,包括直接导致细胞损害的氧化应激等因素,以及脂肪酸代谢扰动等更间接的机制,这些通路对于解释特异性肝毒性尤为关键(Suppl. Table S2)。
以Sulindac为例,模型将其与脂肪酸合成与胆固醇生物合成受扰动联系起来,与近期将其与肝脂肪变性相关联的研究相吻合。通过精准定位相关途径,模型为特异性肝损伤提供了机制性解释,而这种类型的肝毒性传统上被认为难以预测(Fig. 3C)。此外,模型能够以剂量分辨的方式评估肝损伤风险,揭示剂量变化如何影响肝损伤概率。例如,尽管Sulindac的特异性肝损伤被认为难以用剂量关系解释,模型仍能准确预测其风险(Fig. 3D)。这些结果表明,该模型能够输出具备可操作性的预测,并通过关注关键通路,为优化药物安全性提供方向。
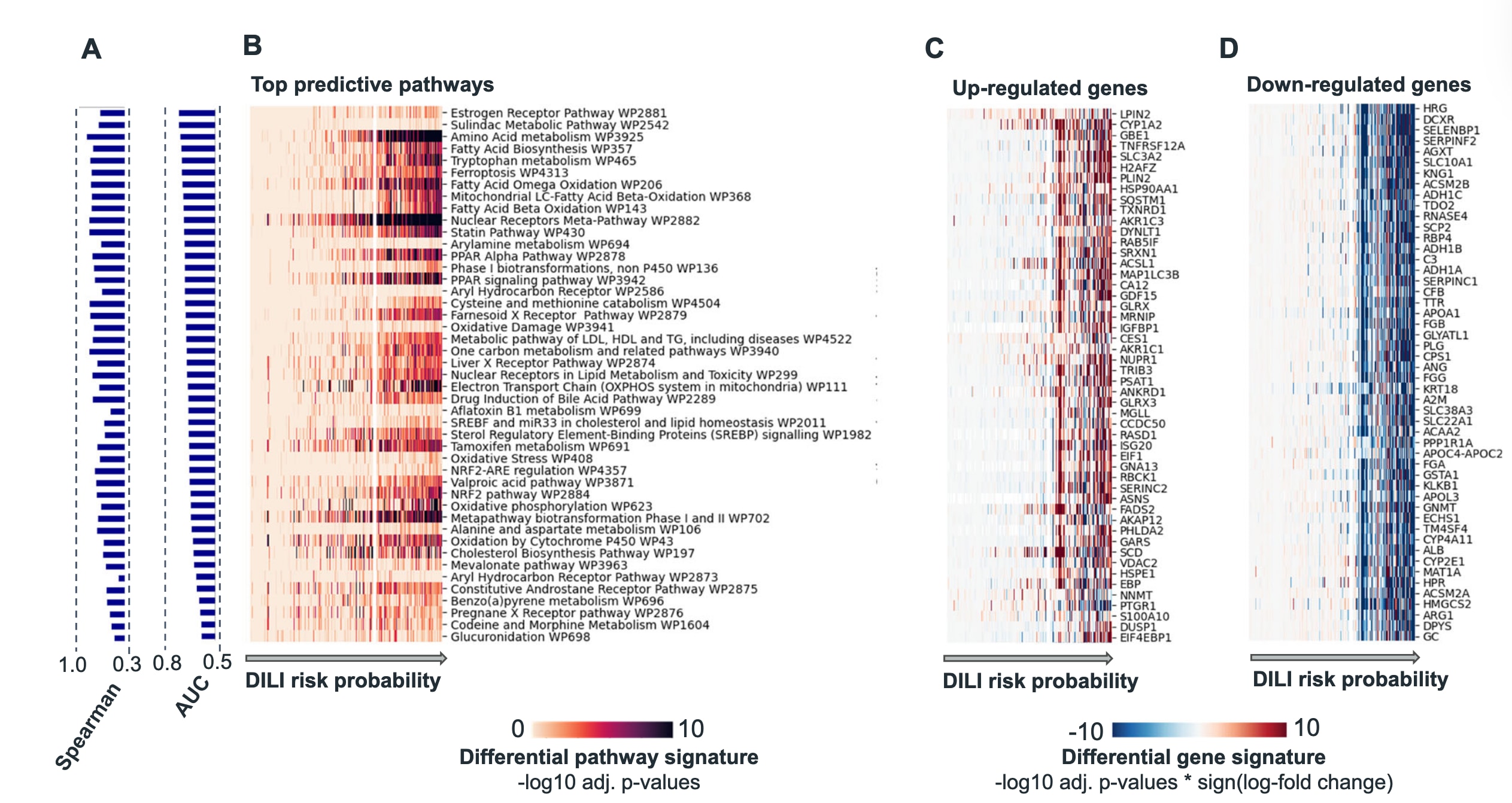
Fig. 4 | 与肝损伤密切相关的关键通路激活与频繁变化的基因 A 根据区分肝毒性药物与无肝毒性药物的能力(以AUC衡量),通路被进行排序。为量化通路失调与预测肝毒性风险之间的关联性,图中展示了通路激活得分与肝毒性概率之间的Spearman相关系数。结果突出显示多个涉及转录与代谢应激的关键通路,例如核受体信号、一碳代谢与胆汁酸调控,这些过程是肝毒性发生的重要贡献者。B 将化合物按肝毒性概率排序后,可观察到与肝损伤相关的通路激活呈现明确的梯度分布。通路富集分析基于Enrichr(gseapy),采用Fisher精确检验与Benjamini–Hochberg校正。最显著的通路包括药物代谢、氧化应激与脂质稳态等核心机制。例如细胞色素P450氧化、NRF2-ARE调控(氧化应激响应)以及脂肪酸β氧化等通路,揭示了解毒、线粒体功能与能量代谢之间的交互作用。此外,核受体通路如PXR与SREBP信号显示脂质与胆汁酸调控受扰动。C 基因差异表达特征基于DESeq2的Wald检验获得,并经Benjamini–Hochberg校正。图中数值表示–log10调整后p值乘以log-fold change的符号。最常上调的100个基因涉及药物代谢(CYP1A2、CYP51A1、UGT1A8、AKR1C1、AKR1C2、AKR1C3)、转运(SLC3A2)、应激反应(TXNRD1、SRXN1、GLRX、GLRX3、GCLM、HSP90AA1、HSP90AB1、HSPE1)以及脂质代谢(PLIN2、INSIG1、SREBF1、SCD、LPIN2、FADS2)。一些较少研究但具有意义的基因也被显著上调,如炎症相关基因(GDF15、TNFRSF12A、S100A10、LITAF)、自噬相关基因(MAP1LC3B、SQSTM1)与线粒体功能基因(VDAC2、RAN、CHCHD10)。D 最常下调的100个基因包括参与药物代谢与解毒(CYP4A11、CYP2E1、AKR1C4、GSTA1、GSTA2、UGT2B10、UGT2B15)、物质转运(SLC10A1、SLC22A1、SLC22A7、SLC38A3、SLC27A5)以及蛋白处理(ALB、AHSG、TTR、APOA1、APOE)的关键基因。涉及氨基酸代谢与线粒体功能的基因(MAT1A、ARG1、CPS1、HMGCS2、ACAA1、ACAA2、ECHS1)亦显著下调。关键氧化应激调控与氧化还原酶基因(CAT、ABAT、GNMT、DHTKD1)同样下降。脂质代谢相关基因(CIDEB、ACSM2A、ACSM2B、ACSM5)以及凝血与炎症反应基因(SERPINF2、SERPINC1、SERPINA6、SERPINA10、FGB、FGG、FGA)亦广泛受影响。数个与肝损伤关联尚不明确但显著下调的基因,如ITIH4、PGLYRP2、BHMT与GUCA2B,可能代表新的机制或通路,为理解肝损伤进展提供新线索。这些表达变化反映肝损伤发生前的早期细胞变化,强调肝毒性机制的复杂性,其涉及肝脏功能的多个方面。
2.5 已知与新发现的肝损伤相关基因与通路
药物性肝损伤源于多条生物通路的扰动。模型揭示了一组与肝损伤风险高度相关、且具有较高预测价值(AUC≈0.8)的通路,包括氨基酸代谢(有毒代谢物累积诱发氧化应激与肝细胞损伤)、脂肪酸生物合成(失衡导致脂质堆积与肝细胞损害)、色氨酸代谢(中间产物推动氧化应激与炎症)以及铁死亡(铁依赖氧化应激引发脂质过氧化)。此外,与预测风险高度相关的通路还包括核受体信号(PXR/CAR/FXR)、一碳代谢与胆汁酸调控,强调转录重编程与代谢压力在肝毒性形成中的关键作用(Fig. 4A)。
当化合物依据预测的肝损伤概率排序后,可观察到一条通路激活的梯度,显示不同生物过程的富集模式在高低风险化合物之间明显分化。这种模式强化了模型预测的机制关联性与可解释性,并将这些通路凸显为可能的致损驱动因素(Fig. 4B)。
为了识别与肝损伤最密切相关的基因,研究者统计了每个基因在肝毒性药物中差异上调或下调的频率(调整后p值阈值0.05)。分析聚焦于首次出现预测毒性的剂量,以捕捉可能位于上游的早期调控因子。最常上调的基因多涉及药物代谢、物质转运、应激反应与脂质代谢,同时也出现与炎症、自噬与线粒体功能相关的新基因(Fig. 4C)。最常下调的基因则与肝脏关键功能密切相关,包括药物代谢、物质转运、脂质与氨基酸代谢、线粒体功能、凝血以及炎症反应。其表达改变可能作为肝损伤的早期指标,反映肝毒性的多层次机制(Fig. 4D)。
尽管明确区分模型识别的通路与基因是否特异于肝毒性而非一般性毒性本质上较具挑战性,但模型的精确度可从其对多种非肝毒性药物的准确分类看出。例如Valdecoxib(心血管毒性)、Bupropion(神经与心血管毒性)与Warfarin(血液系统毒性),均被模型正确归为无肝损伤风险,这表明模型能够将肝脏特异性损伤与其他器官毒性区分开来。
2.6 ToxPredictor能够准确识别近期临床失败药物的肝损伤风险并提供剂量建议
尽管布鲁顿酪氨酸激酶(BTK)抑制剂在肿瘤与自身免疫疾病中展现出显著潜力,但多款候选药物因肝损伤而在临床阶段遭遇搁置。Evobrutinib、BMS-986142与Orelabrutinib均在2023年的Ⅲ期试验中因肝损伤事件而撤回或暂停。研究者将ToxPredictor应用于四种临床失败药物(Evobrutinib、BMS-986142、Orelabrutinib与TAK-875,一种2型糖尿病药物),并以两种在研BTK抑制剂(Rilzabrutinib、Remibrutinib)及两种FDA批准的JAK抑制剂(Tofacitinib、Upadacitinib)作为阴性对照进行评估。
模型在四个浓度下计算肝损伤风险概率,并基于安全边界(MOS)进行风险分类:高风险(MOS≤2.5)、中高风险(MOS≤12.5)、中风险(MOS≤80)以及低风险(MOS>80)。所有临床失败药物均被判定为高风险或中高风险,尤其是TAK-875、Evobrutinib与BMS-986142,与其Ⅲ期撤市的临床结局一致。在研药物分别被归为中风险(MOS=14)与低风险(MOS=101),目前尚无临床肝损伤事件;而两种JAK抑制剂均被归为低风险,预测与临床安全性记录高度吻合(Fig. 5A)。
ToxPredictor还能基于多组假设Cmax推断剂量依赖的肝损伤风险曲线,从而提供剂量调整建议。例如,Rilzabrutinib在每日100 mg以下被归为低风险,但其有效剂量为400 mg;相比之下,Remibrutinib的有效剂量100 mg落在其预测的低风险剂量范围(小于155 mg q.d.)之内。此类分析展示了ToxPredictor在提供安全剂量策略方面的价值,使其成为降低候选药物开发风险的重要工具(Fig. 5B)。
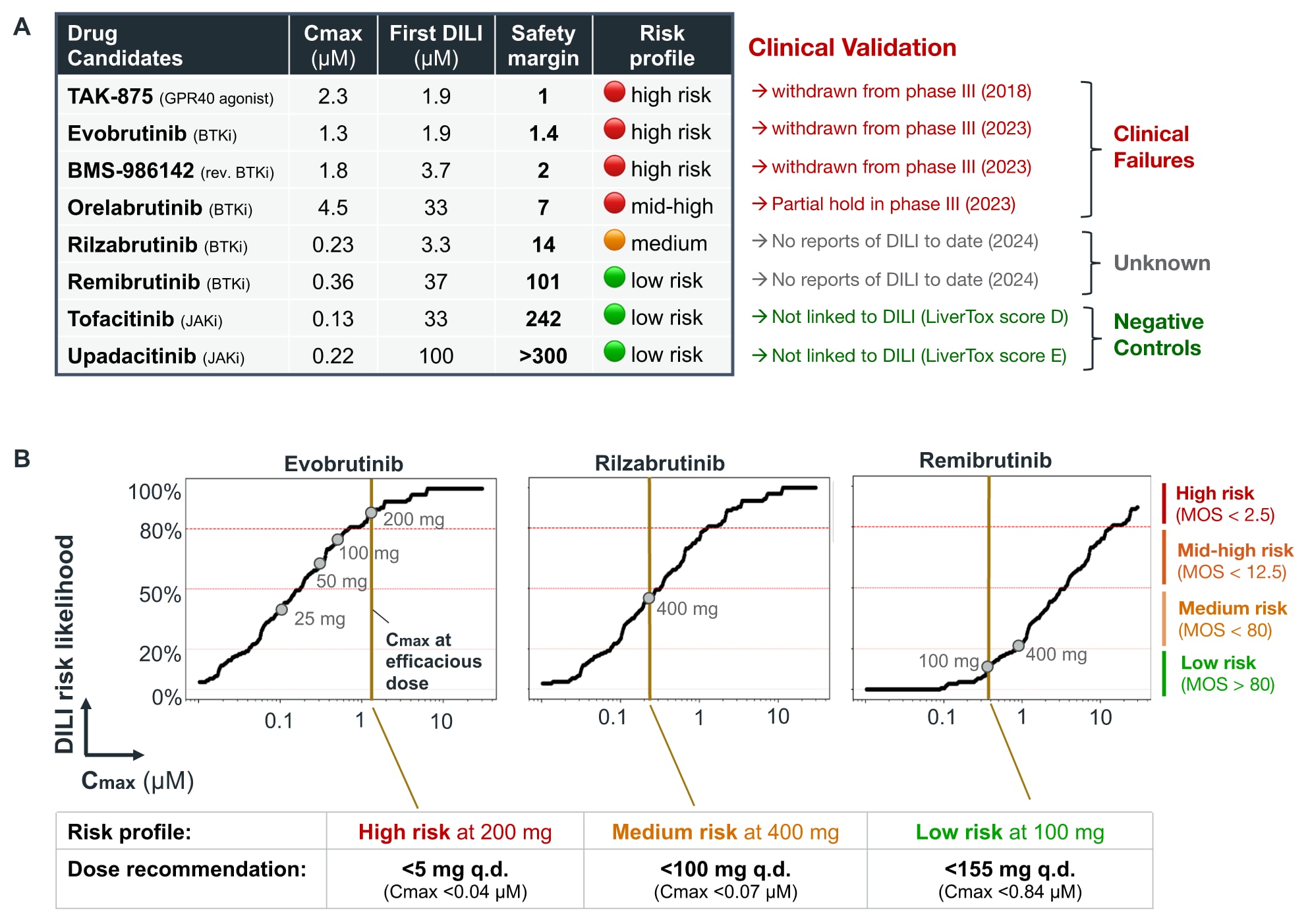
Fig. 5 | 在真实场景中识别近期临床失败药物的肝损伤风险 A ToxPredictor输出包括首次出现肝损伤的预测浓度、安全边界以及对应的风险分类。临床验证表明,被模型判定为高风险的药物,如TAK-875、Evobrutinib与BMS-986142,均已在近期的Ⅲ期临床试验中因肝损伤事件而撤回或终止。相反,被模型归类为低风险的药物,如Tofacitinib与Upadacitinib,在临床中亦未出现肝损伤关联,结果高度一致。B 模型基于不同假设Cmax值绘制代表化合物的肝损伤风险曲线,用以界定低、中、中高与高风险对应的剂量区间,从而为低风险剂量提供推荐。风险分类依据各化合物的有效Cmax确定:Evobrutinib被划分为高风险,Rilzabrutinib为中风险(推荐剂量<100 mg q.d.),而Remibrutinib则属于低风险(推荐剂量<155 mg q.d.)。这些结果展示了模型在剂量选择中的实用价值,可用于降低肝损伤风险并指导更安全的临床给药策略。
2.7 基于毒理基因组学在现有前临床肝损伤模型格局中的定位
研究者从预测性能与可扩展性两条维度对模型进行基准测试。预测性能以平衡准确率衡量,反映模型区分肝毒性阳性与阴性化合物的能力;可扩展性则涵盖技术通量与生物学覆盖度,即模型在不同结构、不同机制甚至未知机制的化合物上保持泛化能力的程度(Fig. 6A)。在多类前临床肝损伤模型中,包括机制检测、细胞毒性标志物、理化性质、生物活化以及BSEP相关方法,模型均表现优越。在对照相同化合物集合的比较中,模型识别出46/66的肝毒性化合物,而Xu等人的HCI检测仅识别27/66;对Garside等人的HCI检测表现也更强(37/46 vs 29/46),对Vorrink等人基于CD类球体的细胞毒性检测亦更优(37/43 vs 30/43),对Sakatis等人GSH加合物生物活化终点及其组合检测也显著提升(47/65 vs 25/65与32/65)。与Kohonen等人基于转录组-细胞毒性的模型相比,模型灵敏度亦明显更高(26/36 vs 16/36)。所有这些比较均在100%特异性下进行,体现出系统层面、机制非依赖性读数的优势(Fig. 6B)。
结构驱动的计算模型如TxGemma、DILIGeNN与DILIPredictor在基准测试中表现不及体外数据驱动的方法。研究者将其应用于314种独立化合物(45种肝毒性阳性、269种阴性)进行评估,分类依据主要来自LiverTox。这些模型的特异性普遍较低,且略低于文献报道的平衡准确率。进一步在与DILImap重叠的未见化合物子集(n=97)中比较,TxGemma的敏感性与特异性为63%与57%,而ToxPredictor达到76%与86%。DILIGeNN在敏感性上表现极高(5/5),但特异性一般(2/3);ToxPredictor在两者上均达到100%。DILIPredictor虽然对23种阳性化合物全部识别成功,但特异性极低(1/7),而ToxPredictor在保持高敏感性(20/23)的同时显著提升了特异性(5/7)。低特异性是结构模型的重要限制,因为缺乏生物背景往往导致毒性过度预测,从而将如生物素、维生素D、培美曲塞等临床安全药物误判为肝毒性阳性。此外,这类模型仅提供二元输出,缺乏剂量信息与机制解释。
相比之下,基于转录组的模型可以提供剂量分辨预测、机制解释能力与安全边界估计,对于评估毒性与制定后续实验具有关键意义。三维肝模型虽然提供生理相关性,但其高内容成像在小型、精炼化合物面板上可取得类似性能,却在更广泛的化学与机制空间中受限。例如在与Vorrink与Fäs的大规模三维检测比较中,三维细胞毒性检测各自独特地识别了少量依赖急性细胞死亡或结构代谢背景的化合物,而ToxPredictor独特识别的化合物数量更多,涉及免疫激活、代谢应激或酶调节等难以通过单一活性终点检测的机制。这表明单终点模型适用于窄机制范围,而在更复杂的化合物多样性中难以泛化。转录组技术通过系统层面的解析能够同时捕捉细胞毒性机制与更微弱、非致死性的早期信号,因此在识别罕见特异性肝损伤方面表现突出。在65种特异性肝损伤化合物中,模型成功识别29种,为所有评估模型中最高,并保持88%的特异性。
进一步地,将毒理基因组学与其他正交检测方法结合可显著提升整体检测能力。例如,与Walker等人的三维HCI检测结合可将肝毒性识别从23/27提升至26/27;与Persson等人在二维系统中的HCI检测结合提升从28/37至30/37;与Sakatis等人的GSH缺失检测结合可将47/65提升至53/65。这类组合策略的平衡准确率可达98%。
基于这些见解,研究者提出了一个分层的去风险策略:以Cmax(<25 μM等PK指标)与细胞毒性检测作为初筛手段识别明显的肝毒性风险;对于未出现明显信号的候选,毒理基因组学提供最全面的风险评估;对于少量进入后期开发的候选,在资源充足的情况下,可进一步在三维肝模型中应用毒理基因组学,以获得更贴近体内反应的预测。这一策略兼顾资源效率与机制广度,有助于在药物研发中实现更可靠的肝损伤风险评估。
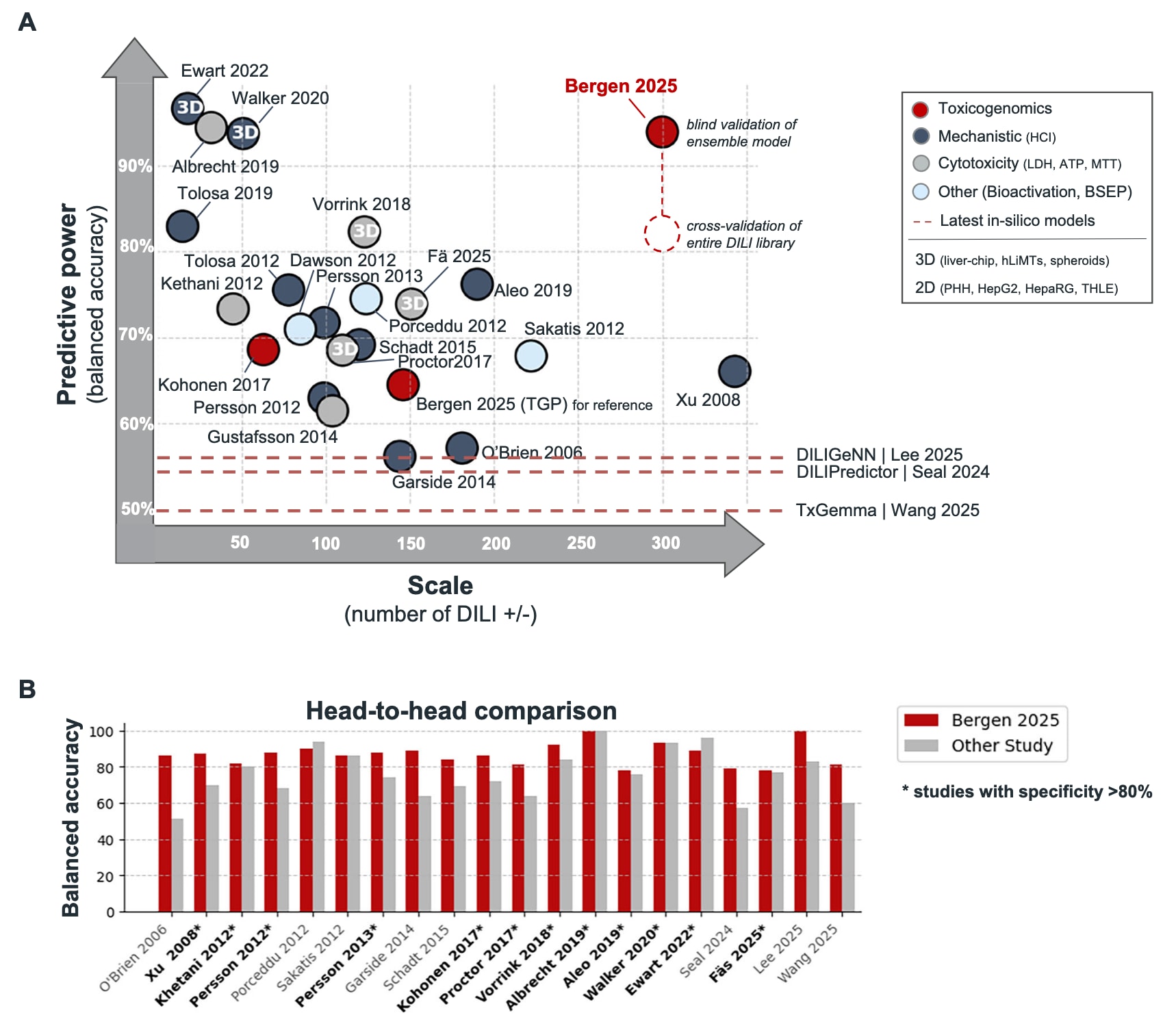
Fig. 6 | ToxPredictor在准确性与可扩展性上优于现有前沿预测模型 A 各前临床模型的平衡准确率与规模(肝毒性阳性/阴性化合物数量)对比显示,ToxPredictor在体外与计算模型中均达到最高性能。相较高内容成像、细胞毒性检测、生物活化端点以及结构基础模型,ToxPredictor在扩展至数百种化合物时仍保持稳健表现,体现其在规模化应用中的优势。B 在重叠化合物上的直接对比进一步显示模型在多个研究体系中均稳定优于其他方法。特异性超过80%的模型(以*标记)更适用于可信赖的前临床去风险评估。整体来看,这些结果凸显了基于转录组的毒理基因组学方法作为一种机制驱动且可扩展的肝毒性预测策略的领先地位。
3 讨论
基于毒理基因组学的方法从系统层面捕捉细胞对药物的响应,能够提供远超传统方法的丰富信息,从而更细致地理解肝毒性,包括机制未明的特异性反应。通过从单一指标转向系统视角,并结合机器学习,这一策略在毒理预测中展现出显著潜力,能够提供剂量依赖的预测与安全边界,而不仅仅是简单的DILI/非DILI分类。其广泛的机制覆盖为前临床筛查带来重要优势。依托专为肝损伤研究构建的全面转录组资源,该方法在性能上显著超过现有技术。在盲测中获得88%敏感性与100%特异性,同时准确识别出动物模型与临床研究均未察觉的撤市药物或临床失败案例,凸显其实用价值,也标志着预测毒理学向前迈出重要一步。
作为更大AI/ML平台的核心组件,这一方法具备良好的可扩展性与适应性,可提升药物研发流程的预测性能与效率。然而,需要认识到其固有局限性。首先,肝损伤由遗传、环境与生活方式等多因素共同驱动,即便最先进的模型也无法穷尽所有潜在机制,模型无法覆盖全部个体差异与罕见遗传易感性。其次,模型的预测能力受限于DILImap数据库的完整性,尤其是在特异性肝损伤案例记录不足时可能影响准确性。第三,二维肝细胞培养体系无法再现肝细胞与其他细胞类型之间的复杂相互作用,因而难以捕捉某些依赖组织结构或多细胞互作的机制。第四,24小时的检测时间难以识别延迟性或免疫介导的毒性。第五,与所有前临床模型一样,其最终价值仍取决于临床预测的一致性,而临床结果往往受多种不可控因素影响。
展望未来,毒理基因组学将在肝毒性理解与预测中发挥更大作用。将RNA-seq与先进的三维培养体系,如类器官、球体及肝芯片技术结合,可进行更长时间的药物暴露,并纳入库普弗细胞、肝星状细胞等非实质细胞,从而捕捉免疫激活、炎症与纤维化等关键过程,这是二维培养无法再现的。尽管三维体系目前多依赖高内容成像或细胞毒性终点,其机制覆盖有限、规模扩展受限,但随着RNA-seq在这些体系中逐渐可行,与三维系统的结合有望形成强大的协同效应,既具生理相关性又具系统机制深度。该研究为此类融合奠定基础,展示转录组技术本身即可在多种机制下稳健识别肝损伤风险。
进一步结合分子结构信息可深化对药物与细胞组分相互作用的理解,有助于提高毒性预测精度。以早期的靶点活性估计作为Cmax替代,可在药物发现早期引入安全边界评估。未来构建包含肝损伤调控网络的多变量模型、引入染色质可及性、蛋白质组或代谢组等多组学数据,都将进一步丰富对肝毒性机制的理解。随着这些技术在药物研发早期的整合,有望减少与肝脏相关的不良事件,提升研发效率并节约成本,更重要的是提高患者安全性。毒理基因组学与机器学习、多组学技术的持续发展,标志着药物安全评价正迈向更可预测的未来,有望在新药开发中发挥日益重要的作用。