NMI 2022 | MolCLR: 基于分子对比学习的图神经网络模型
今天介绍的这项工作来自 Nature Machine Intelligence。在标注分子数据极其有限、化学空间又极其庞大的现实下,怎样通过自监督学习获得通用而且可迁移的分子表示,用于分子性质预测和药物发现。传统方法要么依赖人工构造的分子指纹(如ECFP),要么使用SMILES+RNN/Transformer,或者直接在分子图上训练GNN,但在小数据、分布偏移的场景下,它们往往泛化能力不足。研究团队提出了基于图对比学习的自监督框架MolCLR,在包含三种分子图增强策略(原子掩蔽、键删除、子图移除)的大规模无标注分子数据上进行千万级预训练,并在多种下游分子性质预测任务上取得SOTA性能。t-SNE 可视化和指纹相似性分析进一步表明,MolCLR 学到的表征能够有效捕捉分子骨架与官能团层面的结构相似性,同时在分子筛选与药物发现中开创性地引入对比学习以及“预训练和微调”的等方法,对后续方法设计具有重要的启发意义。

获取详情及资源:
0 摘要
分子机器学习在高效分子性质预测和药物发现中具有巨大潜力。然而,标注分子数据的获取往往代价高昂且耗时。由于标注数据有限,监督学习模型在面对庞大化学空间时很难实现良好的泛化能力。为此,该研究提出MolCLR(Molecular Contrastive Learning of Representations via Graph Neural Networks),这是一种利用大规模未标注数据(约一千万个独特分子)的自监督学习框架。在MolCLR的预训练阶段,首先构建分子图,并设计图神经网络编码器来学习可微的分子表征。该方法提出了三种分子图增强方式:原子遮蔽(atom masking)、键删除(bond deletion)和子图移除(subgraph removal)。对比学习估计器通过最大化同一分子不同增强视图之间的一致性、最小化不同分子之间的一致性来进行训练。实验表明,该对比学习框架显著提升了图神经网络编码器在多种分子性质基准任务上的表现,涵盖分类和回归任务。得益于在大规模未标注数据库上的预训练,MolCLR在若干具有挑战性的基准数据集上微调后可以达到当前最优水平。此外,进一步分析表明,MolCLR能够将分子嵌入到一种表征空间中,使其能够区分在化学上合理的分子相似性。
1 引言
分子表征在功能化和新型化合物的设计中是基础且关键的。由于潜在稳定化合物的数量极其庞大,要构建一种能够在整个化学空间上具备良好泛化能力的信息表征极具挑战。传统分子表征方法(如扩展连接指纹ECFP)已经成为计算化学中的标准工具。近年来,随着机器学习方法的发展,数据驱动的分子表征学习及其在分子性质预测、化学建模和分子设计等任务中的应用,受到越来越多关注。然而,学习高质量分子表征面临三大主要挑战。
首先,很难彻底地刻画分子信息。例如,基于字符串的表示方式,如SMILES和SELFIES,无法直接编码分子的拓扑结构信息。为了保留丰富的结构信息,近来的许多工作开始使用图神经网络GNN来处理分子图,并在分子性质预测任务中取得了不错的效果。第二,化学空间的规模极其庞大,比如潜在具有药理活性的分子数量被估计在10^60量级,这使得任何分子表征方法在整个潜在化学空间上实现泛化都变得非常困难。第三,用于分子学习任务的标注数据既昂贵又远远不足,尤其是与潜在化学空间的规模相比更是如此。获得分子性质标签通常需要复杂且耗时的实验过程。化学研究本身又极其广泛,涉及的性质从量子力学到生物物理各个层面,这进一步加剧了问题的复杂度,因此大多数分子学习基准数据集中的标签数量都严重不足。在这种有限数据条件下训练得到的机器学习模型很容易过拟合,并且在与训练集差异较大的分子上表现较差。
在过去十年中,伴随机器学习尤其是深度神经网络的发展,分子表征学习取得了快速进展。在传统化学信息学中,分子通常被表示为指纹向量(如ECFP),然后基于这些指纹构建深度神经网络来预测相关性质。除了指纹之外,基于字符串的表示(如SMILES)也被广泛用于分子学习。基于RNN的语言模型天然适用于从SMILES中学习分子表征。随着transformer结构的成功,此类语言模型也被用来从SMILES中学习表示。
近期,自然能够编码结构信息的GNN被引入到分子表征学习中。MPNN和D-MPNN通过消息传递机制在分子图上聚合信息;SchNet在GNN中显式建模分子内部的量子相互作用;DimeNet则通过根据原子间角度变换消息来引入方向信息。随着可用分子数据规模的增长,自监督/预训练的分子表征学习也开始受到重视。类似BERT的自监督语言模型已被用来基于SMILES学习分子表示。在分子图上,N-Gram Graph通过组合短游走路径上的顶点嵌入来构建图表示,不需要额外训练。也有工作为GNN设计了节点级和图级的预训练任务,但其中图级预训练依赖有监督任务,受到有限标签的约束。还有方法将对比学习扩展到非结构化图数据上,但该框架并非针对分子图专门设计,而且只在有限的分子数据上进行训练。
在该研究中,提出了如图1所示的MolCLR(Molecular Contrastive Learning of Representations via Graph Neural Networks),用于解决前文提到的这些挑战。MolCLR是一种自监督学习框架,在约1000万条未标注分子构成的大型数据集上进行训练。通过对比损失函数,MolCLR通过对比正样本分子图对与负样本分子图对,学习有效的分子表示。
MolCLR引入了三种分子图增强策略:原子掩蔽(atom masking)、键删除(bond deletion)和子图移除(subgraph removal)。由同一个分子通过增强得到的图对被视为正样本对,而来自不同分子的增强图对则被视为负样本对。在MolCLR中,采用了两类广泛使用的GNN模型——图卷积网络(GCN)和图同构网络(GIN)——作为编码器,从分子图中提取信息量丰富的表示。随后,将预训练好的模型在MoleculeNet提供的下游分子性质预测基准数据集上进行微调。与直接通过有监督学习训练的GCN与GIN相比,MolCLR在分类和回归任务上都取得了显著性能提升。得益于在大规模未标注数据库上的预训练,MolCLR在多个分子基准任务上优于其他自监督学习和预训练方法。
此外,在若干任务上,即便与那些使用复杂分子图卷积算子或领域特定特征工程的有监督学习基线方法相比,MolCLR也能达到甚至超越其性能。该研究还表明,当将这些分子图增强策略直接作为数据增强模块应用到有监督学习中时,也能显著提升分子基准任务的表现。进一步将MolCLR表征与传统指纹(FP)对比的结果表明,MolCLR通过在大规模未标注数据上的预训练,学会了区分化学上合理的分子相似性。
总结而言:
- 提出了MolCLR这一用于分子表示学习的自监督框架;
- 设计了三种用于生成对比样本对的分子图增强策略:原子掩蔽、键删除和子图移除;
- 受益于在大规模未标注数据上的预训练,经MolCLR训练的简单GNN模型在所有分子基准任务中相较传统有监督学习取得了显著性能提升;
- 与无法利用未标注数据的更复杂GNN模型相比,结合微调策略后,MolCLR甚至可以将这些简单GNN模型提升到若干分子基准任务的SOTA水平。
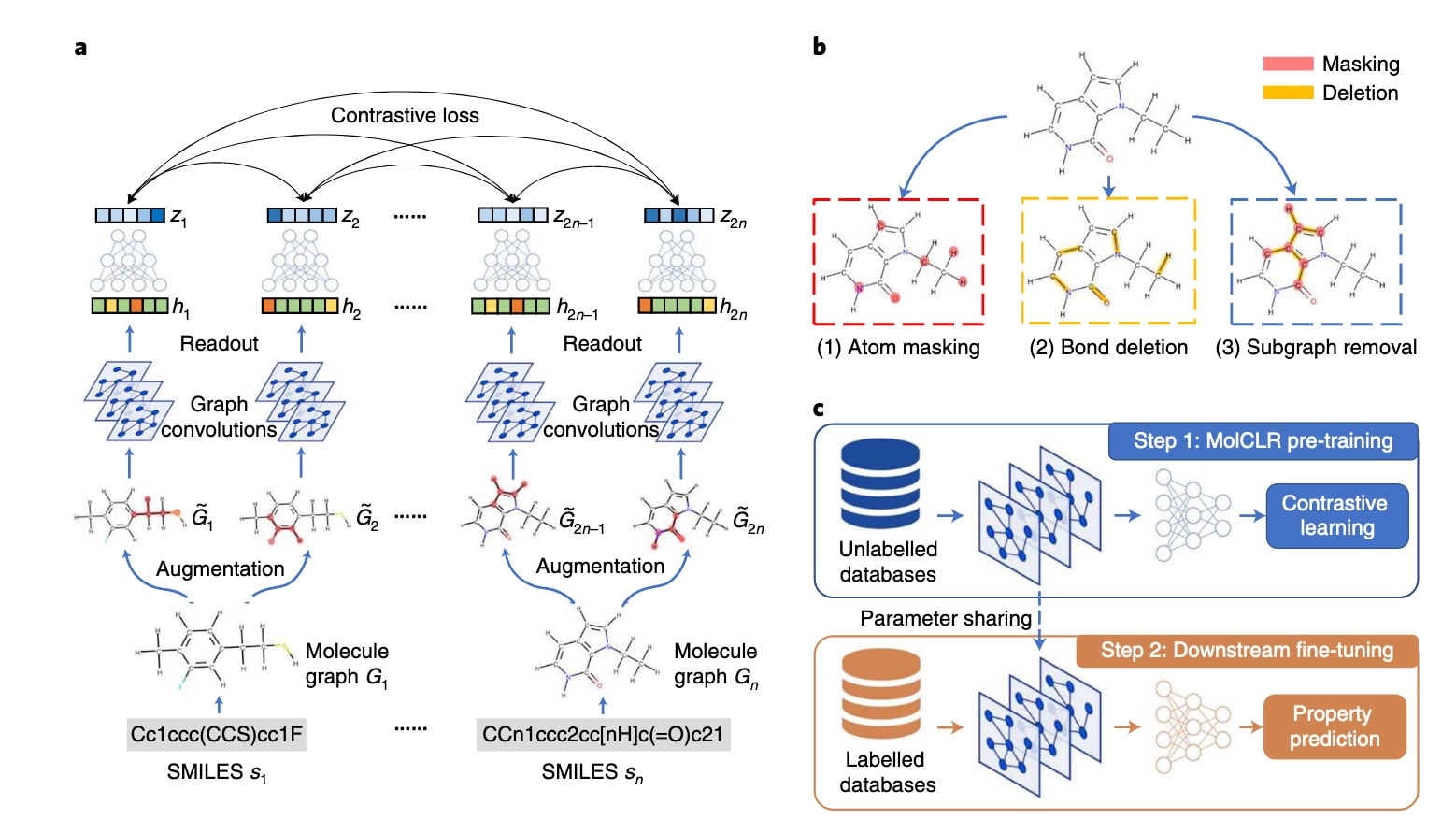
图1 | 展示了MolCLR的框架概览。 a 基于掩码图模型、并引入额外知识增强的知识引导预训练策略。分子被表示为分子线图(molecular line graphs),用于刻画原始分子图中边与边之间的相邻关系。b 基于经典Transformer架构的线图 Transformer。c 下游分子性质预测任务中的微调(finetuning)设置迁移学习,此时预训练好的 LiGhT 参数是可训练的。在该设置下,可以采用多种微调策略,例如分层学习率衰减(layer-wise learning rate decay)、重新初始化(re-initialization)、FLAG 和 L2-SP 等。d 下游分子性质预测任务中的特征提取(feature extraction)设置迁移学习,此时预训练好的 LiGhT 参数保持固定。这里的 **神经指纹(neural fingerprints)**是指由预训练LiGhT生成的分子特征表示,作为信息丰富且具有区分度的分子表示。MLP表示多层感知机(multi-layer perceptron),Linear表示线性层(linear layer),MatMul表示矩阵乘法(matrix multiplication),DE表示距离编码(distance encoding)模块,PE表示路径编码(path encoding)模块。
2 结果
2.1 MolCLR框架概述
该研究的MolCLR模型构建在对比学习框架之上。通过对比来自“正样本”增强分子图对的潜在表示与来自“负样本”分子图对的表示来进行表征学习。整个流程(如图1a所示)由四个部分组成:数据处理与增强、基于GNN的特征提取器、非线性投影头以及归一化温度缩放交叉熵(NT-Xent)对比损失。对于一个批量大小为N的SMILES数据sn,首先构建对应的分子图Gn,其中每个节点表示一个原子,每条边表示原子之间的一条化学键。通过分子图增强策略,由同一个分子增强得到的两个分子图构成正样本对,而来自不同分子的增强图对则构成负样本对。
特征提取器f(·)由GNN实现,用于将分子图映射为表示hi,hj∈ℝᵈ。在该研究中,使用了带有平均池化的GCN和GIN作为特征提取器。非线性投影头g(·)由一个带单隐层的MLP建模,将表示hi和hj进一步映射为潜在向量zi和zj。NT-Xent损失作用于这2N个潜在向量z,通过最大化正样本对的一致性、最小化负样本对的一致性来进行对比学习。
整个框架在约1000万条来自PubChem的未标注分子数据上进行预训练。预训练完成后,MolCLR得到的GNN模型将用于下游分子性质预测任务(如图1c所示)。与预训练阶段类似,下游预测模型同样由一个GNN骨干网络和一个MLP头部组成:前者与预训练时的特征提取器结构相同,后者则将特征映射为具体的分子性质预测值。在微调阶段,GNN骨干网络的参数由预训练模型初始化,而MLP头部则随机初始化。随后,在目标分子性质数据集上以有监督学习的方式对整个微调模型进行训练。
2.2 MolCLR分子图增强策略
MolCLR中对输入分子采用三种分子图数据增强策略(图1b):原子掩蔽(atom masking)、键删除(bond deletion)和子图移除(subgraph removal)。
原子掩蔽(Atom masking)在原子掩蔽中,分子图中的部分原子会按给定比例被随机掩蔽。当某个原子被掩蔽时,其原子特征xᵥ会被替换为一个特殊的掩蔽标记m,该标记与图中任何真实原子特征都不同,如图1b红色方框所示。通过这种掩蔽操作,模型被迫去学习分子内部的本征化学信息(例如某些共价键两端可能连接的原子类型等)。
键删除(Bond deletion)。键删除会以一定比例随机删除原子之间的化学键,如图1b黄色方框所示。与将原子特征替换为掩蔽标记的原子掩蔽不同,键删除是一种更“激进”的增强方式,因为它是直接从分子图中移除边。原子间化学键的生成与断裂决定了分子在化学反应中的性质。键删除在一定程度上模拟了化学键的断裂,从而促使模型去学习一个分子在不同反应参与形式之间的关联。
子图移除(Subgraph removal)。子图移除可以看作原子掩蔽与键删除的组合。该操作首先从分子中随机选取一个起始原子,然后按层次逐步向外扩展:先掩蔽该原子的邻居,再掩蔽邻居的邻居,如此反复,直到被掩蔽原子的数量达到总原子数的某一既定比例。随后,将这些被掩蔽原子之间的键全部删除,使得这些原子及其内部的键构成原分子图的一个诱导子图。如图1b蓝色方框所示,被移除的子图包含所有被掩蔽原子之间的键。通过对比不同子结构被移除后的分子图,模型会学习在剩余子图中识别那些决定分子性质的关键结构模体(motif),而这些模体往往对分子性质具有决定性影响。
表1 | 展示了七个分类基准上测试不同模型的性能。

2.3 MolCLR分子性质预测性能
为验证MolCLR的有效性,该研究在MoleculeNet提供的多个具有挑战性的分类与回归任务上进行了系统评测。表1给出了MolCLR在分类任务上的测试ROC曲线下面积(ROC-AUC(%)),并与多种监督学习和自监督/预训练基线模型进行了对比。结果以三次独立运行的平均值和标准差形式汇报。MolCLRGCN和MolCLRGIN分别表示以GCN和GIN作为特征提取器的MolCLR预训练模型。对表1的主要观察包括:
(1) 与其他自监督或预训练方法相比,MolCLR在7个基准中的5个上取得了最优性能,平均提升幅度为4.0%。这一结果表明,MolCLR是一种功能强大、实现简洁且几乎不依赖领域特定技巧的自监督学习策略。
(2) 与表现最好的监督学习基线相比,MolCLR也展现出具有竞争力的性能。在某些基准(如ClinTox、BACE、MUV)上,预训练后的MolCLR甚至超过了当前的SOTA监督学习方法,这些监督模型往往采用了复杂的图聚合操作或领域特定的特征工程。例如,在ClinTox数据集上,相较于监督版D-MPNN,MolCLR在ROC-AUC上提升了2.7%。
(3) 值得注意的是,在分子数量较少的数据集(如ClinTox、BACE和SIDER)上,MolCLR依然表现出色,说明MolCLR学到的表征具有良好的可迁移性,可以在不同数据集间有效复用。
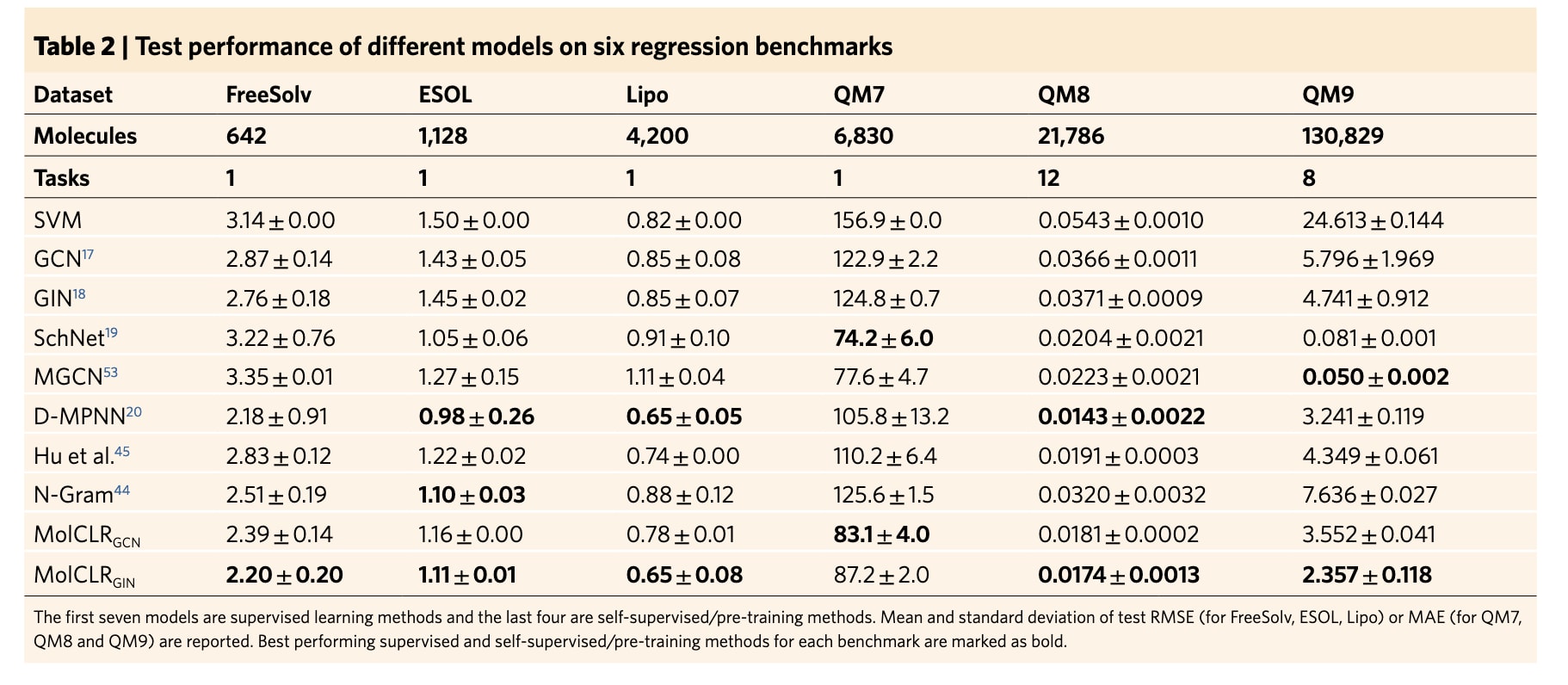
表2展示了MolCLR与各基线模型在回归任务上的测试表现。FreeSolv、ESOL和Lipo以均方根误差(RMSE)作为评估指标,而QM7、QM8和QM9则采用平均绝对误差(MAE),这一设置与MoleculeNet的推荐保持一致24。相较于只依赖人工离散标签的分类任务,回归任务更具挑战性。对表2的主要结论如下:
(1) MolCLR在6个回归基准中的5个上优于其他预训练基线,在剩余的ESOL数据集上也取得了几乎相同的表现。与同样使用GIN作为编码器的文献45相比,MolCLRGIN在全部6个回归数据集上都取得了更好的结果。例如,在QM7和QM9上,相较于Hu等人的方法,提升幅度分别达到20.9%和45.8%。
(2) 与监督学习模型相比,MolCLR在大多数情况下也获得了具有竞争力的结果。例如,在Lipo数据集上,MolCLR与表现最好的监督D-MPNN模型有着相近的性能。同时,在所有回归基准上,经由MolCLR预训练的GCN和GIN都优于未预训练版本。尽管在QM9上,MolCLR尚未能超越专门为量子相互作用设计且使用额外三维位置信息的监督模型SchNet19和MGCN53,但需要强调的是,SchNet与MGCN虽然在涉及量子力学属性的数据集(QM7、QM8、QM9)上表现突出,却并未在其他基准上全面优于常规监督模型。与此同时,在极具挑战性的QM9数据集上,MolCLR预训练依然被证明是有效的:与未预训练的GCN和GIN相比,MolCLR将其性能分别提升了38.7%和50.3%。此外,MolCLR在QM9上的表现也优于其他自监督基线,进一步验证了MolCLR框架的有效性。鉴于QM9中包含多种量纲与数量级差异较大的性质,详细结果见补充表3。
综合表1和表2可以看到,相较于监督版GCN和GIN,经由MolCLR预训练后,所有基准上的性能均得到显著提升,这充分体现了MolCLR的有效性。在分类基准上,MolCLR带来的平均增益为:GCN提升12.4%,GIN提升16.8%;在回归基准上,对GCN的平均提升为27.6%,对GIN的平均提升为33.5%。总体而言,GIN通过MolCLR预训练获得的相对提升更大,这可能是因为GIN的参数量更高,更有能力学习到更具代表性的分子特征。
同时,MolCLR在大多数场景下都优于其他预训练/自监督基线。需要特别强调的是,MolCLR受益于在大规模未标注数据库上的预训练,而其他监督或自监督基线往往无法利用这些未标注数据。对未标注数据的充分利用,使MolCLR在化学空间泛化与多种分子性质建模方面具有显著优势。关于预训练数据库对MolCLR影响的更详细分析,可参见补充表4与补充图1。总体来看,这种出色的泛化能力为未来在药物发现与分子设计中预测潜在分子性质提供了重要支持。
表2 | 展示了在六个回归基准上测试不同模型的性能。
2.4 探索最优分子图增强策略
最优分子图增强策略。为系统分析不同分子图增强策略的效果,该研究比较了原子掩蔽(atom masking)、键删除(bond deletion)和子图移除(subgraph removal)的不同组合。图2a给出了在不同基准数据集上,各种数据增强策略的ROC-AUC(%)均值及标准差。具体考虑了四种增强方式:(1)原子掩蔽与键删除的组合,两者比例p均设为25%;(2)子图移除,掩蔽比例p在0%到25%之间随机采样;(3)子图移除,固定比例为25%;(4)三种增强方式的组合。更具体地说,对于策略(4),首先以0%到25%的随机比例执行一次子图移除;如果被掩蔽原子的比例仍小于25%,则继续随机掩蔽原子直至达到25%;同样地,若键删除比例小于25%,则进一步删除键直到达到设定比例。
如图2a所示,在这四种增强组合中,固定25%比例的子图移除在平均性能上表现最佳。这一优异表现可以归因于子图移除本身就是原子掩蔽和键删除的内在组合,并且相较于策略(1),子图移除能够更好地“拆解”局部子结构。然而,在BBBP数据集上,固定25%比例的子图移除表现较差,因为BBBP中的分子结构对拓扑变化高度敏感,哪怕是轻微的结构改动也可能导致性质发生巨大差异。
此外,值得注意的是,三种增强方式的组合(策略4)在多数基准上反而会削弱性能,相比仅使用子图移除时的ROC-AUC有所下降。一个可能的原因是:三种增强策略叠加后,可能会在分子图中删除过多、过广的子结构,从而丢失关键的拓扑信息。总体而言,子图移除在大多数基准数据集上取得了更优表现。不过,这些结果也表明:最优的分子图增强策略并非完全与任务无关,仍然会受到具体数据集与任务特性的影响。
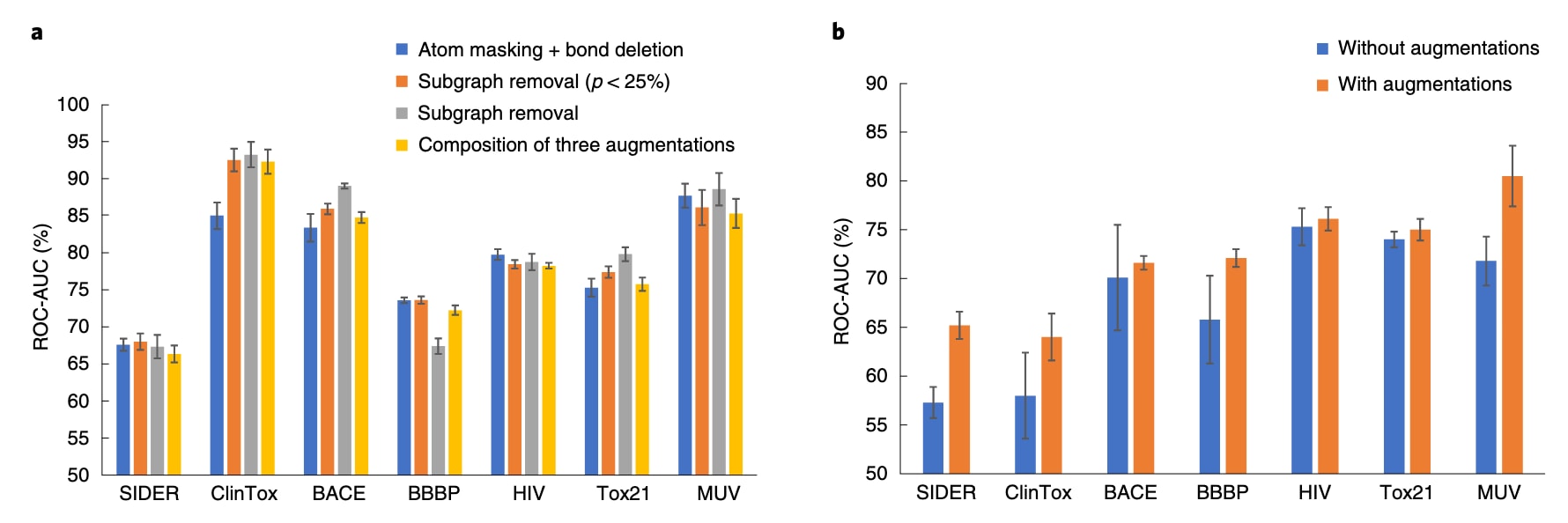
图2 | 展示了在分类基准上的分子图增强策略分析。 a,MolCLR在采用不同分子图增强组合策略时的测试表现。b,使用或不使用分子图增强时,经监督学习训练的GIN模型的测试表现。柱状条的高度表示在各基准数据集上的平均ROC-AUC(%)。
2.5 探索最优分子图增强策略
基于监督学习的分子图增强。该研究中提出的分子图增强策略——原子掩码、键删除和子图移除——可以作为通用的数据增强模块,应用于任意基于图的分子学习方法。为了验证这些分子图增强方法在监督式分子任务中的有效性,研究中从随机初始化出发,分别在“使用增强”和“不使用增强”的条件下训练GIN模型。具体而言,这里采用固定比例为25%的子图移除作为增强方式。
图2b展示了在七个分子性质分类基准上的测试ROC-AUC(%)的均值和标准差。结果表明,在这七个基准任务上,引入增强的GIN模型全部优于未使用增强的模型。分子图增强使平均ROC-AUC提升了7.2%。这说明,即使没有预训练,将分子图增强策略直接用于监督式分子性质预测任务,也能显著提升模型表现。结果进一步表明,分子图增强对于帮助GNN学习鲁棒且具有代表性的特征是有效的。例如,子图移除操作会构造出“部分可见”的分子图,迫使模型从残余子图中识别关键基序(motif),从而显著促进分子性质学习。
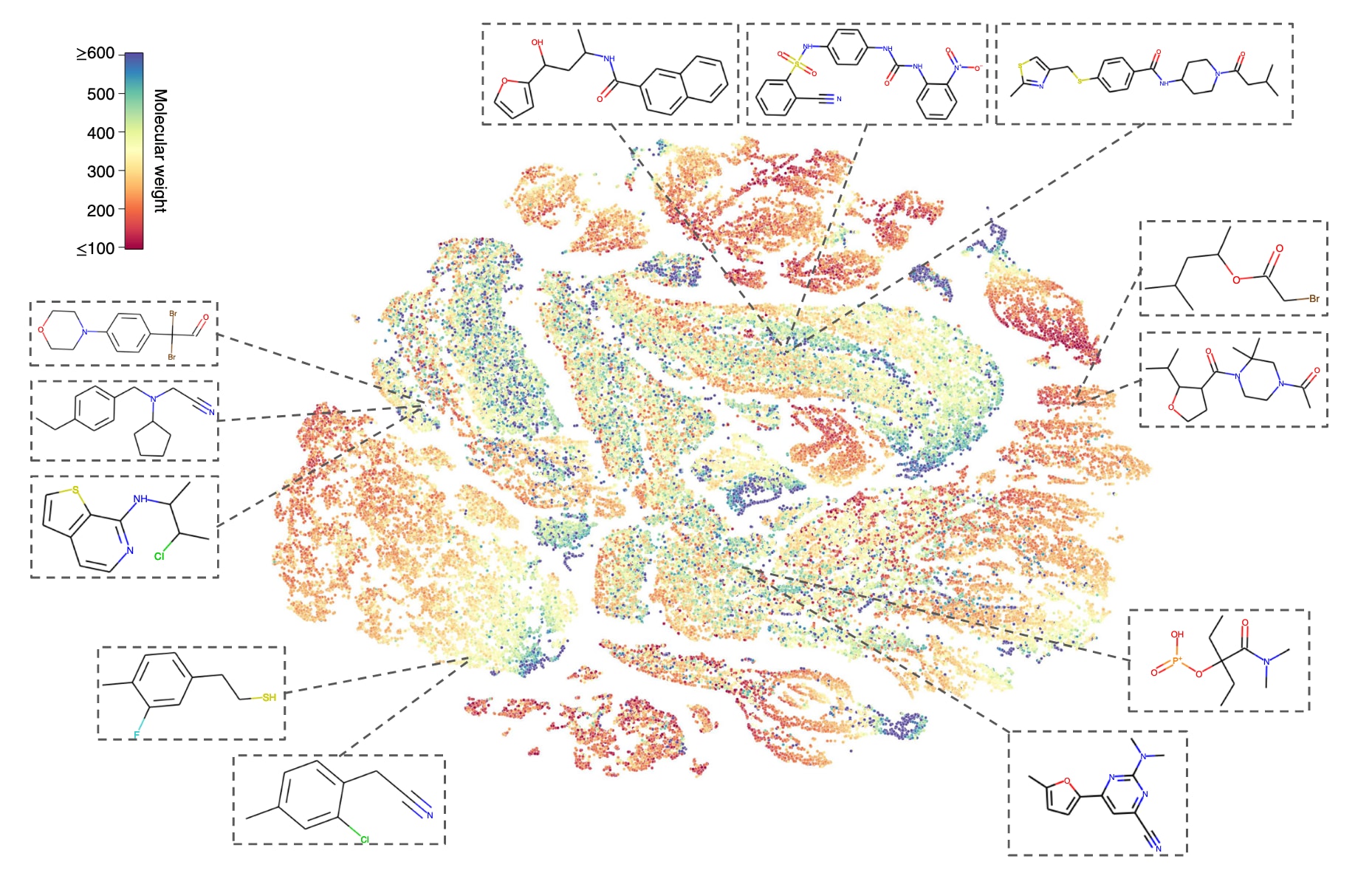
图3 | 展示了通过t-SNE可视化MolCLR学习得到的分子表示。 表示向量提取自预训练数据集的验证集,共包含100,000个不同分子。每个点按其对应的分子量着色,并展示了在表征空间中彼此接近的部分分子结构示例。
2.6 MolCLR学习到的分子表征
该研究利用t-SNE嵌入对预训练MolCLR模型学到的分子表征进行分析。t-SNE算法将高维的分子表示映射到二维空间,使得在表征空间中相近的分子会被投影到二维平面上的相邻位置。图3展示了来自PubChem验证集的10万个分子在t-SNE空间中的二维嵌入,并用分子量对点进行着色。为了说明MolCLR预训练所学到的“相似/不相似”分子的关系,作者还在图中标出了部分随机挑选的具体分子。可以看到,MolCLR会将拓扑结构相似、官能团相似的分子映射到彼此靠近的位置。例如,图中上方的三个分子都含有与芳环相连的羰基;左下方的两个分子则具有类似骨架结构,苯环上连接了卤素原子(氟或氯)。这说明即便在没有监督标签的情况下,模型也能自动学习到分子之间的内在联系:性质相近的分子,其特征表示也会彼此接近。
为了进一步评估MolCLR,研究团队将其学习到的分子表示与传统的分子指纹(如ECFP和RDKFP)进行对比。具体做法是:给定一个查询分子,首先用MolCLR提取其表示向量,然后计算它与预训练数据库中所有参考分子的余弦距离。随后,将所有参考分子按该距离从近到远排序,并根据排名百分位均匀划分为20个区间。距离越小(百分位越靠前),意味着在MolCLR表征空间中越“相似”的分子。对每个区间,随机抽取5000个分子,计算它们与查询分子的Dice指纹相似度(分别基于ECFP和RDKFP)。
图4给出了一个示例查询分子(PubChem ID 42953211)的结果。图4a展示了每个区间内指纹相似度的均值和标准差,图4b则给出了ECFP和RDKFP相似度的分布情况。ECFP通常给出的相似度略低于RDKFP,因为ECFP覆盖了更广泛的、与分子活性相关的结构特征。可以看到:随着MolCLR表征距离的增大,ECFP和RDKFP的相似度都在下降。以RDKFP为例,前5%最近邻的平均相似度约为0.9,而最后5%则降至约0.67;ECFP的平均相似度则从前5%的约0.49降至最后5%的约0.21。尽管随着百分位变化会有一定波动,但整体趋势非常一致:MolCLR表征空间中的“远近”能够有效反映传统化学指纹所刻画的分子相似性。
此外,图4c展示了在MolCLR表征空间中与该查询分子最近的9个分子,并标注了它们的ECFP和RDKFP相似度。这些分子的RDKFP相似度在0.833到0.985之间,说明它们在传统指纹意义上也高度相似。可以观察到,这些分子共享多个关键官能团,包括烷基卤化物(氯代)、叔胺、酮基以及芳香环结构,并且都含有噻吩骨架。值得注意的是,第一排中第二个分子与查询分子的唯一区别在于氯原子在骨架上的连接位置不同,因此指纹相似度最高。整体上,这些结果表明:通过在大规模无标签分子数据上进行对比学习,MolCLR能够将分子嵌入到既具有代表性又“化学上合理”的特征空间中,从而区分和组织分子间的结构与性质相似性。
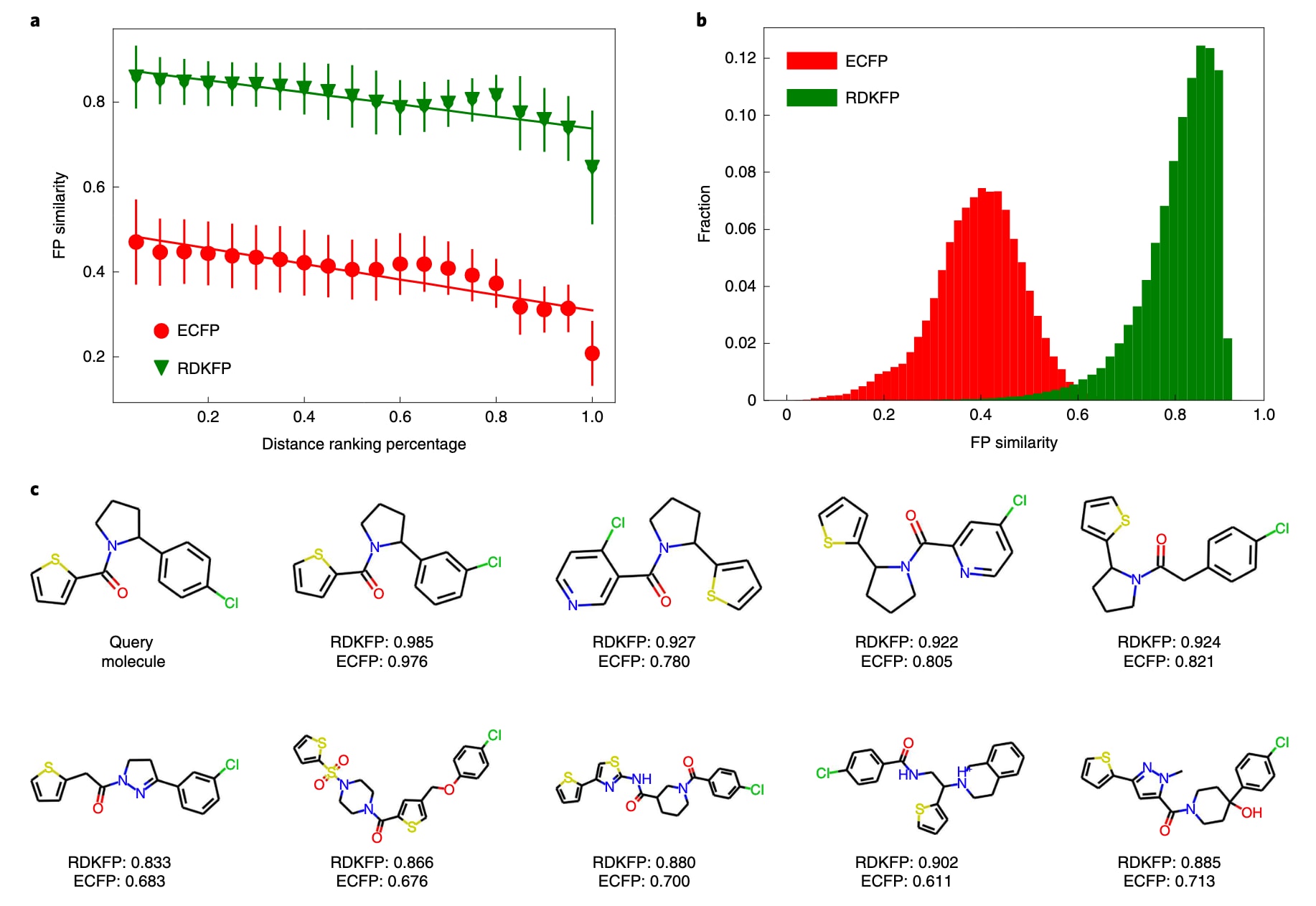
图4 | 展示了基于查询分子(PubChem ID 42953211)对比MolCLR学习的表示与传统指纹。 a,随着MolCLR表示空间距离增大,ECFP和RDKFP相似度的变化趋势。b,与查询分子相比时,ECFP和RDKFP相似度的分布。c,查询分子及其在MolCLR表示空间中最近的九个分子,并标注了对应的RDKFP与ECFP相似度。
3 总结
该研究系统地探讨了用于分子表征的自监督学习方法。具体而言,提出了基于GNN的MolCLR框架,并设计了三种分子图增强策略:原子掩蔽、键删除和子图移除。通过对增强后得到的正样本对与负样本对进行对比学习,MolCLR能够结合通用GNN骨干网络学习到信息丰富的分子表示。实验结果表明,经MolCLR预训练的GNN模型在多种分子基准任务上取得了显著性能提升,相比纯监督学习训练的模型具有更好的泛化能力。MolCLR学习到的分子表示不仅在小样本分子任务中表现出良好的可迁移性,同时也展现出在庞大化学空间上的强泛化能力。
未来仍有许多值得探索的方向。例如,引入更强大的GNN骨干网络(例如基于transformer的GNN架构)有望进一步提升分子表征能力。此外,对自监督学习得到的分子表示进行可视化与可解释性分析也是一个非常重要且有意义的方向,这将有助于研究者更深入理解化学分子特征,并更好地服务于药物发现过程。