NMI 2023 | 基于SMILES的分子从头生成:课程式训练与深度强化学习的能力边界探索
今天介绍的是发表在**《Nature Machine Intelligence》**上的一项研究,工作围绕基于SMILES的深度强化学习分子生成方法展开,系统评估其在分子从头设计中的能力边界。研究不仅探讨这些模型在缺乏训练示例时是否仍能外推生成具备目标性质的分子,也深入分析训练数据组成与SMILES表示方式对生成分子质量和多样性的影响。结果显示,虽然通过奖励函数可以一定程度上控制生成分子的性质分布,但标准方法在训练分布边缘往往失效,容易退化为训练数据的再现。为解决这些问题,作者提出了课程式的迭代优化流程rIOP,在简单与复杂优化任务中均能显著提升生成质量与多样性,并优于现有的课程学习方法ReCL。研究同时指出SMILES表示的固有限制,强调未来更高维、结构化分子表示的重要性。

获取详情及资源:
0 摘要
深度强化学习方法已被证明在分子从头设计中具有潜在的强大能力,其中以循环神经网络为基础的技术最为常用。该研究考察了当训练数据中几乎没有具备目标性质的分子示例时,此类循环神经网络方法的行为表现。结果显示,尽管通常仍能实现针对性的分子生成,但生成分子的多样性往往下降,且难以控制生成分子集合的组成。为缓解这些问题,研究者提出一种受课程学习启发的循环迭代优化流程,能够在已见与未见的分子特征条件下进行生成分子的优化,并允许用户控制对于某一分子特征的探索或利用。借助这一方法,可以生成更加具体且多样的分子集合,在相同样本量下,其骨架数量可达到传统方法的18倍。然而,这些结果也揭示了一维分子表示在该领域中的重要局限。研究发现,某一分子优化任务的成败在很大程度上取决于所选用的SMILES表示方式。
1 引言
研发一种新药是一个复杂且困难的过程,在多个阶段都可能失败。若能够在早期获得质量更高的先导化合物,并生成具备特定性质的新分子,将有助于降低成本、提升研发速度并提高成功率。理论上,在明确靶点与所需分子特征后,应当在整个类药化学空间中寻找潜在分子;然而,类药化合物的可合成数量约为
生成对抗网络也被应用于从头药物设计,并与强化学习结合后可生成多样性高且具备目标性质的分子库。随着自然语言处理技术的发展,transformer模型也被用于SMILES生成,并表现出稳定甚至更优的性能。与transformer类似,循环神经网络因能够处理序列中的长期依赖而在分子生成中广受青睐。基于RNN的模型在采用与语言模型相同的训练策略时,能够生成覆盖更广化学空间的SMILES,特别是结合随机化SMILES时性能更佳。早期方法通常先利用RNN训练一个通用SMILES语言模型,再在包含目标性质的小型数据集上微调。强化学习同样是分子生成中的重要方向,理论上能够在没有明确示例的情况下生成具备指定性质的分子。真正成功的分子设计往往需要产生具有全新性质组合的分子,而这些分子在现有数据集中通常不存在,因此强化学习必须具备外推能力。然而,这些方法能在多大程度上完成外推仍不明确。此外,强化学习还需同时在复杂分子特征空间中实现探索与利用。
基于已有的on-policy强化学习框架,该研究考察了当训练数据中相关特征极少甚至缺失时的优化表现,并系统测试了不同性质在训练集中占比变化下的优化极限。结果显示,强化学习能够在一定程度上外推,但生成分子往往缺乏多样性,且难以满足复杂分子特征的组合要求。在此基础上,研究者提出一种受课程学习启发的优化流程,使生成分子能够同时满足复杂且未见过的性质组合,并保持良好的多样性。研究还指出,SMILES这一一维表示本身存在重要局限,其选择方式会显著影响分子优化任务的成败。
2 结果和讨论
深度强化学习分子生成模型在分子性质优化中具有强大能力,但其有效性高度依赖于输入的训练数据。研究首先展示了此类模型能够针对特定性质进行优化,但优化范围通常被限制在该性质在训练数据中所覆盖的取值区间内。同时也表明,这些方法可以生成训练集中不存在的分子特征组合,而训练数据的表示方式会直接影响生成分子的组成结构。通过系统增加训练集中具备目标特征的分子比例,研究进一步揭示了训练数据表征对于生成结果的影响。
为突破训练数据表征带来的限制,研究者提出一种受课程学习启发的循环迭代优化流程rIOP,用于优化训练中占比极低的性质,并可将生成分子引导至复杂性质组合,这在传统REINVENT框架下难以实现。同时,该流程允许控制生成分子库的多样性。总体而言,这些研究结果突出了深度强化学习模型在单参数优化任务中的能力与局限,理解这些特点对于在实际药物研发流程中将其扩展到多参数优化至关重要。

图1|生成分子库的分布 a,b分别展示cLogP(a)与氢键受体数量(HBA)(b)优化过程中生成分子库的性质分布。图中每条曲线对应一个模型代理在特定奖励区间下生成的分子集合的性质分布。两幅图均显示,优化只能在限定范围内实现(例如HBA的0–20区间),一旦超出该范围,优化即告失败,模型无法生成满足要求的分子,最终仅再现训练数据的分布。
2.1 对生成分子库的控制
从头设计的深度强化学习工具通常希望生成在单一或多种性质上具有可控分布的新分子。已有研究利用REINVENT与ReLeaSE表明,可以让生成分子在特定性质上产生偏移,例如疏水性、熔点或针对DRD2的预测活性。为测试REINVENT的优化能力,该研究调整了cLogP以及氢键受体数量(HBA)的奖励区间。图1a展示在cLogP奖励范围从−15到20的条件下,生成分子的性质分布。结果显示,奖励范围确实能够控制生成分子分布的位置,但在极端奖励区间(如cLogP介于−15到−10)时优化失败,生成分子的分布并未移动,而是完全再现了训练数据的分布。对于HBA的测试也出现类似现象(如图1b所示)。在0–20的范围内能够实现优化,但当目标设定为生成HBA≥20的分子时,模型同样无法实现偏移,最终的分布仍类似训练集,其中包含多个HBA超过30的分子。
补充材料第二部分中提供了完整的生成分子集合、奖励区间以及分子示例。研究推测,这种失败源于在极端条件下,先验模型生成的分子几乎无法获得任何奖励。在理论上,如果训练时间无限延长,模型最终会随机生成一次可获正奖励的SMILES,但由于训练数据对应性质在极端区间的表征不足,优化难以有效开展。优化过程失效后,模型会不断生成训练中出现过的相同SMILES,使最终结果退化为训练数据的再现。
2.2 关于比例表征的影响
前文已指出,当目标性质在训练数据中占比极低时,深度强化学习分子生成模型的优化可能无法实现。为系统评估这一问题的普遍性,研究进一步测试了模型在训练数据内部与外部性质区域生成分子的能力。为此构建了多套训练集,使具备目标性质的分子在其中占比不同。扩展数据表1显示,对于所有测试性质与所有占比条件,REINVENT均能实现优化,且生成分子的性质分布相较训练数据均朝着目标性质方向偏移。这表明,对于这些性质,模型能够从训练数据中邻近的结构–性质关系中学习,即便训练集中不存在明确示例,也能够进行外推。
扩展数据表1同时显示,生成分子库的组成明显受训练集中目标性质占比的影响。例如,虽然在所有条件下都能生成HBA大于八(奖励阈值)的分子集合,但当训练集占比更高时,在相同奖励函数下,生成分子的HBA值也会提高。0%占比条件下生成分子的平均HBA约为九,而在10%占比时平均值提升至20。对所有性质而言,都能观察到类似的趋势(详见补充材料第三部分)。
研究推测,生成分子的变化趋势反映了训练数据本身的分布特性。例如,当目标性质值(如HBA=5)的占比提高时,若训练数据在该性质区间的结构多样性增加,则生成的分子库也会更具多样性;反之,若训练数据在此区间高度相似,则生成分子的多样性也会减少。因此,对大多数性质而言,较低占比往往对应生成分子库的较低多样性。综上,模型能够生成训练中未出现过的分子性质组合,但生成集合的组成仍受训练数据中目标性质的占比强烈影响。因此,根据具体应用场景,应当选择合适的训练占比;然而,当目标是生成完全未见过的分子特征(0%占比)时,传统方法无法让用户掌控生成分子库的组成。
2.3 基于课程学习的生成分子库控制
前文已展示,尽管训练数据中不存在目标性质的分子(扩展数据表1),模型仍能够生成具备这些未见过特征的分子;然而,在性质分布的极端区域(图1)这一能力会受到显著限制。生成分子库的组成取决于训练集中具备目标性质的分子占比。基于先前研究的结论,可以推测当训练数据中目标性质的表征更丰富时,模型往往能够学习到更广泛的结构–性质关系,从而生成更具多样性的分子集合。
为进一步提升深度强化学习生成方法的有效性,研究提出一种受课程学习启发的新方法,即rIOP。该方法能够在优化过程中,让深度强化学习模型针对已见与未见的分子特征生成尽可能多样的分子。同时,它还能支持模型完成标准方法难以实现的复杂优化任务,从而在生成质量与性质满足度之间取得更好的平衡。
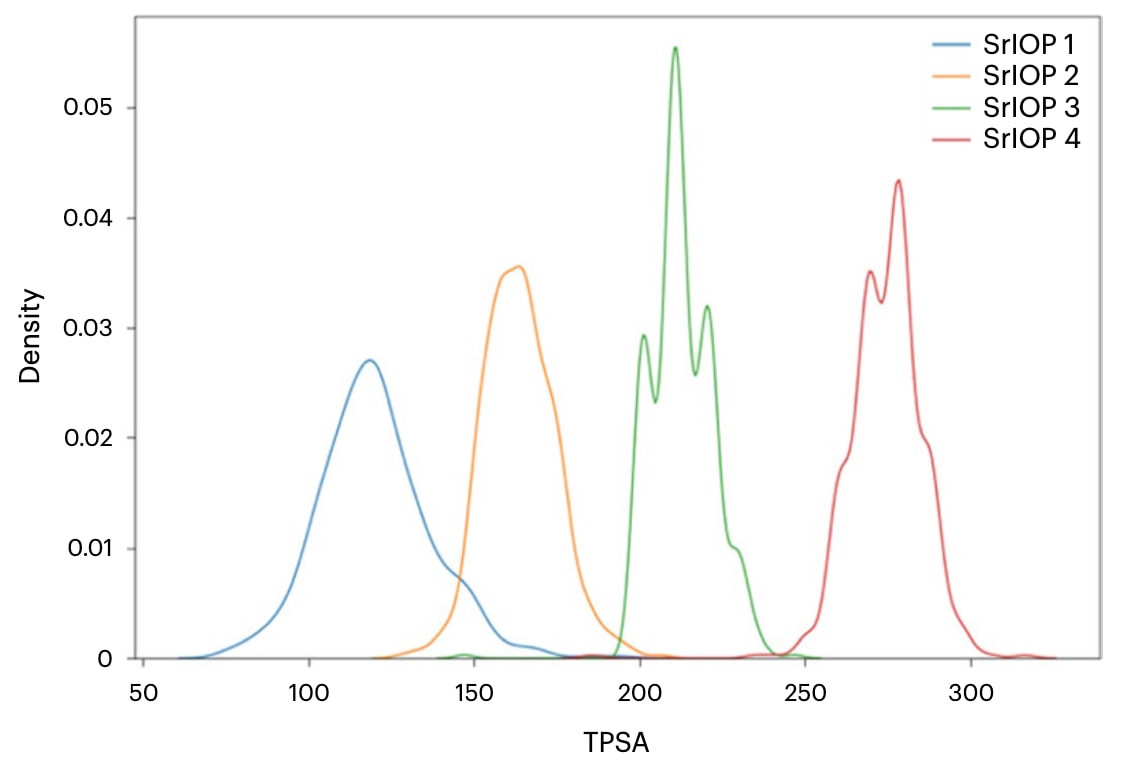
图2|分子TPSA分布 图中展示在rIOP流程中训练得到的各阶段代理(SrIOP 1–3)以及最终代理(SrIOP 4)所生成分子的TPSA分布。可以看到,TPSA分布在逐步迭代中不断向目标性质区间移动。各步骤对应的TPSA奖励范围为:100–150(SrIOP 1)、150–200(SrIOP 2)、200–250(SrIOP 3)以及250–300(SrIOP 4)。
2.3.1 迭代优化流程以提升多样性
为展示rIOP如何提升简单优化任务中的分子多样性,研究以拓扑极性表面积(TPSA)为目标,分别采用单模型SrIOP与REINVENT的标准实现进行比较。尽管标准方法能够生成满足该范围的分子,但预期SrIOP在多样性方面会表现更优。由于TPSA的调整属于较为简单的优化任务,因此在训练当前prior时,仅对上一阶段的代理进行采样(方法部分“rIOP”章节提供了细节)。
图2展示了SrIOP流程的各个步骤,以及每一步生成分子性质分布随奖励函数变化的情况。为衡量生成分子库的多样性,研究统计了每一轮产生的唯一Murcko骨架数量。可以看到,随着SrIOP逐步推进,骨架数量呈下降趋势。这一现象符合预期,因为随着性质值逼近TPSA分布的极限区间,可实现该性质的结构组合逐渐减少。
扩展数据表2显示,在最终阶段(SrIOP 4),SrIOP生成的骨架数量约为REINVENT的18倍(55个对3个)。此外,在相同500个采样分子中,SrIOP生成的满足奖励条件的分子更多(494对476)。扩展数据表2还给出了生成集合的内部多样性,以及生成分子中来自训练数据的比例。分子示例链接见“数据可用性”部分。
2.3.2 用于多样性控制的迭代优化流程
若希望从头设计工具真正发挥作用,生成分子的特异性必须可控。前文已经说明,训练数据中目标性质的表征会影响生成分子集合的组成,从而影响其特异性。此外,结果也展示了SrIOP在简单优化任务中能够提升生成分子的多样性。为进一步验证这一点,研究在训练数据不含高QED分子(QED < 0.8)的条件下,尝试生成类药性良好的分子(QED > 0.8)。
图3展示了在有无多样性过滤条件下生成高QED分子的情况。图3b显示,启用多样性过滤后生成分子的分布更为宽广。在500个采样分子中,SrIOP生成了490个满足奖励条件的分子,而REINVENT仅为317个。在490个分子中,SrIOP生成了22种骨架;标准方法则从317个分子中生成了130种骨架。然而,当启用多样性过滤后,SrIOP的表现有显著提升:在301个生成分子中骨架数量达到297种。REINVENT在启用多样性过滤后也有变化,其236个分子中包含234种骨架。
这些结果表明,通过SrIOP可以生成特异性高(未启用多样性过滤)或多样性高(启用多样性过滤)的分子库;相比之下,REINVENT只能生成多样化的分子,无法让用户控制生成集合的组成。数据可用性部分提供了各生成数据集的示例链接。
SrIOP为用户提供了更强的分子库特异性控制能力。例如,若希望“利用”某一性质,未启用多样性过滤的SrIOP会返回极为集中且完全符合奖励特征的分子;相反,当需要“探索”性质空间时,启用多样性过滤即可获得高度多样的分子库。该例中选择的是相对简单的优化任务,因为提升QED的方法较多,因此预期标准方法表现不会太差。然而,SrIOP在特异性与多样性两方面仍均优于REINVENT,且预计在更复杂的优化任务中,该性能差距会进一步扩大。
2.4 与其他课程学习方法的比较
课程学习长期以来被用于解决多种复杂的机器学习问题,但在深度强化学习的分子生成领域中,其应用依然有限。目前仅有REINVENT的作者提出过一种类似方法,用于应对复杂的优化任务,该研究称其为ReCL。
2.4.1 用于复杂任务的迭代优化流程
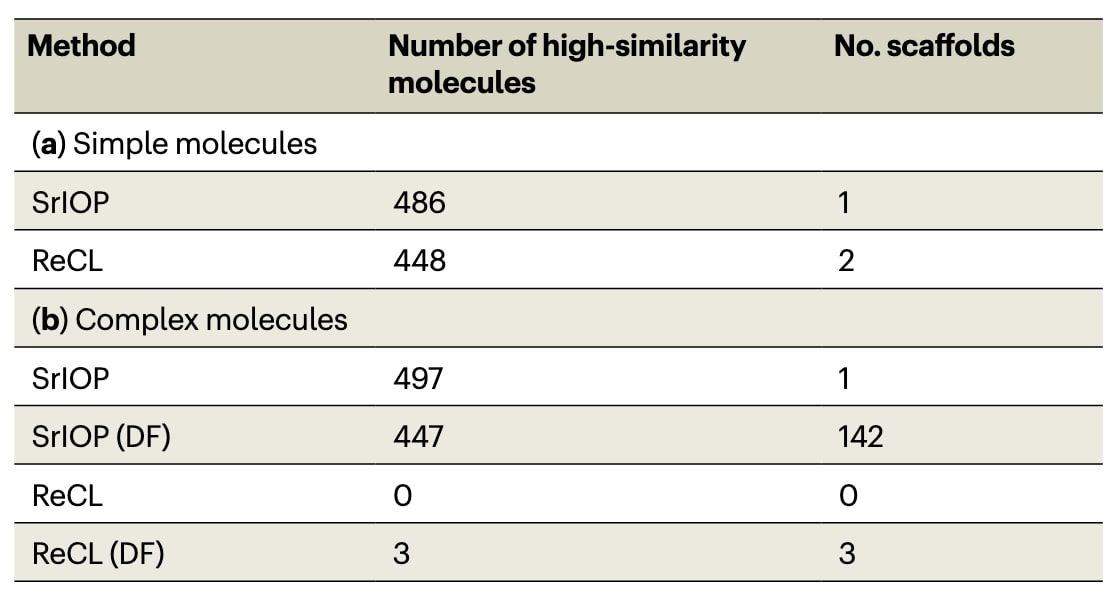
深度强化学习分子生成模型的常见应用之一,是生成与某一目标结构相似的分子。在此类场景中,训练数据中往往几乎不存在与该目标结构接近的示例。为评估SrIOP在这一实际任务中的有效性,研究利用它生成与目标分子相似的结构(扩展数据图1),并逐渐提升任务难度。表1a显示,当目标结构较为简单(扩展数据图1a)时,可以生成与目标完全相同的分子。SrIOP与ReCL的表现均良好,但SrIOP生成Tanimoto相似度介于0.9至1.0的分子更多(486个,ReCL为448个)。
然而,当目标结构更复杂时(扩展数据图1b),ReCL的性能明显下降(扩展数据图2b,c),无法生成任何高相似度分子(Tanimoto相似度>0.7)。相比之下,SrIOP几乎在全部500个样本中都生成了与目标结构高度相似的分子(497个)。
SrIOP的另一优势是在复杂任务中仍能控制生成分子的多样性。表1显示,在不启用多样性过滤时,497个分子均由同一骨架构成;但启用多样性过滤后,SrIOP在447个分子中生成了142种骨架。该示例展示了SrIOP在处理复杂奖励函数时的显著优势,而类似课程学习方法(ReCL)在此类任务中常常失败。此外,通过加入多样性过滤,SrIOP还能让用户灵活调控生成分子库的多样性。
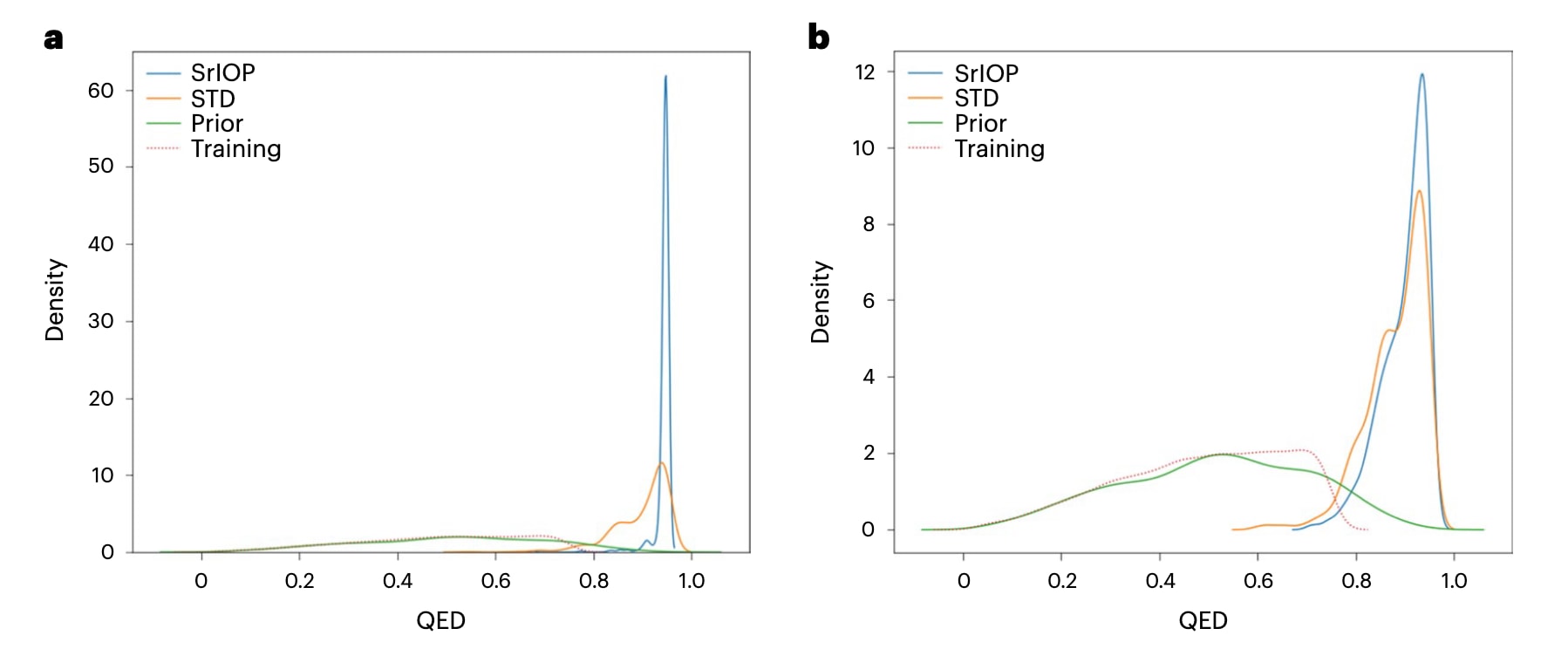
图3|SrIOP与REINVENT在有无多样性过滤条件下的比较 a,b展示SrIOP与REINVENT(STD)在未启用(a)与启用(b)多样性过滤情况下生成类药性分子时的对比。图中给出了高QED分子(QED > 0.90)的生成分布。训练过程中仅使用低QED分子(QED < 0.8),随后在每一步SrIOP迭代中逐步提升QED奖励区间。两种方法均能生成目标性质的分子,但在两种条件下SrIOP生成位于目标区间的分子数量更多。
2.5 基于SMILES的分子生成器的局限性
该研究评估并提出了新的基于SMILES的深度学习分子生成工具。此类模型通过学习生成三维分子的二维连接关系,并以一维SMILES串表示。由于其结构简单、训练迅速,SMILES模型广受使用。然而,SMILES本身存在若干固有限制:它无法表达三维结构信息,只记录原子连接方式;且同一分子可对应多个不同的SMILES串。尽管规范化SMILES提供了统一生成方法,但已有研究表明利用随机SMILES训练能提高模型覆盖率并减少过拟合。
缺乏结构信息与表示冗余,使得基于SMILES的模型难以充分理解分子间的真实化学与结构关系,因为SMILES之间的相似度与分子真实结构的相似度常并不一致。这一局限性在该研究中表现得尤为明显。例如,当尝试生成结构愈加复杂的分子时,模型性能高度依赖于用于表示各子结构的具体SMILES串。研究发现,为使模型表现最佳,不同SMILES串之间的差异必须尽可能小。换言之,SMILES的选择直接影响模型的生成能力。
表1|生成分子集合的构成情况
研究尝试生成包含特定子结构的分子,并在强化学习中使用多个不同SMILES来表示同一子结构。结果显示,在所有测试中约有四分之一的子结构生成任务失败(即最终未能生成含目标子结构的分子;见扩展数据表3)。但对于每次失败,都存在另一套替代SMILES可实现成功生成。这种“同分子不同SMILES、性能差异巨大”的现象甚至出现在几乎相同的分子之间,突显模型成功与否更多取决于SMILES选择,而非分子本身的复杂性。
图4展示了两个结构相似的分子(A与B)中,最终代理所生成的含目标子结构的SMILES数量与骨架数量。对于分子A,ReCL与SrIOP均至少有一次未能生成目标子结构,且两者都在最终步骤失败(补充材料第7部分)。但对于分子B,无论选择哪一版本的SMILES,都能成功生成目标子结构,尽管其结构与分子A的中间子结构和最终子结构几乎相同。这进一步说明SMILES选择对模型性能影响远大于分子本身的复杂性。
SMILES的选择也会极大影响生成分子的多样性。例如,分子B的SrIOP系列3(图4c)在500个采样分子中生成了超过250种独特骨架,而其他系列的SMILES在ReCL与SrIOP中仅产生50至150个骨架。类似的性能波动在所有测试分子中均有出现(补充图13)。
虽然SELFIES与Deep-SMILES试图解决SMILES的一部分问题,但对于机器学习而言,更高维且包含结构信息的表示方式可能更适合用于分子生成与优化。
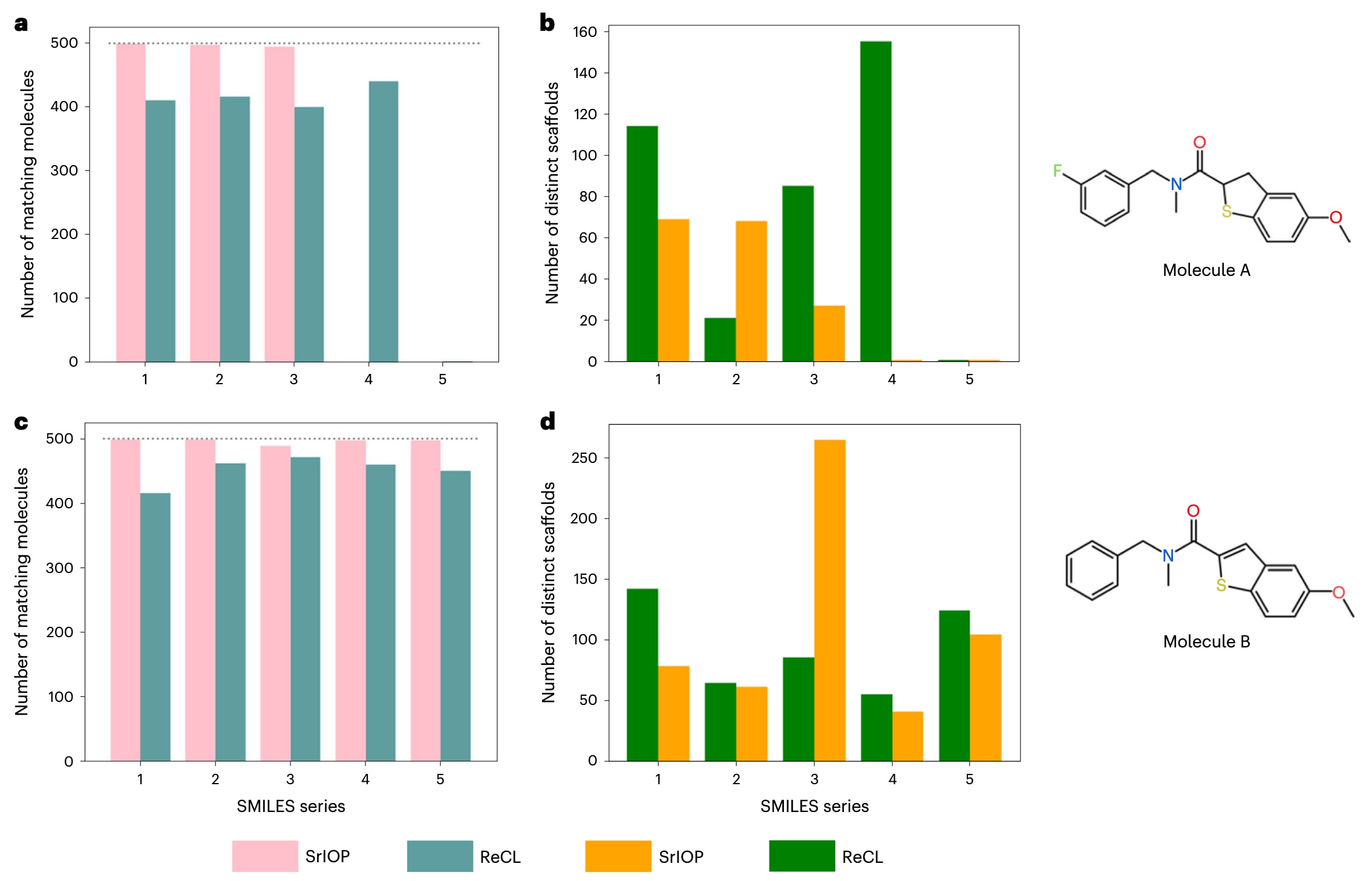
图4|SMILES选择对ReCL与SrIOP子结构生成性能的影响 a–d为每个目标分子生成的五组不同的中间SMILES,并在优化过程中使用。每一步使用的不同SMILES版本均代表同一分子。a,c显示最终代理生成的包含目标子结构的SMILES数量(共采样500个分子);b,d展示成功样本中不同骨架的数量。a,b与c,d分别对应目标分子A与B。图中可以看到,SMILES的选择会直接影响优化性能以及生成分子的多样性。例如,对于分子A,尽管各步骤的中间SMILES都代表完全相同的结构,两种方法仍至少各有一次未能生成目标子结构(即无任何分子包含所需子结构)。
3 结论
该研究考察了深度强化学习分子设计方法在多大程度上能够突破训练数据所覆盖的化学空间,以及训练数据组成如何影响生成分子集合。结果表明,通过调整奖励函数可以在一定范围内控制生成分子的性质分布;然而,标准方法(REINVENT)在训练数据分布的边缘区域容易失效。研究发现,可以生成训练集中未出现过性质的分子,但目标分子特征在训练数据中的表征程度仍会显著影响最终生成分子库的性质分布。此外,标准方法在生成分子的组成与多样性方面缺乏有效控制能力,而基于SMILES的一维表示方式本身也存在固有局限。
为缓解这些问题,研究提出一种新的课程学习方法rIOP,当训练集中目标性质的示例极少或缺失时,能够显著提升生成分子的多样性。利用该方法,可以生成与多种未见过目标结构相似的分子,并在性能上优于其他课程学习方法(ReCL)。文中进一步介绍了单模型SrIOP与双模型DrIOP,它们能够在简单与复杂优化任务中为用户提供对生成分子多样性的灵活调控能力。
此外,在目标结构生成过程中使用不同SMILES表示的实验表明,SMILES的选择会直接影响基于SMILES的模型能否成功生成目标分子以及其整体表现。与所有一维分子表示方法一样,由于缺乏直接的三维结构信息,该方法在本质上仍受表示能力的限制。