Commun. Biol. 2025 | MoleculeFormer: 一种用于分子性质预测的 GCN-Transformer 架构
今天介绍的这项工作来自 communications biology。该研究围绕分子性质预测中“局部–全局信息难以统一建模、三维结构与键级信息利用不足以及模型可解释性和抗噪性较弱”等关键问题,提出了 MoleculeFormer 这一基于 GCN–Transformer 的多尺度可解释分子预测模型。该模型通过原子图与键图双通道编码,引入图表示节点和注意力机制,结合 EGNN 融合三维几何结构,并与精心筛选的分子指纹进行协同建模,在同一框架下统一了局部化学环境、键级拓扑、电性信息与全局三维构象。相较传统仅依赖单一分子图或指纹的模型,MoleculeFormer 多源特征的分层融合设计、具备物理约束的等变图编码、可视化的原子/键注意力解释,以及系统化的指纹组合优化策略。实验结果表明,该模型在 MoleculeNet、表型筛选及 ADME 等 28 个数据集上整体性能优于或媲美现有图深度学习方法,在大规模数据、噪声标签和跨任务场景下均表现出良好的稳健性与泛化能力,为药物发现与分子设计提供了一种兼具预测精度与化学可解释性的通用解决方案。

获取详情及资源:
- 📄 论文: https://doi.org/10.1038/s42003-025-09064-x
- 💻 代码: https://github.com/qinmingyuan-hub/molecular_graph
0 摘要
人工智能在药物发现中日益重要,尤其是在分子性质预测方面。图神经网络(Graph Neural Networks, GNNs)能够将分子结构建模为图结构,利用分子的结构数据有效预测分子性质和生物活性。然而,分子特征优和模型集成仍然是挑战。为解决这些问题,该研究提出了 MoleculeFormer,一种基于 GCN-Transformer 架构的多尺度特征融合模型。该模型采用独立的图卷积网络(GCN)和 Transformer 模块,从原子图和键图中提取特征,同时融入旋转等变性约束以及分子指纹等先验信息。通过结合局部与全局特征,并引入对旋转和平移不变的三维结构信息,模型能够更全面地理解分子表示。在 28 个数据集上的实验结果表明,MoleculeFormer 在多种药物发现任务中表现稳健,包括疗效/毒性预测、表型筛选以及 ADME 评估。注意力机制的集成提升了模型的可解释性,同时在噪声条件下依然保持良好性能,使 MoleculeFormer 成为一种高效且具有良好泛化能力的分子预测解决方案。
1 引言
近年来,基于人工智能的方法显著提升了小分子性质预测的准确性与效率。这些方法利用机器学习和深度学习技术解析分子结构与其性质(包括理化性质及其他相关分子特征)之间的关系,从而实现对分子生物活性的预测。随着计算能力的不断增长以及大数据资源的广泛可得,人工智能方法能够更加全面且快速地探索分子信息空间,揭示大规模数据中隐藏的结构模式与规律。此类方法包括但不限于 QSAR 和 QSPR,它们能够有效处理分子结构与多种性质之间的复杂关联,使人工智能成为药物发现、材料设计、环境科学等领域的变革性工具。
小分子特征的嵌入仍然是关键步骤,因此如何有效提取分子相关特征成为重要研究方向。该过程主要包括分子级表示和原子级表示,这两类方法通常以 SMILES 为输入,最终输出整个分子的特征表示。
分子级表示依赖先验知识,通过分子描述符或分子指纹来表达分子特征。常见的分子描述符包括理化描述符、拓扑描述符与量子化学描述符。然而,部分与特定性质具有明确关系的描述符在缺乏相应先验知识的任务中表现可能不佳。分子描述符基于数值计算提供丰富的理化信息,而分子指纹则采用更结构化的编码方式,通过识别分子的结构片段、官能团或子结构生成二进制或哈希编码。由于不同指纹的编码逻辑与表征内容存在差异,通常会组合多种指纹用于描述同一分子结构。上述指纹可搭配传统机器学习算法(如朴素贝叶斯、支持向量机、随机森林、XGBoost)或深度学习方法进行建模。已有研究比较了多种分子指纹类型,并探讨如何选择最优的分子表示方式。尽管分子描述符和指纹都能用于性质预测,但二者均会丢失一定的结构信息,并较大程度依赖先验知识。
原子级表示则在原子层面对分子进行编码,将 SMILES 转换为图结构(原子为节点、化学键为边)。原子与化学键特征(如原子序数、价电子、键类型等)会被嵌入图结构,节点通过聚合邻域信息实现表示更新。GCN 通过图卷积实现特征聚合;GAT 则通过为不同邻接节点分配注意力权重实现精细的结构编码;图 Transformer 则将自注意力机制视为输入序列元素间的软邻接矩阵。Chemprop 采用定向消息传递神经网络(D-MPNN)提取分子特征,而 HiGNN 通过层次化图卷积捕获更复杂的分子结构。
在最终预测阶段,为保证维度一致性,通常需将多个原子节点的特征聚合为单一分子表示,因此常使用池化操作(如最大池化或平均池化)。然而,池化操作存在粗糙性,会导致大量特征损失,且难以解释其有效性。此外,现有方法在对原子特征进行编码时,往往未充分考虑化学键的邻接关系。事实上,键能不仅影响分子的热力学稳定性(如分解温度、反应活化能),也与分子间相互作用(如氢键强度、π–π 堆叠效应)及动态行为(如振动频率、光化学反应路径)密切相关。因此,将键能参数引入边特征编码,或通过量子化学计算生成与键能相关的派生描述符并纳入图编码范围,有助于提升模型的分子性质预测能力。
图结构表示与描述符/指纹表示的组合通常能够进一步提升模型性能。例如 HRGCN+ 将分子图与分子描述符联合输入改进的图神经网络;FP-GNN 则融合三类分子指纹与图注意力网络,从而增强模型性能与可解释性。
该研究提出了一种基于多尺度特征的可解释分子预测模型。在图编码方面,该研究团队并未采用传统的单分子图结构,而是引入长度为 39 的键图特征,将每条化学键视为节点,利用键之间的连接关系补充原子图中有限的结构表征能力。相较于原子图主要描述原子属性,键图可更准确地描绘键类型、键长、键角等几何与化学信息,这些信息对理解分子的三维构型与化学性质至关重要。例如,有机分子中双键和三键的存在会显著影响反应活性和空间构型,将键图引入图模型可使结构特征提取更加全面,从而提升性质预测准确性。
在图聚合方面,传统的 GCN 和 GAT 采用节点级特征聚合,最终需使用池化将所有节点的特征缩合成图特征,但该过程会造成信息损失且难以解释。受到自然语言处理(NLP)的启发,该研究团队使用 GCN 替代分子图中的位置编码,并将分子图视为句子,通过 Transformer 编码器进行特征提取,因此引入了图表示节点(graph-representation node)。通过分析图表示节点与各原子节点之间的相关性,模型实现了对整个分子结构的有效聚合。此外,结合注意力权重可以明确哪些结构对于分子性质具有更大影响,增强模型的可解释性。
在表征与聚合阶段,该研究团队引入三维(3D)特征用于分子编码,并整合了等变图神经网络(EGNN),确保模型在旋转与平移变换下保持等变性。EGNN 通过在节点更新过程中保持邻近原子间距离不变,使节点特征与三维结构的更新过程具备物理合理性。在此基础上,该模型形成了多维度可解释设计,通过注意力机制结合原子图与键图的特征提取,实现了分子结构的微观可视化。同时,模型融合了分子指纹的先验知识,进一步提升了预测精度与拟合速度(见图 1)。这一多维分子特征提取方式最大程度地挖掘了分子特征信息,从而保证模型在实际应用中的可靠性与准确性。
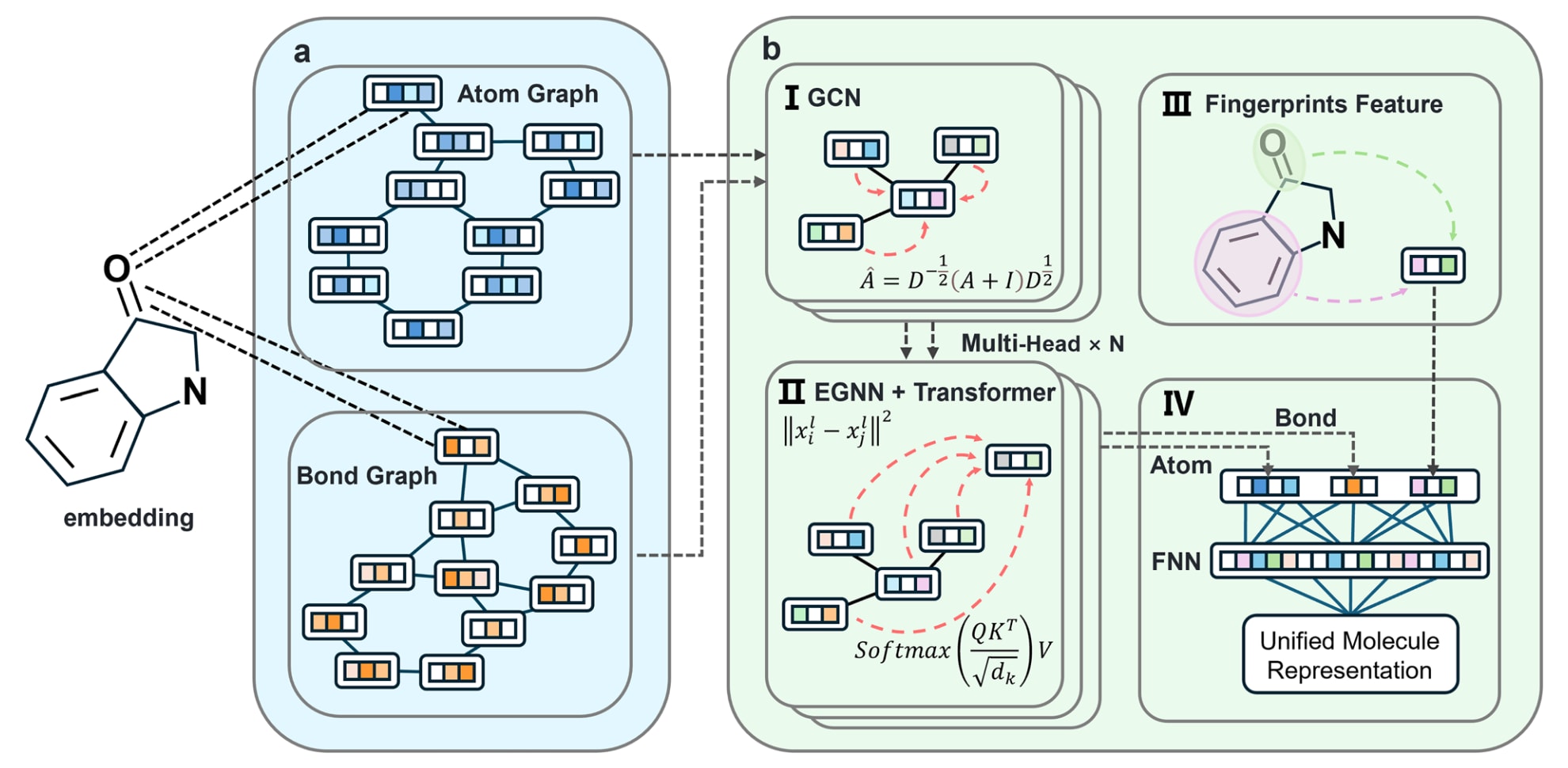
图1 | 展示了MoleculeFormer 的模型结构。 a 将 SMILES 表示分别转换为原子图和键图。b GCN–Transformer 等变编码器及输出模块。I 使用 GCN 进行特征聚合,将图转换为包含局部信息的图结构。II 结合 EGNN 进行 Transformer 编码,输出分子特征。III 将先验知识融入分子指纹编码。IV 通过前馈神经网络(FNN)输出预测结果。
2 结果
2.1 分子指纹组合的选择
为筛选最优的分子指纹,该研究团队基于“权威性、编码完整性及广泛使用程度”三个维度,对化学信息学中常用的八种分子指纹进行了系统评估。为确保实验设计的严谨性,该研究采用控制变量的方法,分别对单指纹、双指纹和三指纹组合进行独立的编码选择实验。具体而言,每一种组合均构建相同结构的前馈神经网络(FNN)模型,仅在输入层根据不同指纹组合的长度调整维度,从而确保性能差异完全源于指纹选择本身。
实验结果显示,分子指纹的选择与任务类型之间存在显著相关性。为深入探究这一现象,该研究分别对分类任务与回归任务进行了统计分析。其中,分类任务包括 MoleculeNet 中的 7 个分类数据集以及乳腺癌数据集中的 14 个分类任务;回归任务包含来自 MoleculeNet 的 3 个数据集以及 ADME 数据集中的 4 个数据集。
图 2 所示结果表明,不同分子指纹在不同任务中的表现存在明显差异。在分类任务中,ECFP 指纹与 RDKit 指纹表现最佳,平均 AUC 达到 0.830。这可归因于分类任务关注对不同类别分子的精确区分,而 ECFP 能细致描述局部结构及原子环境,对于识别不同类别分子的结构差异至关重要。相对而言,在回归任务中,MACCS Keys 指纹表现最优,平均 RMSE 为 0.587。回归任务旨在预测连续值,如分子某些物理或化学性质。MACCS Keys 的编码方式可能包含与这些连续属性高度相关的信息,可有效反映分子关键特征与目标值之间的线性或非线性关系。
在双分子指纹组合实验中,不同任务的最优组合亦存在差异。在分类任务中,ECFP 与 RDKit 的组合仍表现最佳,平均 AUC 达到 0.843,表明它们在提供分类所需的特征方面具有高度互补性。此外,EState 与 RDKit 的组合也表现出相近的预测能力。然而,EState 单独使用时平均 AUC 为 0.783,显著低于最优组合,说明与 EState 相比,ECFP 在分类任务中提供了更具区分度的结构特征。在回归任务中,组合指纹的表现有明显提升,其中 MACCS Keys + EState 的组合最优,平均 RMSE 降至 0.464。EState 指纹强调分子的电子态与原子环境信息,而回归任务需要更全面的分子性质表征,以预测连续变量。MACCS Keys 与 EState 在信息层面高度互补,一个关注关键特征与目标值的关系,一个聚焦电子态及原子环境,因此该组合能为回归模型提供更丰富、全面的特征描述,从而显著提升预测性能。
为了确定不同数据集中表现最优的三种分子指纹,该研究统计了每个数据集中表现排名前三的指纹组合,进而得到分类任务与回归任务中分子指纹的平均表现排名。在分类任务中,各模型性能相对均衡,最优组合为 ErG、ECFP 与 RDKit。ErG 指纹侧重于药效团相关信息,反映化合物的生物活性特征;ECFP 指纹基于原子周围的子结构,体现整体的原子环境;RDKit 指纹基于分子图路径信息,可有效捕捉局部键特征。三者结合可在结构与性质层面提供全面覆盖,是分类任务中区分分子类别的可靠特征来源。在回归任务中,EState 指纹表现最为突出,而 RDKit 指纹表现不佳,未进入排名。EState 指纹基于原子的电子态与拓扑性质,反映分子内部原子的内在状态。许多连续性质(如极性、反应活性等)与原子的电子环境密切相关。例如,分子的溶解性与原子电荷分布及空间构型相关,EState 指纹可有效捕捉这些原子级信息,因此更适合构建分子结构与连续性质之间的定量关系。
值得注意的是,在单指纹预测任务中表现最差的 ErG 与 ECFP 两类指纹,却在回归模型的指纹排名中进入前三。这一现象说明 ErG 与 ECFP 能为其他指纹提供有价值的补充信息,增强模型的特征维度。因此,该研究最终选取 EState、ErG 与 ECFP 作为回归模型的分子指纹组合。在热图中,颜色越深表示性能指标越优。与分类任务相比,分子指纹在回归任务中表现出更大的差异性。EState 指纹在单指纹预测中分别位列分类与回归任务的第四名与最后一名,整体表现弱于其他指纹,但在组合指纹中却展现出显著优势,反映了指纹特征互补性的关键作用。RDKit 指纹在回归任务中的干扰效应较为明显,导致其整体表现不如其他指纹。
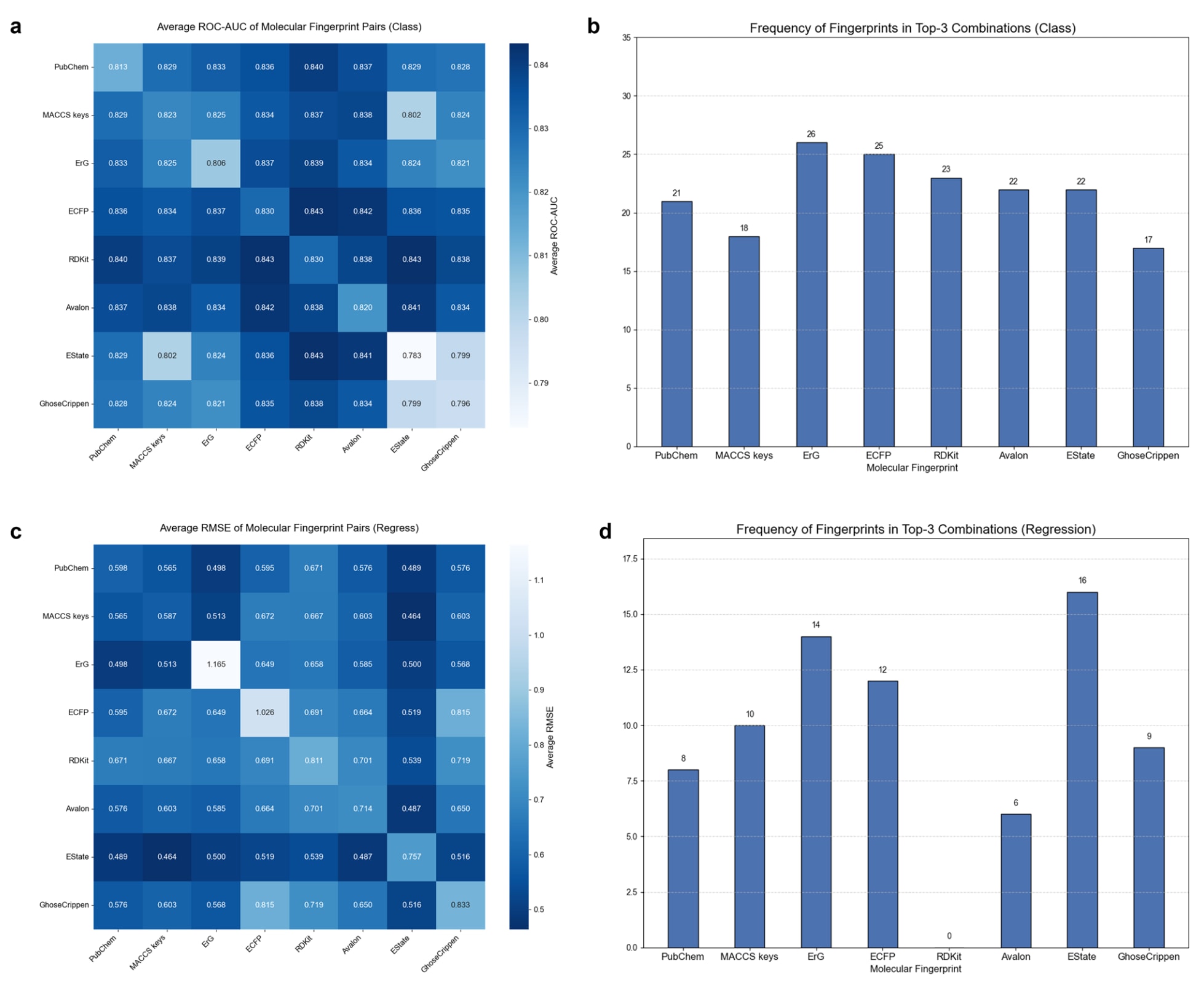
图2 | 展示了MoleculeFormer 分子指纹筛选实验结果。 实验分别采用 1 种、2 种和 3 种分子指纹的组合进行。a 分类任务中双分子指纹组合的热图。b 分类任务中,各数据集表现排名前三的指纹组合中所包含指纹的统计计数。c、d 回归任务的对应结果(分别为热图与统计计数,对应 a 与 b)。
2.2 MoleculeFormer 在 MoleculeNet 数据集上的性能表现
该研究团队使用 Wu 等人提供的公开基准数据集,对 MoleculeFormer 在 10 个广泛应用于药物性质预测的经典基准数据集上的性能进行了系统评估。相关任务涵盖分类和回归两类,数据集采用随机划分或基于骨架(scaffold)的切分方式。这些基准数据集包含单任务和多任务情景,在药物发现领域具有高度代表性与广泛研究价值,因而具有重要的评测意义。该研究采用随机划分和基于骨架划分两种方式对数据集进行切分,每个数据集按照 8:1:1 的比例划分为训练集、验证集和测试集,并在 10 个不同随机种子下独立重复实验,以确保结果的稳健性与泛化能力。
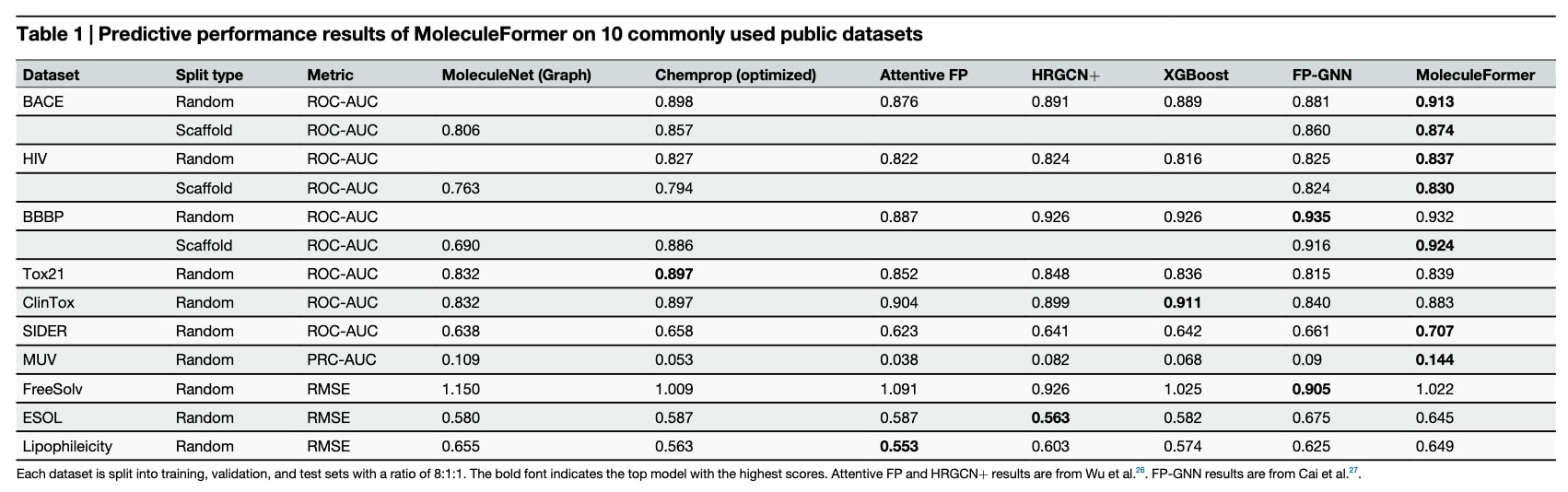
所有数据集均采用 ROC-AUC 和 PRC-AUC 作为评价指标,其中 PRC-AUC 尤其适用于类分布不均衡或关注少数类别的任务。在回归任务中,采用 RMSE(均方根误差)来衡量模型预测的准确性,RMSE 表示预测值与真实值平方差平均值的平方根。如表 1 所示,MoleculeFormer 在 13 个任务中获得了最佳平均性能,验证了该模型的有效性。具体而言,BACE 数据集用于预测分子与 β-分泌酶(BACE)的结合亲和力,是药物设计与分子对接研究中的重要数据集;HIV 数据集用于预测化合物对人类免疫缺陷病毒(HIV)的抑制活性;BBBP 数据集用于评估化合物穿越血脑屏障的能力,对中枢神经系统药物的筛选尤为关键;Tox21 与 ClinTox 数据集主要用于药物毒性评估;SIDER 数据集用于刻画药物与其可能引发的不良反应之间的关系;MUV 数据集用于评估化合物与靶蛋白之间相互作用的预测性能;FreeSolv 用于预测化合物的溶解度;ESOL 数据集用于评估化合物水溶性(logP)的预测精度;Lipophilicity 数据集则用于预测化合物的脂水分配系数(logD)。
表1 | 展示了MoleculeFormer 在 10 个常用公开数据集上的预测性能结果。
在二分类任务中,MoleculeFormer 在 BACE 与 BBBP 数据集上表现尤为突出:在 BACE 数据集上,MoleculeFormer 的 PR-AUC 达到 0.87 ± 0.04,在 BBBP 数据集上,PR-AUC 达到 0.97 ± 0.01,在 ClinTox 数据集上,其平均 PR-AUC 进一步提升至 0.98 ± 0.01。对于 SIDER(PR-AUC = 0.69 ± 0.02)、HIV(PR-AUC = 0.41 ± 0.15)、Tox21 以及 MUV 数据集,PR-AUC 整体偏低,说明这些数据集的分类难度本身较高,并可能存在类别不平衡、分子特征高度复杂与多样等问题。然而,相比其他模型,MoleculeFormer 在这些任务中仍展现出更为明显的性能优势。
值得注意的是,在三个回归任务中,由于训练分子数量相对较少,限制了 MoleculeFormer 注意力机制的完全发挥,但该模型仍保持了较强的竞争力,在相关数据集上的表现依旧稳健出色。图 3 展示了不同模型在输入 10 万个分子(包含三维结构信息)后的训练与推理时间及其参数规模。MoleculeFormer(FP)直接使用分子指纹进行全连接编码,因此在训练和推理阶段具有较高的运行效率;相较之下,MoleculeFormer-Mini 移除了 EGNN 模块,从而显著缩短了训练时间,同时仍保留了 GCN 与 Transformer 的核心结构与特征提取能力,在效率与性能之间取得了良好平衡。
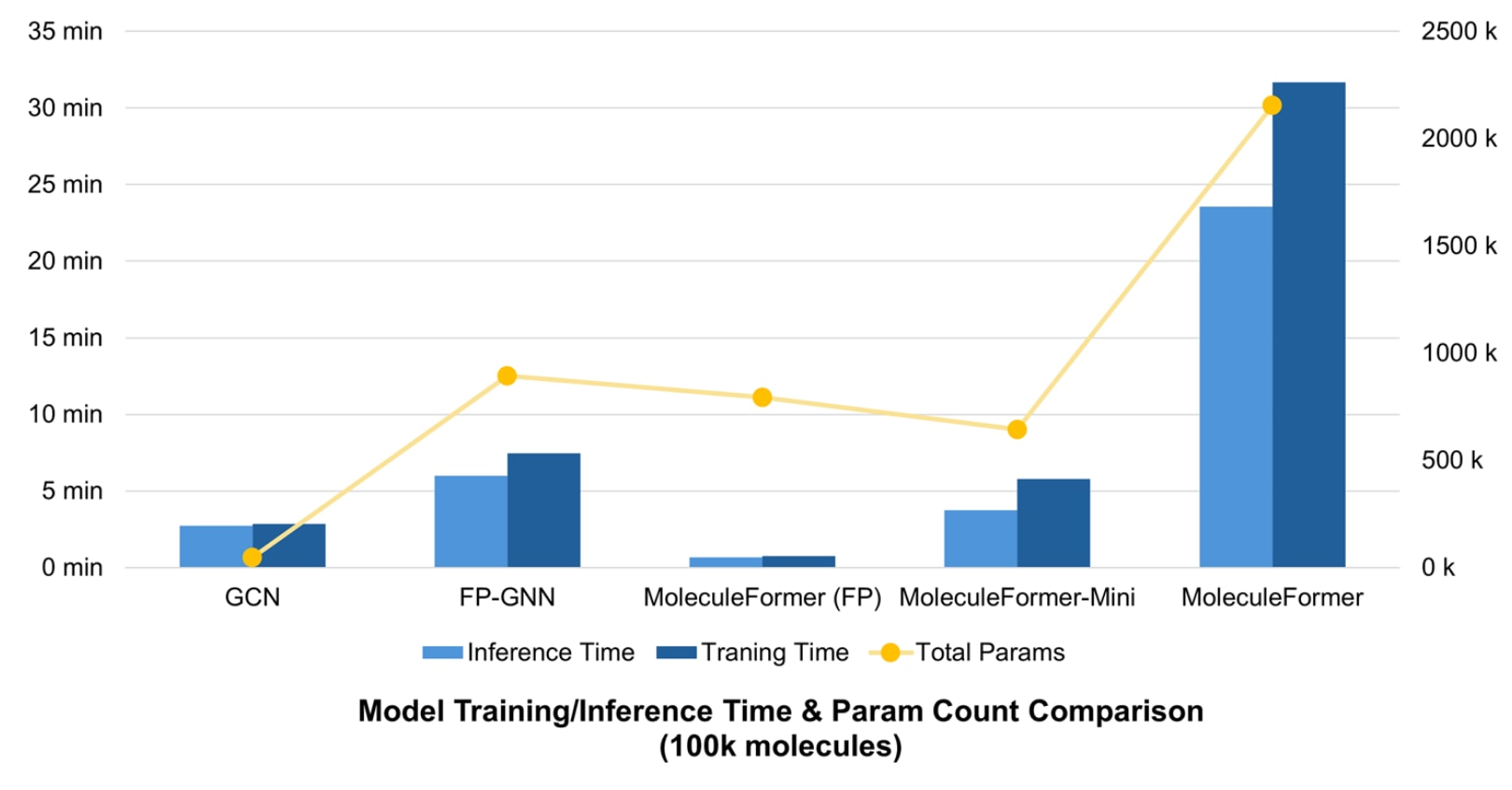
图3 | 展示了针对 10 万个小分子预测任务,不同模型的训练/推理时间与参数量比较。 该图展示了在输入 10 万个小分子(不包括 3D 特征生成与力场优化时间)的条件下,不同模型的训练时间、推理时间以及参数数量的比较结果。为解决大规模数据训练时间过长的问题,该研究团队开发了 MoleculeFormer-Mini 版本。与完整的 MoleculeFormer 相比,MoleculeFormer-Mini 在牺牲少量预测精度的前提下大幅缩短了训练时间,其训练速度甚至快于 FP-GNN。
2.3 MoleculeFormer 在细胞表型筛选数据集上的性能表现
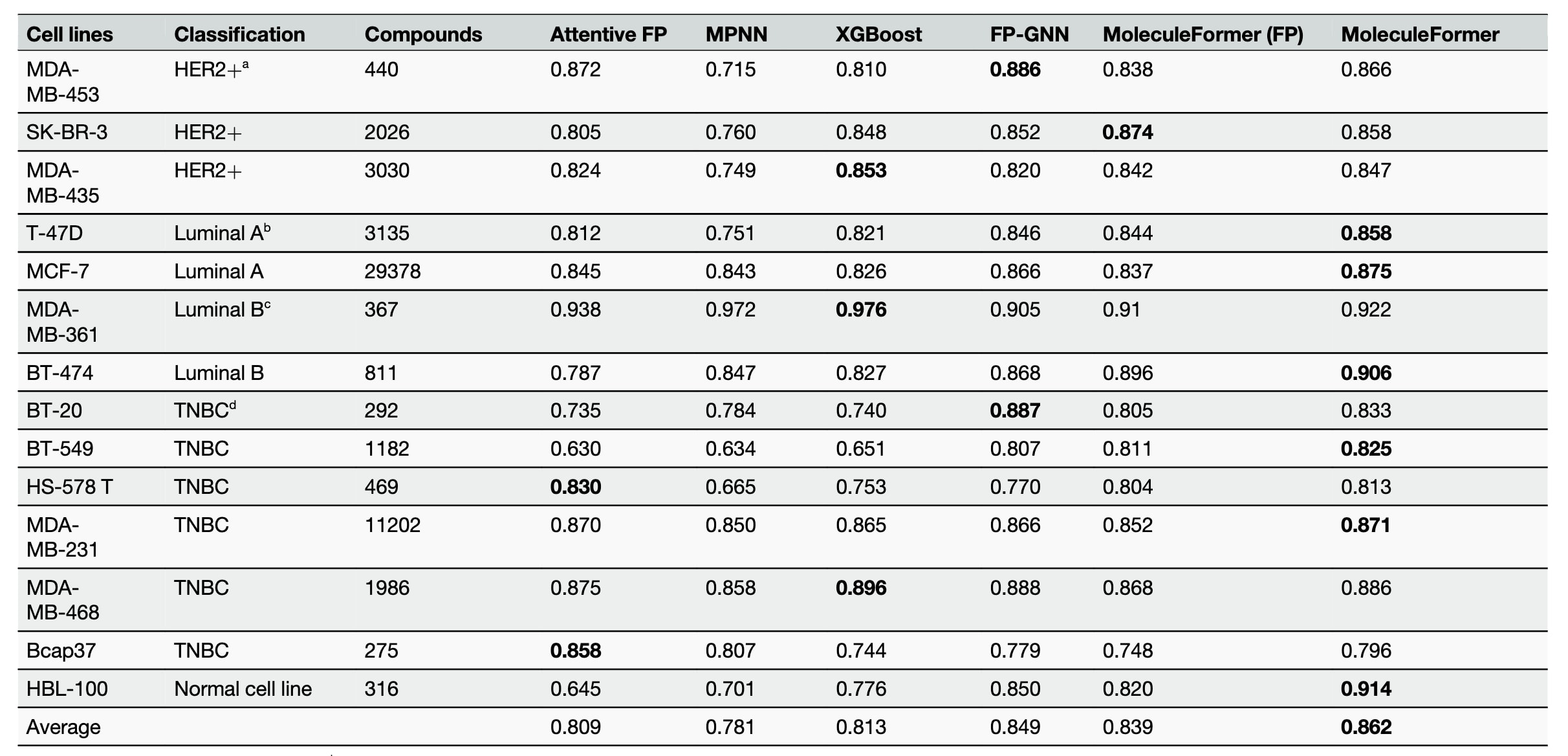
为评估 MoleculeFormer 是否能够捕捉药物的整体效应,并衡量其对真实细胞的综合影响,该研究团队在化合物–乳腺癌细胞表型筛选数据集上进行了实验。该数据集由 He 等人整理,汇总了目前可获得的关于 13 种乳腺癌(BC)细胞系及 1 种正常乳腺细胞系的定量化合物–细胞交互数据。仅保留通过标准指标(IC50、EC50 或 GI50)量化活性的化合物;未具有可靠活性记录的分子均被排除。
根据 Fields 等人和 Ye 等人提出的阈值设定,活性值 ≤ 10 μM 的化合物被划分为活性类,而活性值 > 10 μM 的化合物划分为非活性类;无法分类的样本被剔除。最终数据集中包含 33,757 个活性化合物和 21,152 个非活性化合物,这些化合物均常用于体外抗增殖实验。该研究在上述 14 个细胞系的数据集上对 MoleculeFormer 进行了评测(表 2)。为便于比较,同时纳入了 He 等人发表的五种深度学习模型的结果,包括基于图注意力机制的 Attentive FP 模型、基于消息传递神经网络(MPNN)的模型、先进的指纹–XGBoost 模型、FP-GNN 模型以及 MoleculeFormer 的指纹模块。MoleculeFormer 在相同的表型筛选数据上进行训练与测试,其性能指标与这些参考模型直接对比。如表 2 汇总所示,每个细胞系表现最优的模型均以加粗标注。值得注意的是,MoleculeFormer 在所有细胞系上的平均 ROC-AUC 得分最高,表明该模型在此类表型预测任务中具有优秀的预测准确性与竞争力。
表2 | 展示了MoleculeFormer 在 14 个乳腺细胞系数据集上的预测 ROC-AUC 性能结果,与图深度学习模型的对比。
2.4 MoleculeFormer 的消融研究
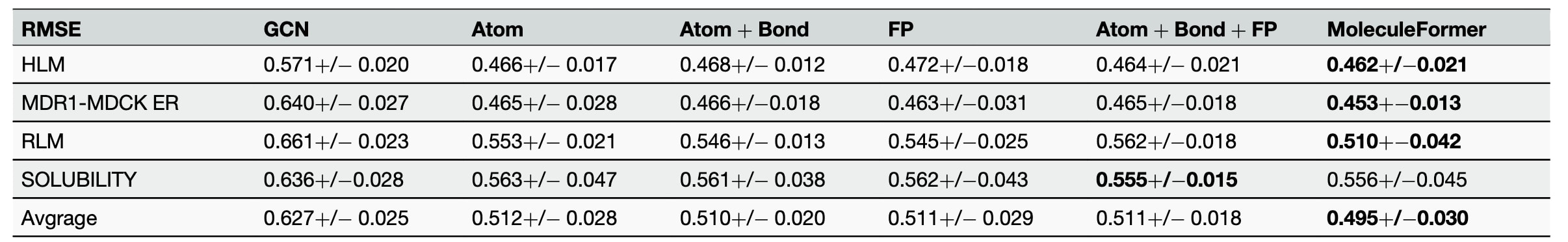
在药物研发中,药物分子的吸收、分布、代谢与排泄(ADME)性质直接决定其疗效与安全性。该研究使用的 Biogen 数据集聚焦于商业可得化合物的 ADME 特性,涵盖结构多样的分子,以模拟真实候选药物的复杂性。该数据集可用于评估 MoleculeFormer 学习广泛分子特征的能力,并用于量化其各组成模块对预测性能的贡献。
在消融实验中,该研究团队比较了六种模型结构:基线图卷积网络(GCN),基于原子图的 Transformer 模型(Atom),原子图与键图联合 Transformer 模型(Atom + Bond),基于分子指纹(FP)的编码模型,融合原子图、键图与分子指纹的 Transformer 模型(Atom + Bond + FP),以及综合原子图、键图、指纹模块与 EGNN 的完整模型,即 MoleculeFormer。实验采用 RMSE 作为评估指标,并通过固定 10 组随机划分种子确保不同数据集间对比的公平性(表 3)。
主要结论包括:
(1)**原子图与键图的协同作用:**两者融合能够增强隐式分子特征的提取能力,并提升预测稳定性。
(2)**多模态特征的互补性:**引入分子指纹进一步强化了模型的表征能力,其与图结构表征之间具有互补效应,有助于构建更加全面的分子表示。
(3)**对三维特征的敏感性:**在涉及分子构象或空间依赖性的任务中,引入 EGNN 模块能够显著提升预测性能,体现其在捕捉三维结构特征方面的优势。
表3 | 展示了MoleculeFormer在 4 个不同数据集上的消融实验结果,评估指标为 RMSE。。
2.5 MoleculeFormer 的抗噪性能
深度学习模型高度依赖数据集标签的准确性,因为错误的标签会导致模型学习到不正确的特征权重。然而,实验数据采集过程中难以避免产生误差,因此模型对噪声的鲁棒性是衡量其质量的重要标准。该研究团队在 HIV 数据集上进行了抗噪实验,将训练集与验证集部分样本的标签进行反转,并采用 10 个不同的随机种子进行实验。随后,将结果与在相同条件下进行的噪声实验进行对比,包括 Wu 等人的 Attentive FP 模型、Cai 等人的 FP-GNN 模型,以及使用 ErG、ECFP 和 RDKit 分子指纹分别运行的 10 组 XGBoost 独立实验。图 4 显示,MoleculeFormer 在噪声环境下依然保持出色的鲁棒性,这表明该模型即使在数据质量较低的情况下也能获得稳定的预测表现(见补充表 3)。
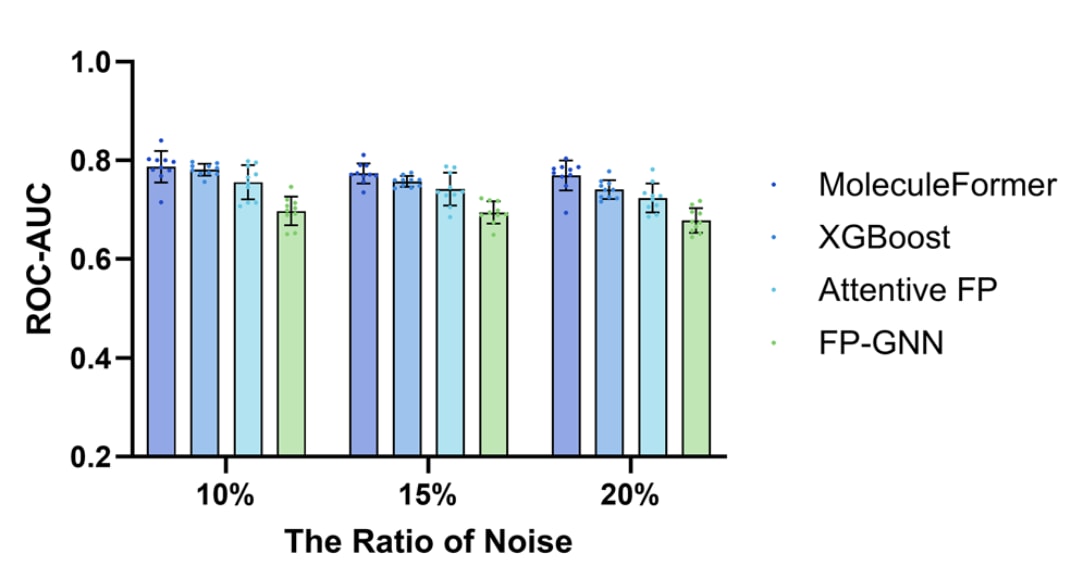
图4 | 展示了不同噪声率下的抗噪性能比较。 在 HIV 数据集上评估了 Attentive FP、XGBoost、FP-GNN 和 MoleculeFormer 在不同噪声率条件下的抗噪表现。FP-GNN 模型来源于 Cai 等人的研究;Attentive FP 模型来源于 Xiong 等人的工作。XGBoost 使用与 MoleculeFormer 相同的分子指纹选择方案。每组实验均包含 10 个独立样本,且相互之间无干扰。误差线表示均值 ± 标准误。
2.6 MoleculeFormer 的可解释性分析
为评估 MoleculeFormer 的可解释性,该研究团队选用了 BBBP 数据集。该数据集经过两阶段清洗:(1)移除 74 个重复的小分子以保证数据唯一性;(2)剔除 4 个无效的 SMILES 结构以确保数据完整性。在此基础上,模型在精炼后的数据集上重新训练,并开展可解释性分析实验。Xiong 等人对提升小分子脑部暴露能力的结构改造研究进行了综述,指出药物化学家通常通过调整脂溶性、降低氢键供体数量等策略,提高小分子穿越血脑屏障(BBB)的能力。MoleculeFormer 的预测结果与实验室研究得到的关键分子特征高度一致,表明该模型能够有效提取与 BBB 渗透性相关的结构特征,并将其作为重要的判别依据。
2.6.1 脂溶性(Lipophilicity)
脂溶性指化合物对脂类或非极性溶剂的亲和能力,而计算 LogP(cLogP)是衡量其油-水分配能力的重要指标,反映分子的脂溶性。提高分子的脂溶性通常能够增强其跨越 BBB 的能力。
在对 MoleculeFormer 的原子注意力机制研究中,模型利用自注意力层量化分子中每个原子的相对重要性,并通过可视化揭示其决策依据。如图 5a 所示,原子级注意力权重以颜色梯度呈现:深色区域对应具有高 cLogP 的结构特征(如疏水基团、芳环取代基等),浅色区域对应低 cLogP 区域。模型对高 cLogP 区域表现出显著的关注,这与 Xiong 等人提出的“提升脂溶性可增强 BBB 渗透性”的理论相一致。尤其在含有长链烷基或卤素取代基的分子中,模型的注意力显著集中于这些高脂溶性结构域,而实验研究表明此类基团可通过提高分子脂相分配系数有效促进跨膜扩散。
这种注意力分布模式验证了模型能够准确捕捉影响 BBB 渗透性的关键化学特征,并从计算角度解释了脂溶性基团提高药物脑输送效率的机制。通过将抽象的特征提取过程转化为可解释的可视化表示,该研究证实 MoleculeFormer 的决策逻辑与药物化学直觉高度一致,为其预测结果提供了可靠的理论支持。
Brand 等人通过优化高通量筛选所得的初始化合物,开发了先导化合物 1(DDD85646),其对 N-肉豆蔻酰转移酶(NMT)具有显著活性,但 BBB 渗透性较差(Kp < 0.1)。为改善渗透性,Wyatt 团队进行了两轮优化:首先设计并合成化合物 2,显著改善了 BBB 渗透性;随后进一步提升脂溶性并合成化合物 3,使其表现出更高的脑部渗透水平(图 6a)。模型预测结果显示,化合物 1 的预测渗透能力较弱,而化合物 2 和 3 的预测渗透能力逐步增强,与实验结果完全一致。
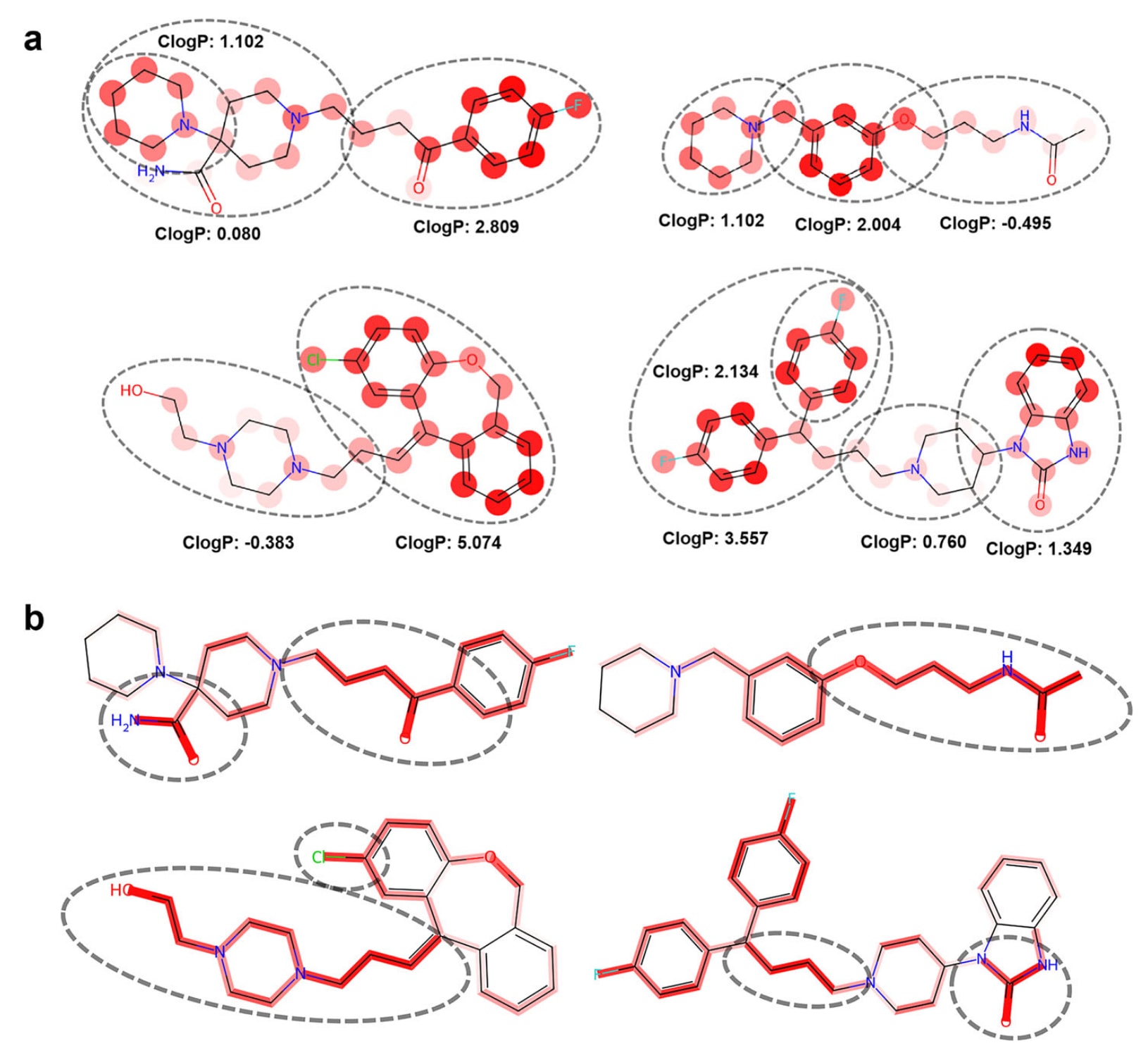
图5 | 展示了Transformer 层注意力权重的可视化。 每个分子被划分为两个部分,并分别计算其 cLogP 值。同时对 Transformer 层的注意力权重进行了可视化,其中颜色深浅代表各原子的注意力权重,颜色越深表示权重越高。该模型在注意力分布上更关注具有较高 cLogP 的结构区域(a)。键图在注意力分布上更关注分子中的极性基团,从而影响分子的氢键供体能力(HBD)。碳链结构具有较低的三维刚性,有利于通过 BBB 的孔径限制(b)。
2.6.2 氢键供体能力(Hydrogen-Bond Donor Capacity, HBD)
研究已证实,氢键供体数量(HBD)是影响 BBB 渗透性的关键因素之一,降低 HBD 通常有助于提高 BBB 渗透性,因此调控 HBD 是常用的药物设计策略。
在键注意力实验(图 5b)中,模型的注意力更多集中在分子中的极性键上,而极性键会影响分子的 HBD 能力。碳链结构具有较低的三维刚性,这有助于分子通过 BBB 的孔径限制;而具有较高极性或较大体积的官能团结构可能反映其对 HBD 的敏感性。HBD 会显著增强分子与 BBB 膜中极性基团的相互作用,从而阻碍被动扩散。碳链和非环状结构通常具有较小分子量(MW)和较低三维刚性,更易通过 BBB(一般要求 MW < 500 Da)。大环或稠合环结构由于体积大或刚性高,可能被模型视为“渗透障碍特征”。
键注意力集中于分子骨架的现象表明,该模型不仅依赖局部脂溶性特征,还能够通过全局结构信息学习复杂的构效关系规则,进一步验证了其多尺度特征提取机制的合理性。Fushimi 等人通过移除化合物 4 中不必要的 HBD 制得化合物 5,从而增强其 BBB 渗透性;随后通过屏蔽吗啉环上的氧原子合成化合物 6,使 BBB 渗透性进一步提高。模型预测结果表明,化合物 4 几乎无渗透能力,而化合物 5 和化合物 6 的预测得分逐步提高,与实验趋势一致(图 6b)。
Gunaga 等人研究了化合物 7,其作为潜在的维持正常窦性心律的安全药物,但存在 BBB 渗透风险。通过引入 HBD 改造为化合物 8 后,其 BBB 渗透性降低,同时保持药效。模型预测显示化合物 8 的预测渗透得分显著下降,与实验结果一致(图 6c)。
综上结果表明,MoleculeFormer 具备强大的可解释性,能够成功识别并量化影响 BBB 渗透性的关键分子特征(脂溶性与氢键供体能力),这些特征与传统药物化学原理高度一致。这种可解释性使模型不仅能够预测 BBB 渗透性,还能提供可用于实际药物设计的化学直觉依据,对中枢神经系统(CNS)药物研发具有重要应用价值。
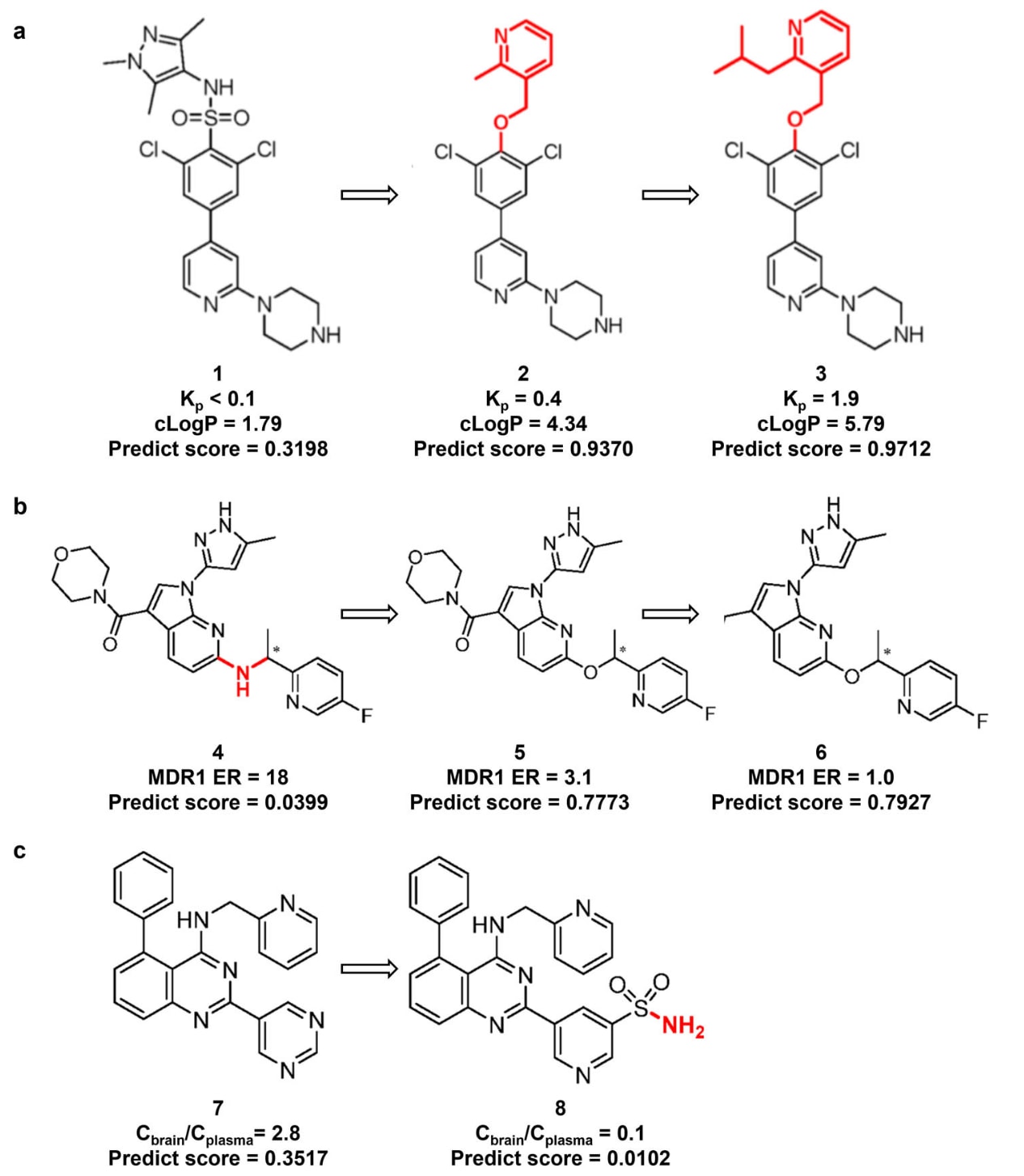
图6 | 展示了化合物 BBB 渗透性优化过程及模型预测一致性验证。 Brand 等人对初始 BBB 渗透性较差的化合物 1 进行了两轮结构优化。更高的 Kp 与 cLogP 值意味着更强的 BBB 渗透性(a)。Fushimi 等人对 BBB 渗透性较弱的化合物 4 进行了两轮优化。MDR1(P-糖蛋白)是 BBB 中的外排转运蛋白,会主动将药物排出脑组织,从而降低其渗透性;因此更低的 MDR1 水平通常对应更强的 BBB 渗透能力(b)。Gunaga 等人通过结构改造降低了具有 BBB 渗透潜力的化合物 7 的渗透风险。更低的 Cbrain/Cplasma 比值代表更弱的 BBB 渗透性(c)。该研究团队基于 MoleculeFormer 的预测结果与实验观测数据保持一致,其预测得分范围为 0 至 1。
3 总结
该研究提出了一种基于多尺度特征的可解释分子预测模型。模型以 GCN-Transformer 为基础结构,分别从原子图和键级图中提取特征。在原子图通道中,模型通过全局注意力权重动态捕捉分子内部原子之间的长程相互作用;在键图通道中,则重点关注局部化学键的拓扑特征与电子效应。该协同机制不仅增强了模型在多层级分子特征分析方面的能力,还通过注意力分布的可视化揭示了分子预测任务中的关键基团和隐含特征等作用机制。此外,模型同时考虑了分子图的旋转等变性以及分子指纹的先验知识嵌入,在局部与全局特征的协同提取上表现出色。
该研究系统性地选取多维度数据集,覆盖药物研发关键阶段的核心分子性质,以验证模型在分子预测任务中的通用性。实验数据集包括:(1)MoleculeNet 基准库中的十个核心子集,例如 HIV(抗病毒活性)、BBBP(血脑屏障渗透性)、Tox21(化合物毒性)和 SIDER(药物不良反应)等,这些任务从多个层面评价分子的理化性质、生物活性及安全性;(2)十四个细胞表型筛选数据集,反映分子在生物体系层面的活性响应;(3)ADME 药代动力学数据集,模拟药物在体内的吸收、分布、代谢与排泄全过程。该层次化设计不仅覆盖分类与回归任务,还贯穿药物研发流程从早期分子筛选到后期效能评价的多个阶段。
通过综合对比实验与消融实验可以看出,该模型以互补方式整合多种编码模块,在跨领域与多任务分子预测中均表现出稳定且优异的性能,验证了其在处理复杂分子表征与多目标预测方面的泛化能力。该研究观察到分子指纹的选择与任务类型之间存在显著相关性,最优的指纹选择高度依赖于任务属性。在分类任务中,ECFP 和 RDKit 指纹因能够捕捉具有区分度的结构差异而表现突出;它们的联合使用或与 ErG 指纹结合,可提供互补的结构信息,实现更优的分类效果。相比之下,在回归任务中,MACCS Keys 作为单一指纹表现最佳,而 MACCS Keys 与 EState 的组合通过整合关键特征–目标关系与电子/拓扑环境描述,显著提升了连续值预测能力。任务特征决定了有效指纹的选择:分类侧重区分性结构特征,而回归则依赖于与连续性质相关的定量描述。
值得注意的是,尽管 ErG 与 ECFP 单独用于回归任务时表现较弱,但在作为补充特征与 EState 组合时却显著提升了模型的预测性能(推荐回归任务指纹组合为:EState + ErG + ECFP),这一结果进一步凸显了在分子指纹选择中利用互补性进行组合策略的重要性。