Nature Methods 2025 | OpenStructure: 用于比较大分子复合物的全自动基准评测工具套件
今天介绍的这项工作来自 Nature Methods。该篇文章围绕大分子复合物结构预测中的评测难题展开,系统介绍了一套完全自动化的基准评测工具,用于在不依赖人工干预的情况下,将预测模型与实验参考结构进行全面而稳健的比较。随着基于人工智能的方法在蛋白质及其复合物结构预测中取得显著进展,模型数量和体系规模迅速增长,传统依赖人工设置或仅适用于小体系的评测方法已难以满足需求。该文章正是在这一背景下提出并总结了一套能够扩展到大型、复杂体系的统一评测框架。该作者首先回顾了结构预测领域长期以来的基准评测实践,包括针对单体结构、蛋白质复合物以及蛋白质-配体相互作用的多种国际评测活动,并指出现有评分方法在处理多链装配体、复杂界面以及配体构象时面临的局限。随后,该文章从化学实体一一对应映射这一核心问题出发,强调只有在建立合理的链映射和配体分配关系后,评分结果才能真实反映模型与参考结构之间的结构一致性,而非映射策略带来的偏差。在此基础上,该文章系统介绍了多类结构比较评分方法,涵盖三级结构、四级结构、蛋白质-蛋白质界面以及蛋白质-小分子配体复合物等不同层面,并讨论了刚性叠合类评分与局部、非叠合评分在应对结构柔性和结构不完整性方面的差异。该作者进一步提出并评估了新的链映射算法和配体评测指标,使得这些评分方法能够在大型复合物场景中高效、稳定地运行。整体而言该文不仅总结了当前结构评测方法的技术现状,也通过统一的实现框架将多种评分策略整合到同一工具体系中,为大规模、高通量的结构预测评测提供了可复现、可扩展的解决方案。该工作为未来复杂大分子体系的自动化评测奠定了方法学基础,在结构生物信息学和数据驱动的结构预测研究中具有重要参考价值

获取详情及资源:
0 摘要
蛋白质结构预测领域长期以来一直依赖系统化的基准评测工作,例如结构预测的关键评估、连续自动化模型评估以及相互作用预测的关键评估。随着基于人工智能的方法被广泛应用于大分子复合物结构预测,使用大规模数据集以及稳健、无需人工干预的评分指标,将预测结果与参考结构进行比较,已成为不可或缺的环节。同时,随着通过晶体学或冷冻电子显微镜解析的参考结构在规模和复杂性上的不断提升,现有结构比较方法也面临新的挑战。该文综述了当前结构评分方法的发展现状,分析了其中存在的局限性,并提出了更适合用于评估三级结构、四级结构、蛋白质-蛋白质界面以及蛋白质-配体复合物的评分策略。这些方法在设计上具备良好的可扩展性,能够支持对大型、复杂体系的评估。所有相关方法均已整合进OpenStructure结构基准评测框架中,该框架为开源软件,可通过https://openstructure.org/免费获取。
1 引言
蛋白质结构建模领域在预测方法准确性的基准评测方面具有悠久历史。研究中通常使用多种数值评分指标,将计算生成的模型与实验获得的真实结构进行系统比较,这些参考结构也常被称为“目标”或“金标准”。自1994年以来,结构预测关键评估实验以双盲形式每两年评估一次最先进的方法,成为推动该领域发展的重要动力。与之互补的连续自动化模型评估项目则以每周为周期评估自动化预测服务器,持续促进全自动预测与评测方法的发展。相互作用预测关键评估实验主要关注蛋白质-蛋白质复合物中界面预测的准确性,评测轮次大约每六个月公布一次,并且自2016年起与结构预测关键评估展开协作。与此同时,围绕小分子配体构象预测也开展了多项挑战性评测,涵盖不同类型的蛋白质-配体体系,相关配体评估自2022年起被正式纳入结构预测关键评估。在过去几十年中,客观、盲测且相互独立的基准评测工作一直是高精度结构预测方法得以发展的核心驱动力。尽管近年来预测精度取得了显著进展,即便是当前最先进的结构预测方法仍然存在局限性,这进一步凸显了持续开展基准评测工作的必要性。对于以数据驱动的人工智能预测方法而言,构建大规模数据集,并采用能够在高通量条件下、无需人工干预即可应用的稳健评分指标,已成为不可或缺的基础条件。
2 化学映射
为了能够在预测模型与参考结构之间进行比较,必须在参考结构与模型中,为所有等价的化学分子建立一一对应的映射关系,包括聚合物链、小分子配体等。为了实现稳健的基准评测,链映射问题被定义为在模型与参考结构之间建立链的一一对应关系,并且该映射应当在用于评估模型质量的评分指标意义下是最优的。这样可以确保评测结果真实反映最佳可能的结构对应关系,而不是由任意链分配方式引入的人为偏差。
对于聚合物而言,只有在单体预测的情况下,这种“链映射”才是直接且简单的。**将该问题扩展到包含多条聚合物链的复合物中,长期以来一直是一项挑战。**最朴素的方法是枚举所有可能的映射方式,分别计算评分并选取最优结果,但随着链数增加,问题复杂度呈阶乘增长,很快便变得不可行。为了解决这一问题,历史上提出了多种简化策略以支持复合物的评分。CAPRI社区提出了仅关注界面的评分方法,其适用对象局限于二聚体体系,从而规避了全局成对映射的需求。直到最近,CAPRI才开始通过对各个界面评分取平均的方式,评估更大规模的组装体。
当前大多数工具的目标是在刚性叠合类评分指标的基础上,通过启发式方法推导出明确的一一映射关系。例如,MM-align通过枚举完整解空间来最大化模型与参考结构之间的模板建模评分TM-score,但会跳过对不太可能映射方案的高成本评分计算。US-align同样以最大化TM-score为目标,但通过贪心搜索算法获得初始映射,并在此基础上进行迭代优化,从而避免穷举。AlphaFold-Multimer中描述的算法则从参考结构中选取一条锚定链,将所有序列匹配的模型链与其叠合,随后依据质心距离最小的原则逐步配对链,搜索质心均方根偏差最小的映射方案。Foldseek-Multimer通过对所有链进行两两叠合,并对得到的变换矩阵进行聚类,以识别彼此兼容的链集合用于映射。对于不依赖叠合的评分指标,有时可以将问题简化为在对称相关的分组之间识别映射关系。然而,针对这类评分指标专门设计的链映射方法,目前仍然缺乏系统性的解决方案。
小分子配体可以通过基于图的方法进行匹配,这里将其称为“配体分配”,以区别于聚合物的“链映射”。其面临的挑战与聚合物类似,但还需额外考虑配体内部的对称基团,例如苯环等情况,在这些情况下原子无法被唯一地确定对应关系。
在此基础上,该文章提出了一个统一且一致的框架,用于在参考结构与预测模型之间,为任意数量的化合物建立映射关系,无论这些化合物是蛋白质、DNA、RNA还是小分子配体。该灵活框架支持计算多种评分指标,从不同层面评估预测得到的大分子复合物结构质量。
3 比较评分
在此语境下,“评分”一词专指用于基准评测的定量指标,其目的是衡量预测模型与参考结构之间的一致程度。这一用法不同于基于能量的评分,例如某些对接或建模过程中使用的能量函数,这类评分并不能直接反映结构相似性。
针对三级结构的评分方法大体可以分为两类。第一类依赖于代表性主链原子的刚性叠合,通常以蛋白质的Cα原子为代表,典型指标包括r.m.s.d.、全局距离测试以及TM-score。由于对离群点和单个结构域运动极为敏感,r.m.s.d.在该领域中已逐渐被弃用。此外,r.m.s.d.只对已映射的原子位置进行计算,因此不会对不完整模型中缺失的残基施加惩罚,也会忽略仅存在于其中一个结构中的额外原子。CASP在一定程度上通过人工将参考结构划分为多个刚性子结构并分别评估,来缓解结构域运动带来的影响。然而,对于完全自动化的基准评测体系,例如CAMEO,以及面向人工智能的大规模结构比较任务而言,结构柔性仍然是一个难以回避的问题。
在这种情况下,第二类评分方法发挥了关键作用。这类方法侧重于局部环境差异,不依赖整体叠合,因此对结构域相对运动不那么敏感。典型代表包括接触面积差异评分和局部距离差异测试评分。这两种评分都会考虑所有重原子,因此只有在侧链构象被正确预测的情况下才能取得理想分值。
针对蛋白质装配体的基准评测,还需要一组专门关注界面准确性的评分指标。CAPRI通常依据配体r.m.s.d.、界面r.m.s.d.以及fnat等指标,将预测结果与参考结构的相似性粗略划分为“错误”“可接受”“中等”或“高质量”。为避免离散分类带来的局限,并更适合自动化建模方法的优化目标,DockQ评分被引入以将上述多个指标整合为一个连续数值。CASP在评估多聚体时,主要依赖基于界面接触的评分指标,即界面接触相似性和界面斑块相似性。为了同时反映各个亚基的准确性以及整体拓扑结构,这些以界面为中心的评分又与最初用于三级结构比较的评分方法相结合,包括LDDT和TM-score。当建模任务还涉及复合物化学计量数的预测时,例如在CAMEO中,QS-score尤为合适,因为它能够区分不同的四级结构形式和不同的计量关系。
小分子配体如何与蛋白质靶标结合,即配体构象预测,是药物发现中的重要问题。以往的配体构象预测挑战主要采用两类评分方法来评估受体-配体复合物的预测质量,一类是经过对称性修正的r.m.s.d.,用于衡量配体在结合位点中的绝对构象精度,另一类是基于接触的评分,用于评估天然受体-配体非共价相互作用的再现程度。在GPCR Dock以及D3R大挑战的早期阶段中,参赛者还需要预测受体蛋白本身的构象,评估过程因此需要额外将模型与参考结构进行叠合。
CASP15提出了更为复杂的挑战,要求参赛者预测完整的蛋白质-配体复合物,其中受体有时以寡聚体形式存在,并且可能包含多个配体。已有方法无法直接对这类复杂预测结果进行评分。Spyrmsd只能在相同参考坐标系下,对单一参考-模型配体对计算对称性修正的r.m.s.d.,不包含结合位点的识别与叠合,也无法推广到包含多个配体的体系。类似地,以往基于指纹的方法在评估蛋白质-配体相互作用时,通常只考虑一组主观选定的相互作用类型,例如氢键、离子作用、疏水作用、π相互作用或金属配位,并且依赖人工准备步骤,难以实现一致且可复现的评测。因此,有必要开发新的配体评估方法,以实现自动化的配体识别、支持寡聚体的链映射,并对受体进行适当叠合。由此产生的评分指标包括结合位点叠合后的对称性修正r.m.s.d.(BiSyRMSD)以及蛋白质-配体相互作用的LDDT评分(LDDT-PLI),这些方法随后得到了进一步完善,其具体实现细节在该文中进行了系统阐述。
4 该文目标
该文介绍了一套完全自动化、运行高效且结果可靠的工具体系,用于将理论预测模型与实验参考结构进行比较,并将其实现于OpenStructure结构生物学框架中。该文章系统讨论了不同评分方法的优势与局限,并针对在实际评测中需要重点关注的问题给出了方法学层面的建议。OpenStructure在评分计算过程中能够自动执行所需的全部步骤,其中包括当前先进的链映射算法,从而减少人工干预并提高评测一致性。
该框架提供了种类丰富且相互补充的评分指标,用于评估不同类型预测结果的准确性,其总体结构如图1所示。评测对象涵盖蛋白质、DNA或RNA的三级结构,单个或多个聚合物-聚合物界面,小分子与离子的构象及配体结合口袋的几何特征,以及预测得到的大分子复合物的化学计量关系。这些评分指标从多个层面共同刻画了预测模型对目标结构的再现程度,从而形成对模型准确性的整体认识。
将上述所有评分方法整合到一个统一、一致且高度优化的框架中,显著降低了开展高通量基准评测实验的技术门槛,无论是对方法开发者还是评测组织者而言都具有重要价值。该评分框架已被应用于近期的CASP和CAMEO评测工作中,验证了其在实际大规模评测场景下的实用性。
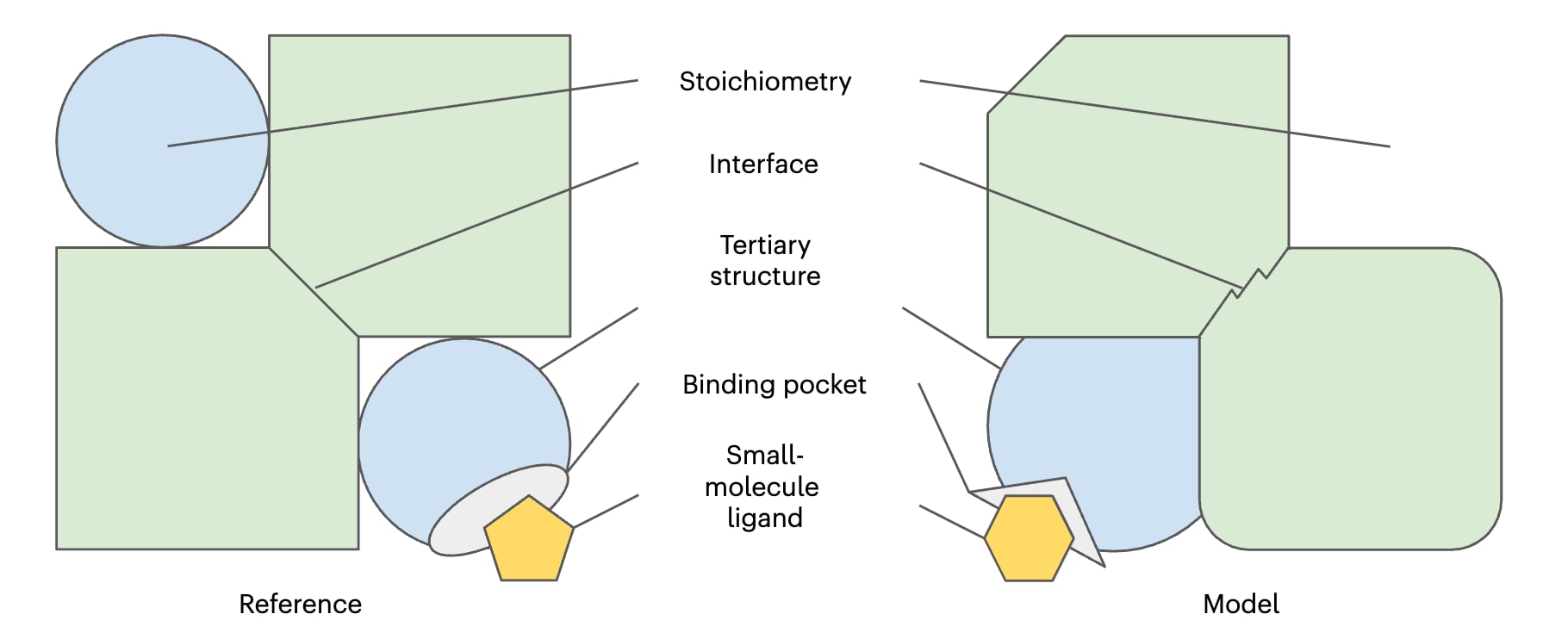
图1|结构预测评测的不同层面。 示意图展示了一个包含小分子配体的异源2-2二聚体参考结构(左)。对模型的评估(右)包括对等价组分进行映射,并对模型与参考结构之间的差异进行评分。模型中的不准确之处可能体现在多个方面,例如复合物的化学计量数错误、三级结构及界面结构上的差异、结合位点大小和形状不正确,以及小分子配体构象或结合姿态的偏差。与“找不同”游戏只进行定性比较不同,这里的差异需要以定量方式进行评估。
5 结果
5.1 聚合物链映射
截至目前,该领域仍缺乏一种既能适用于大型装配体,又能够基于不依赖叠合的评分指标进行链映射的方法,从而规避叠合类方法自身的局限性。该文提出了QSMap这一启发式算法,其优化目标为基于界面接触的QS-score或主链LDDT,其中蛋白质采用Cα原子,核酸采用C3′原子。对于需要重点关注整体拓扑结构的应用场景,同时引入了QSMapR算法,该方法以主链r.m.s.d.为优化目标,同样以蛋白质的Cα原子和核酸的C3′原子作为代表坐标。与AlphaFold-Multimer中采用的链映射算法类似,QSMap与QSMapR均依赖序列信息,即仅在被视为化学等价的链分组内部进行参考结构与模型之间的映射,这一特性在基准评测场景中尤为重要。所有相关算法细节均在方法部分中给出了完整说明。
为评估QSMap和QSMapR在实际应用中的性能上限,并比较其在真实基准评测场景下的表现,该文构建了两个测试数据集。同时,QSMapR还与US-align、Foldseek-Multimer以及对AlphaFold-Multimer中链映射算法的自有实现进行了对比,这些方法均依赖全局叠合并以评估整体拓扑为主要目标。第一个数据集由同源多聚体组成,其链数N逐渐增加,每种N最多随机选取十个结构,并通过RCSB PDB搜索接口获取。当可用结构不足十个时,通过随机选取更大的实验结构并截取前N条链的方式进行数据扩充。在该数据集中,同一结构同时作为模型和参考结构,用于测试链映射算法本身的行为。第二个数据集来源于CASP15装配体建模挑战,剔除了二聚体以及具有一一对应链分配的异源多聚体等简单情况,最终包含3,559个模型,其化学计量数范围从同源三聚体到异源9-9-9聚体不等。
总体而言,QSMap和QSMapR能够在秒级时间内处理最多约10条聚合物链的问题,在单线程AMD EPYC 7742处理器上,处理约30条链的问题通常需要数百秒。随着链数进一步增加,运行时间会逐渐上升并变得不再实用。这一性能显著优于朴素穷举方法,后者通常只能处理不超过约10条链的情况 (图2a)。在基于接触的比较中,QSMap得到的链映射在QS-score (图2b) 以及ICS、LDDT等类似评分指标上整体优于QSMapR(图2c)。相反,在关注整体拓扑结构的比较中,例如基于刚性叠合的TM-score,QSMapR生成的链映射表现更佳。可以看出,两种算法各自在其优化目标对应的评测维度上表现突出。
从运行时间角度来看,QSMap和QSMapR在所有测试案例中均能成功建立完整的链映射,且极少超过100秒。QSMapR生成的映射结果整体优于Foldseek-Multimer和AlphaFold-Multimer,在准确性上与US-align相当,但运行速度快约一个数量级。综合来看,在以界面接触为核心的评测场景中更推荐使用QSMap,而当整体拓扑结构是主要关注点时,QSMapR则是更合适的选择。
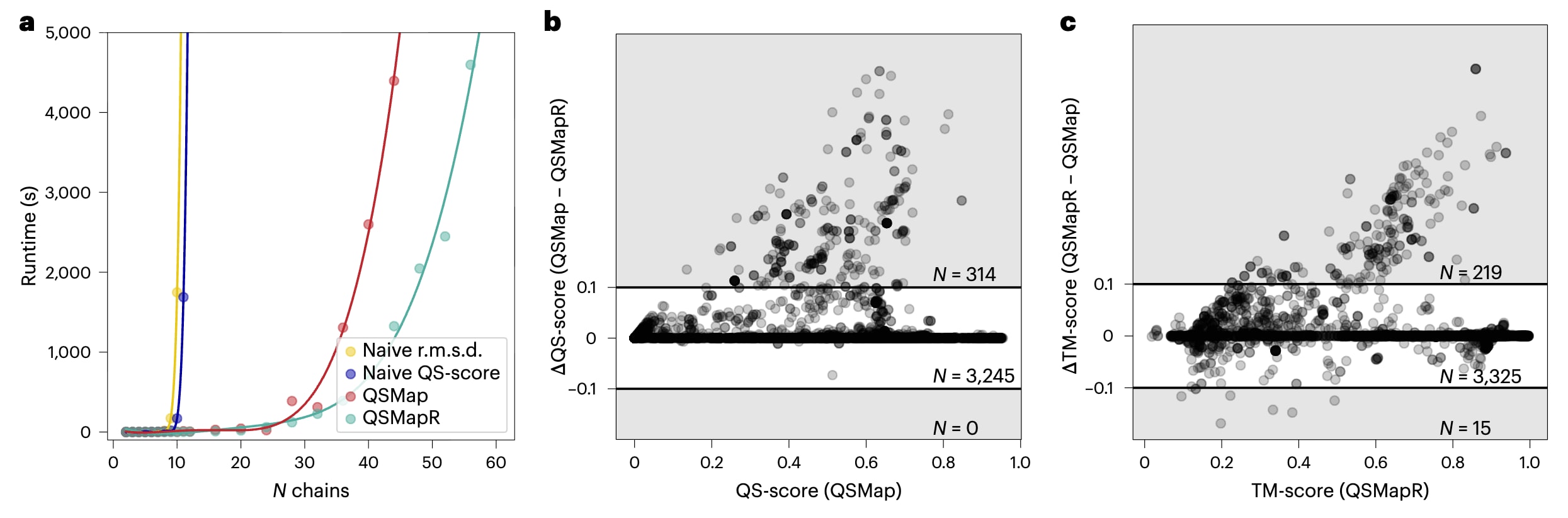
图2|QSMap与QSMapR的基准测试结果。 a, 展示了同源N聚体在不同链数条件下的平均运行时间,用于界定可处理的问题规模。当N大于10时,朴素穷举方法已难以实际应用,而QSMap和QSMapR仍能够完成更大规模结构的链映射。b, 在基于接触的比较中,QSMap通常能够获得比QSMapR更高的QS-score,表明其在此类评测场景下更具优势。c, 在依赖全局刚性叠合的比较指标中,QSMapR表现更优,因此更适合用于关注整体拓扑结构的评测。b和c中以阴影标出的区域表示评分差异较为显著的数据点,其绝对差值不小于0.1。
5.2 更新后的LDDT参考实现
LDDT通过计算位于给定包含半径内的原子对之间的距离差异来衡量结构差别,这些原子对在下文中被称为“接触”。该评分最早在CASP9中提出,由于对结构域相对运动具有良好的鲁棒性,能够支持完全自动化评估,随后被CAMEO采用为三级结构比较的主要评分指标。LDDT在计算过程中还包含立体化学检查,用于对严重的立体化学异常进行惩罚,并且最初仅适用于单链蛋白质结构。
自CASP13和CAMEO起,LDDT被扩展用于评估蛋白质的四级结构。然而,在CASP13中就已经发现,当时采用的链映射算法在处理大型装配体时能力不足,在这类情况下需要借助外部工具进行辅助。随着建模领域的研究重心逐渐转向大分子复合物,并且希望将不依赖叠合的距离差异概念应用于RNA或小分子体系,该文提出了一套新的LDDT参考实现。
该实现通过与QSMap链映射算法的紧密集成,能够成功处理大型装配体结构,并进一步扩展以支持核苷酸体系。在此基础上,还引入了两种变体评分方法:(1)i-LDDT,仅考虑跨界面的距离;(2)bb-LDDT,仅基于代表性的主链坐标进行计算,其中肽链采用Cα原子,核苷酸采用C3′原子。围绕这些方法,该文章在CASP15 RNA建模挑战的背景下开展了大量测试,并与其他评分指标进行了系统比较。尽管在CASP15中尚未对核苷酸实施立体化学检查,但相关检查已在该文工作以及CASP16中得到实现。
5.3 小分子配体
为了评估小分子配体在与靶标结构形成复合物时的预测准确性,例如辅因子、抑制剂或药物分子,在CASP15的背景下提出了三种新的评分指标。其中,BiSyRMSD是一种经过对称性修正的r.m.s.d.,单位为Å,用于在将模型的结合位点坐标叠合到参考结构之后,衡量配体构象预测的绝对准确性。结合位点仅依据参考结构进行定义,即所有至少有一个原子位于配体4Å范围内的残基,不包括氢原子。由于CASP中的参考结构在规模和柔性上均大于GPCR Dock以及D3R大挑战第一阶段中的体系,因此需要采用局部叠合策略。BiSyRMSD数值越小表示预测越准确,在对接研究中,低于2Å通常被视为预测成功。尽管该评分没有严格的上限,但当配体被放置在距离正确位置超过20Å处时,若其远离实际相互作用的链,结合位点识别可能会失败。为减轻基于r.m.s.d.的评分在不完整预测中的局限性,该方法要求参考结构中的配体必须被完整建模。
LDDT-PLI是一种将LDDT限制于聚合物-配体原子接触的评分方法,通过考察位于给定包含半径内的所有原子对,评估模型对天然接触的再现程度,并对模型中过度预测的接触施加惩罚。与LDDT类似,LDDT-PLI的取值范围为0到1,数值越大表示预测越准确。非零的LDDT-PLI意味着配体被放置在正确的结合口袋中,而随着关键接触无法满足,该评分会迅速下降至0。不过,即使BiSyRMSD数值较大,只要柔性配体中与聚合物发生接触的部分被准确建模,LDDT-PLI仍可能保持在大于0的水平。第三个评分指标是LDDT-配体口袋评分,即LDDT-LP,它将LDDT限制于结合位点中聚合物残基之间的原子接触,取值同样在0到1之间。当模型与参考之间仅映射到一个结合位点残基时,LDDT-LP的值为0。上述所有评分方法均自动处理链映射和配体的对称性修正,并且在存在多个配体时,会生成配体分配方案,确保每个参考或模型配体至多参与一个聚合物-配体相互作用对。
为分析这些评分指标的行为特性,研究汇总了CASP15配体建模挑战中所有被评估的模型。图3a展示了LDDT-PLI与BiSyRMSD之间的关系,其中BiSyRMSD采用对数坐标。缺失值,即评分无法计算或不存在的情况,以三角形标注。尽管两种评分之间呈现出极强的负相关关系,其Spearman相关系数为
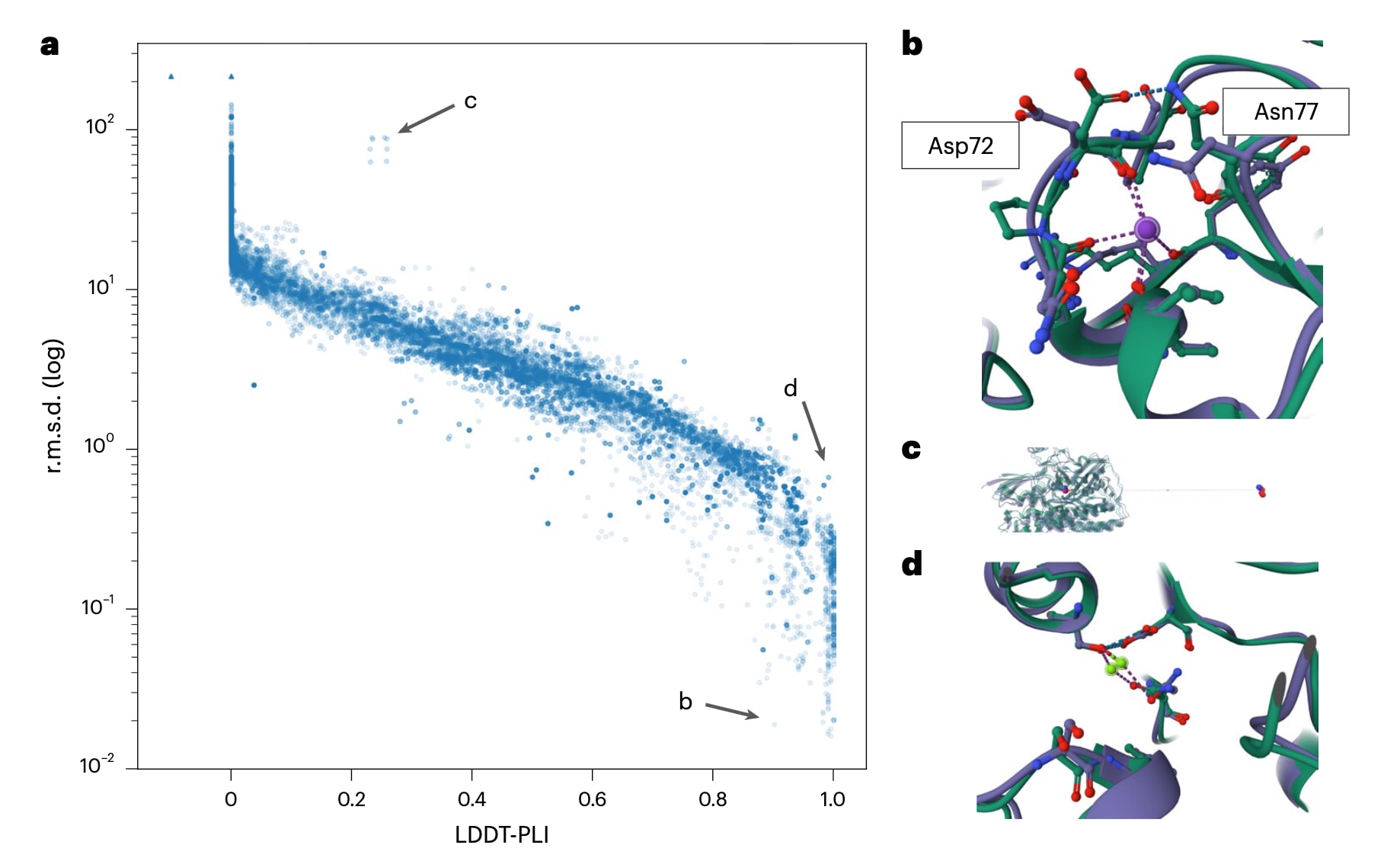
图3|BiSyRMSD与LDDT-PLI之间的关系。 a, 展示了LDDT-PLI(横轴)与BiSyRMSD(纵轴,对数刻度)之间关系的散点图。每个点(n = 31,905)代表一对已分配的参考配体对。对于无法计算LDDT-PLI(n = 7,798)或BiSyRMSD(n = 7,921)的情况,以三角形表示,其数值分别被替换为−0.1(LDDT-PLI)或分析中观测到的最大BiSyRMSD值的1.5倍。所有点的透明度为10%,颜色更为实心的点表示存在多个完全相同的预测结果。图中以箭头标出了若干具有代表性的离群点。b–d, 给出了a中标注的LDDT-PLI与BiSyRMSD表现不一致的配体构象预测示例,分别对应侧链翻转的情况(b)、配体发生断裂的情况(c),以及在保持原子间距离的情况下离子位置错误的情况(d)。模型链以紫色表示,参考结构链以绿色表示,结合位点残基和配体均以球棒模型显示。
图4进一步说明了LDDT-PLI中针对模型中额外接触所施加惩罚的影响。在图4a中,模型中额外预测出一条链穿过结合位点,并同时与配体和结合位点发生空间冲突。如果没有这条链,模型几乎可以获得完美评分,BiSyRMSD小于0.5,LDDT-LP和LDDT-PLI均大于0.9。但在考虑新增接触惩罚后,LDDT-PLI下降至0.53,反映出中等水平的预测准确性。图4b展示了更为细微的情况,即环区和侧链的错位,其中末端环尤其是Arg6被预测得比参考结构更靠近配体,导致LDDT-PLI降至0.65,而BiSyRMSD和LDDT-LP受影响较小,分别为1.11和0.80。尽管新增接触惩罚在检测偏离参考结构方面非常有效,但该机制并不能替代对模型进行系统的立体化学检查。
需要指出的是,这些配体评分方法在定义上仅适用于聚合物链与小分子配体之间的相互作用,并未考虑配体可能与其他小分子或短肽发生的相互作用。这在某些情况下会带来问题,例如在CASP目标T1118v1中,铁离子主要与大环双胞菌素类配体相互作用,而非与蛋白质发生作用,在默认参数下便无法为铁原子计算评分。一种变通方案是增大结合位点和LDDT的包含半径,使其覆盖蛋白质,从而允许评分计算。尽管存在这些局限性,BiSyRMSD、LDDT-PLI和LDDT-LP在实际应用中已被证明是相互补充的评分指标,能够从不同角度有效评估小分子配体构象预测的质量。
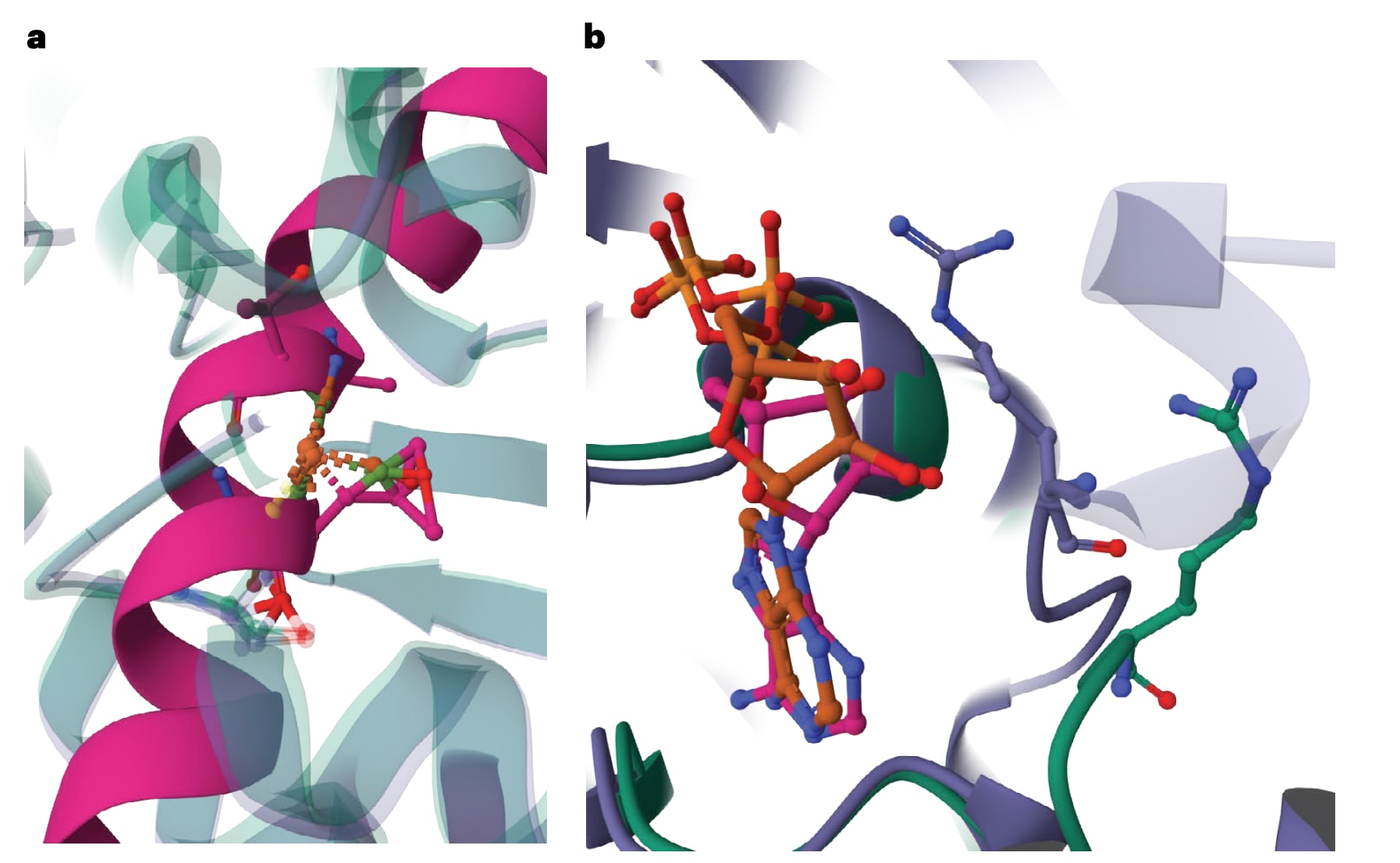
图4|LDDT-PLI中对模型新增接触施加惩罚的影响。 a和b分别展示了两种情形:a中在结合位点内额外预测出一条模型链(粉色),b中则存在一个位置错误的精氨酸残基。两种情况均以球棒模型进行示意。模型链以紫色表示,参考结构链以绿色表示。
5.4 OpenStructure作为全自动基准评测工具套件
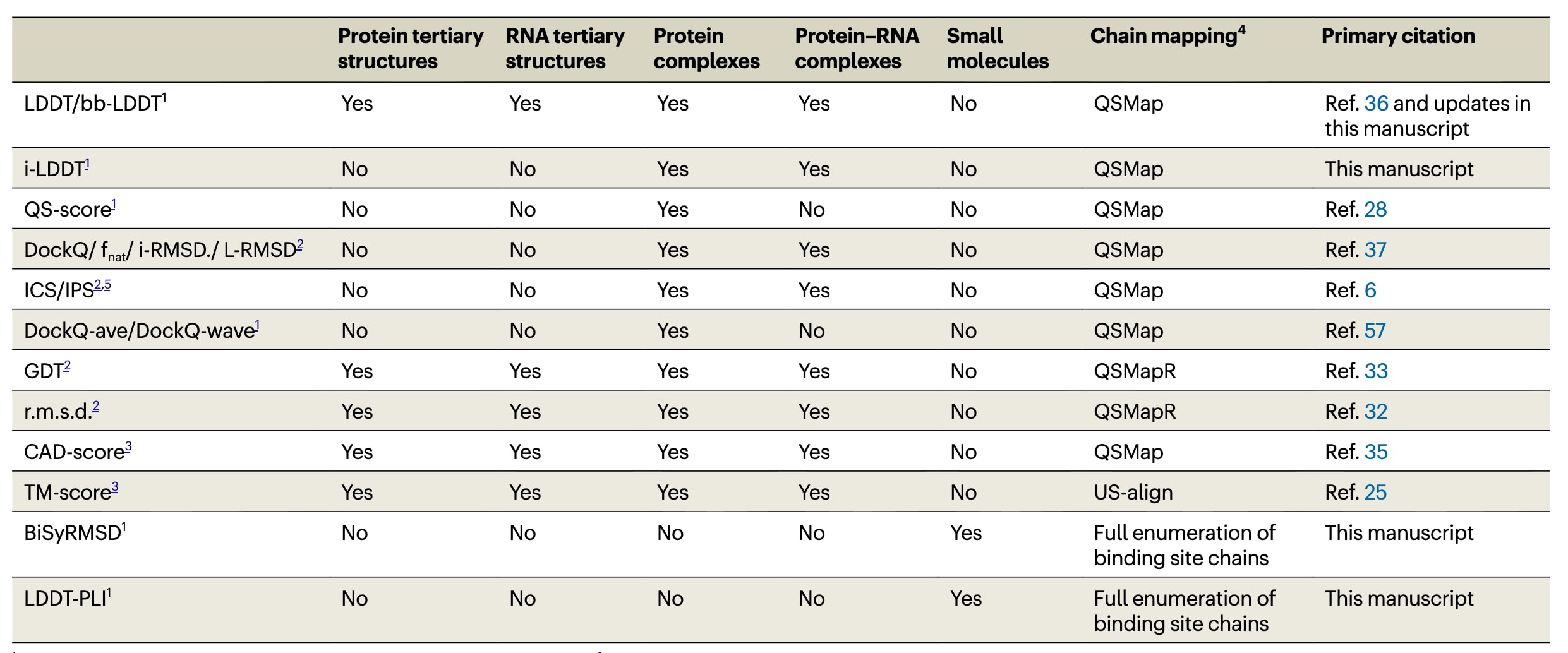
**概述。**OpenStructure计算结构生物学框架对该文中涉及的多种比较评分方法进行了系统化与统一实现,涵盖蛋白质结构、蛋白质复合物以及蛋白质-配体复合物等不同层面的评测需求。同时,该框架还整合了多种外部工具,以补充自身实现的功能,从而为结构建模方法的开发者和基准评测的执行者提供一个集中式的一站式评测环境。所有可用的评分指标在表1中进行了汇总,并可通过命令行接口进行计算,具体使用方式在“命令行接口”部分中给出说明。此外,其中一部分评分功能也可以通过Web服务器调用,相关内容在“Web接口”部分中进行了介绍。
表1|OpenStructure中提供的比较评分指标及其应用范围
**命令行接口。**该基准评测工具套件以可执行程序的形式集成在OpenStructure中,这些程序被称为“actions”。其中有两个核心action专门用于将大分子复合物的理论预测模型与对应的实验参考结构进行比较,并可直接计算该文中介绍的各类评分指标。
“Compare-structures”主要用于涉及聚合物实体的比较,包括蛋白质、DNA和RNA链之间的结构评测。“Compare-ligand-structures”则侧重于评估聚合物实体与非聚合物实体之间的相互作用,即蛋白质或核酸与小分子配体之间的结构比较。
关于这些action的具体使用示例,可在OpenStructure的Git仓库中查阅相应的README文档,其中不仅给出了评分计算的示例命令,还详细说明了如何通过Docker、Singularity或Conda环境部署OpenStructure,以及如何从源代码进行编译安装。
**Web接口。**针对聚合物实体的部分评分功能,也可通过SWISS-MODEL结构评估服务器在线使用。用户只需提供参考结构,即可获得包括LDDT、QS-score、TM-score、r.m.s.d.以及DockQ在内的多项关键评分指标。此外,命令行action所提供的完整功能也可以通过REST API进行自动化访问,从而支持更大规模的程序化评测流程。
**评分使用建议。**在这一部分中,该文章总结了结构预测评分过程中常见的问题,并针对自动化分析场景提出了实践层面的建议。
首先,基于全局叠合的评分方法并未考虑结构柔性这一关键因素。蛋白质本身具有柔性,通常由多个相对刚性的结构域组成,而这些结构域之间的相对取向可能发生变化。依赖叠合的评分方法无法反映这种柔性,往往需要对参考结构进行人工分割。在表1所列评分中,GDT、r.m.s.d.和TM-score均受到这一问题的影响,其结果可能被人为拉低,从而掩盖结构域层面上本已准确的预测。相比之下,不依赖叠合的局部评分方法,例如LDDT和CAD-score,通过关注局部内部接触或距离差异,有效降低了因结构域取向预测错误而带来的惩罚。因此,在评估蛋白质整体结构预测准确性时,除非研究目标在于区分结构域相对取向或整体拓扑,LDDT和CAD-score通常是更合适的选择。
其次,基于r.m.s.d.的叠合与评分对离群区域极为敏感。尽管GDT和TM-score同样依赖叠合,但它们通过优先对齐预测正确的区域,并将错误区域视为离群点,在一定程度上缓解了这一问题。以最小化r.m.s.d.为目标的叠合应谨慎使用,或仅限于局部区域,例如BiSyRMSD或i-RMSD等场景。L-RMSD及其衍生的DockQ评分在某些情况下存在问题,因为L-RMSD首先基于整个受体链进行叠合,随后再对整个配体链计算r.m.s.d.,这两个步骤都可能受到远离界面的错误区域影响。CAPRI社区在模型质量分级规则中已注意到这一问题,并在必要时采用i-RMSD替代L-RMSD。
此外,不完整模型在评分时应当低于覆盖目标序列完整的预测结果。接触类评分以及GDT和TM-score在设计上会自然惩罚不完整模型,而基于r.m.s.d.的指标则不会,因为缺失残基无法计算对应距离。表1中的i-RMSD、L-RMSD、DockQ、r.m.s.d.以及BiSyRMSD均受到这一问题影响。尽管BiSyRMSD通过仅考虑完整配体在一定程度上缓解了该问题,但结合位点的初始叠合仍可能受到坐标缺失的干扰,因此需要结合OpenStructure输出结果进行仔细检查。
另一方面,由于实验方法本身的局限性,参考结构中可能存在缺失区域,例如在X射线晶体学或冷冻电镜结构中电子密度不足而无法建模的部分。在这种情况下,预测模型覆盖这些缺失区域不应受到惩罚。这一问题主要影响QS-score以及ICS和IPS。QS-score在OpenStructure中也称为QS-global,其设计目标是比较复合物并区分不同的四级结构状态,并且在模型与参考结构互换时保持对称性。因此,当参考结构不完整时,模型中独有的接触会被计入惩罚,即便这些残基在实验中并未被解析。若基准评测场景假设模型与参考具有相同的化学计量数且模型覆盖完整,则更推荐使用QS-best变体。该变体在计算时仅评估模型与参考结构中共同存在的残基之间的接触,但也因此不会惩罚不完整模型或化学计量数错误的模型。ICS和IPS也存在类似情况,为此,在假定化学计量一致且模型完整的评测场景中,compare-structures action提供了“trimmed”版本,即在评分前将模型裁剪至仅包含参考结构中存在的残基。
以界面为中心的评估可以通过多种评分方法实现,包括i-LDDT、QS-score、DockQ、fnat、i-RMSD、L-RMSD以及ICS和IPS。独立的CAD-score程序也支持仅基于界面接触的评估,但该功能未被集成进OpenStructure基准评测套件中。DockQ系列评分主要针对二体相互作用,对于高阶寡聚体,可以使用DockQ-ave或DockQ-wave进行汇总。这两种方法在聚合各界面贡献时有所不同,其中DockQ-wave会根据界面大小对贡献进行加权,这可能导致在整体拓扑或生物学功能中至关重要的小界面被低估,在某些情况下,DockQ-ave的简单平均反而更具信息价值。需要注意的是,其他基于接触的评分方法,例如i-LDDT、QS-score以及ICS和IPS,同样可能被大型界面主导。
在建立两条聚合物链之间逐残基对应关系时,序列比对是必要前提。除TM-score外,表1中的所有评分方法均依赖基于序列的成对比对。在CASP或CAMEO等基准评测场景中,模型通常要求按照目标序列进行编号,因此更建议在这些情况下强制使用基于残基编号的比对方式。这一设置不会影响OpenStructure中TM-score的计算,因为TM-score通过US-align以默认参数计算,采用的是不依赖序列的结构比对。但需要注意的是,这种纯结构驱动的方法可能会导致不同身份的链被错误映射。
最后,仅基于主链的评分方法,例如bb-LDDT、QS-score、DockQ、i-RMSD、L-RMSD、GDT、RMSD和TM-score,只考虑聚合物主链的代表性原子位置,不会对侧链建模错误施加惩罚。然而,侧链在蛋白质结构中具有关键作用,在大多数基准评测场景中,确保侧链构象被正确预测仍然是一个重要目标。
6 讨论
尽管该文提供了种类丰富的评分指标,仍然存在一些尚未完全解决的应用场景,例如蛋白质复合物与多种分子实体之间的相互作用,包括肽段、寡糖以及翻译后修饰等。这些情况揭示了该领域在建模层面仍面临的进一步挑战,要获得对大分子复合物更加全面的认识,仍需持续方法学上的拓展。诸如结构质量验证以及更为细致的立体化学分析并未纳入该文的工作范围。结构质量验证的自动化本身具有较高难度,相关检查目前正被逐步引入到基准数据集构建流程中,并计划在未来进一步融入评分体系本身。蛋白质-肽段相互作用的评估也受到方法学限制,主要原因在于缺乏能够适用于任意非标准氨基酸的可靠比对方法。至于结构柔性与无序区域相关的问题,由于涉及完全不同的一类分析方法,同样不在该文讨论范围之内。
总体而言,OpenStructure评分框架构成了一套全面的基准评测工具体系,通过多种相互补充的评分指标,以稳健且自动化的方式评估不同类型的三维结构预测结果。多种评分的联合使用能够从不同层面深入刻画模型的预测准确性。随着数据驱动的人工智能结构预测方法不断发展,高通量的基准评测在评估新方法性能方面将变得愈发关键。通过提供一个统一、一致且高度优化的评测框架,该工作为该领域未来的方法学发展提供了重要支撑。OpenStructure已在CASP16和CAMEO等评测工作中得到广泛应用,其实用性也在这些实际场景中得到了充分验证。