NCS 2025 | Graphinity: 研究用于实现可泛化抗体–抗原ΔΔG预测所需的数据规模与多样性
今天介绍的是发表于NCS的Graphinity工作,聚焦一个长期被忽视却至关重要的问题:要实现可泛化的抗体–抗原ΔΔG预测,究竟需要多少数据,以及什么样的数据。作者提出基于三维结构的等变图神经网络Graphinity,在小规模实验数据上虽然可获得较高相关性,但系统分析表明这些结果主要源于过拟合。通过构建近百万规模的FoldX合成数据集和数万条Flex ddG数据,研究定量评估了数据规模、多样性与模型性能之间的关系,发现当前实验数据远不足以支撑稳健预测,至少需要数万到数十万条高质量ΔΔG数据。同时,抗体序列多样性和氨基酸替换类型对模型泛化能力具有决定性影响。该研究为抗体亲和力预测领域提供了清晰的数据需求下限,也为未来实验设计和机器学习方法开发指明了方向。

获取详情及资源:
0 摘要
抗体–抗原结合亲和力是治疗性抗体开发的核心:疗效取决于特异性结合以及对亲和力的精确调控。该研究提出Graphinity,一种直接基于抗体–抗原结构构建的等变图神经网络架构,在实验测得的结合亲和力变化(ΔΔG)预测任务上,测试集皮尔逊相关系数最高可达0.87。然而,与以往方法类似,该模型似乎在目前仅有的数百个实验数据点上发生了过拟合,其性能对训练集与测试集的划分并不稳健。为探究实现可泛化ΔΔG预测所需的数据规模与类型,构建了近一百万个由FoldX生成的ΔΔG合成数据集,以及超过2万个由Rosetta Flex ddG生成的ΔΔG数据。结果表明,当前实验数据的数量仍不足以对ΔΔG进行准确且稳健的预测,可能需要高出数个数量级的数据规模。除数据规模外,数据多样性同样是影响模型预测能力的重要因素。这些发现为未来方法开发与数据采集工作提供了数据需求的下限参考。
1 引言
抗体通过与靶抗原的特异性结合来介导其生理和治疗功能。因此,在治疗性抗体开发过程中,无论是在先导候选的筛选还是在后续优化阶段,对亲和力的调控都是最核心的考量因素。除亲和力之外,还有许多其他性质同样至关重要,通常被统称为可开发性,例如自聚集性、人源化程度、多反应性以及特异性等。近年来,利用机器学习预测这些性质取得了显著进展。然而,为了改善这些性质而对抗体序列进行的改动,不能以牺牲结合能力为代价。因此,治疗性抗体的开发本质上依赖于解决一个复杂的多参数优化问题。
用于定量测量亲和力变化的实验技术通常过程缓慢且工作量大。若能获得一种快速且准确的计算方法来预测亲和力变化,将在抗体设计流程中填补重要空白。此外,计算方法在理论上可以整合来自不同预测器的信息,在同时优化多种性质的同时仍然保持对结合亲和力的控制。尽管如此,抗体–抗原亲和力的计算预测仍然是一个具有挑战性的问题。传统的亲和力预测工具,如FoldX和Rosetta Flex ddG,基于物理方程和经验测量,能够重现体系的主要物理特性,并在某些应用中表现有效,但在速度和精度方面仍然存在局限。
近年来,研究重心逐渐转向机器学习方法,大体可分为两类:基于序列的方法和基于结构的方法。基于序列的方法在数据量充足且针对特定抗原的情况下,已成功用于亲和力预测,但其泛化能力有限,因为训练信息具有抗原特异性,模型难以在无需再训练的情况下直接应用于其他抗原。相比之下,基于结构的方法旨在捕捉多种抗体–抗原复合物中的相互作用模式,因而在理论上具有更强的泛化潜力。现有方法通常基于抗体–抗原复合物结构中提取的特征进行训练,例如结合表面积、原子间相互作用以及能量相关项。然而,这些方法在脱离训练数据分布时预测性能往往较差,同时还依赖于特征提取过程,不仅计算开销较大,也容易受到人为偏差的影响。
在该研究中,研究者系统研究了机器学习方法预测抗体–抗原结合亲和力变化ΔΔG的能力及其数据需求。研究者提出了一种等变图神经网络架构Graphinity,在包含645个单点突变的AB-Bind数据集上,其预测结果的皮尔逊相关系数最高可达0.87。然而,进一步分析表明,这种较高的性能主要源于模型过度拟合,并不具备良好的泛化性,这一现象在以往方法中也曾被观察到。为探究构建高精度预测模型所需的数据规模与类型,研究者生成了近一百万个由FoldX计算得到的ΔΔG合成数据,以及超过2万个由Rosetta Flex ddG生成的ΔΔG数据。在大规模FoldX数据集上,Graphinity的皮尔逊相关系数接近0.9,且对训练集与测试集之间的序列相似性阈值和噪声具有良好的稳健性。通过在不同规模的合成数据上评估模型性能,研究者发现当前实验数据的数量不足以支撑可泛化的ΔΔG预测,很可能需要高出数个数量级的数据。研究者的结果为所需数据量提供了一个下限估计,并强调了数据集多样性对于模型预测能力的重要性。
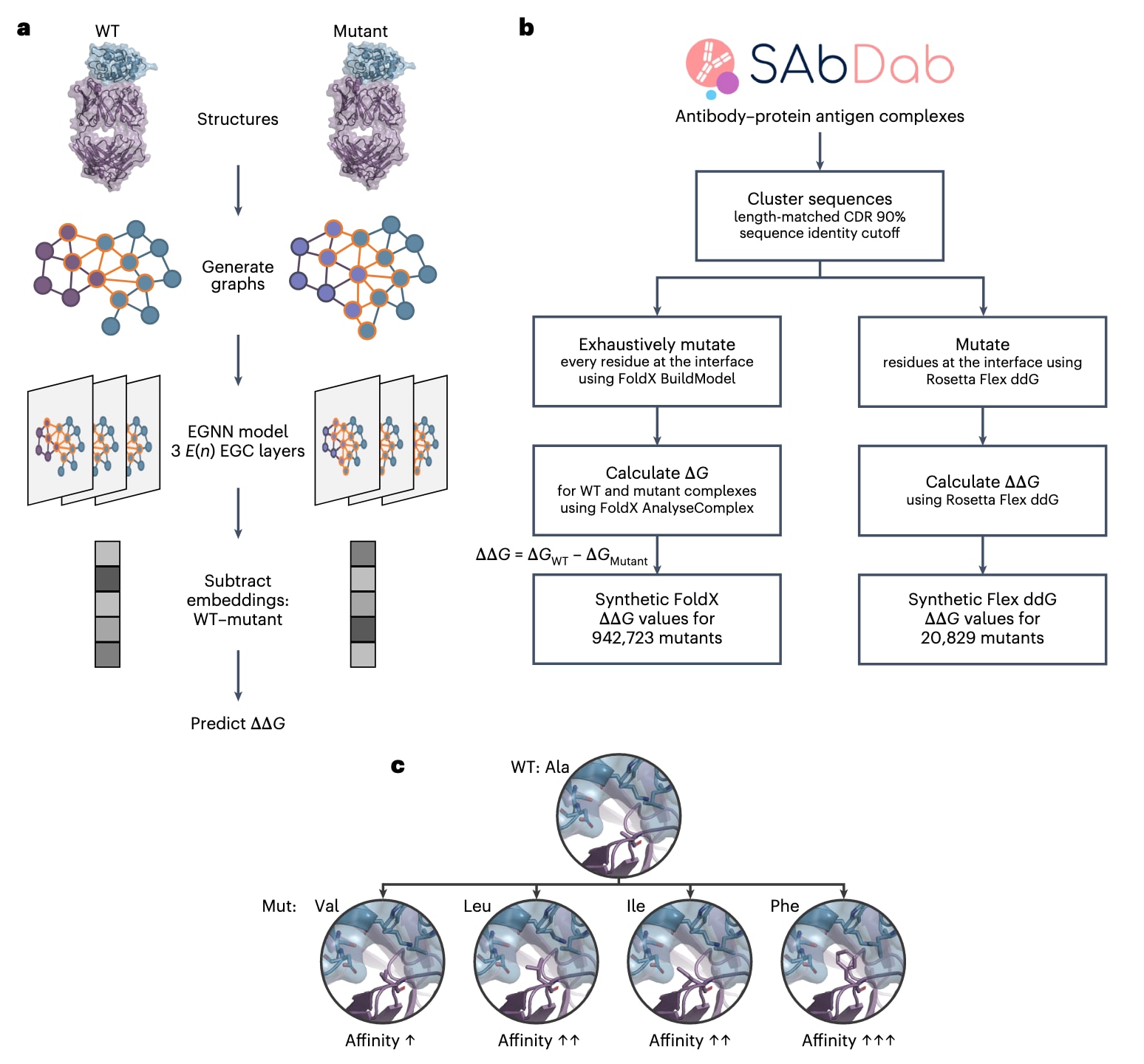
图1|Graphinity架构与合成数据集的构建。 a,EGNN深度学习模型在三维蛋白质结构坐标所构建的图上进行训练。图由突变位点邻域内的原子构成,以圆形表示节点(原子),以连接线表示边(相互作用)。抗体以紫色表示,抗原以蓝色表示,跨结合伙伴的边以橙色表示。模型架构由三层E(n) EGC层组成,随后连接一个线性层。在ΔΔG预测任务中,分别对WT复合物和突变体复合物通过E(n) EGC层生成的嵌入向量进行相减,再输入线性层。b,合成ΔΔG数据集来源于SAbDab中具有已解析结构的复合物。对界面残基进行突变,并使用FoldX和Rosetta Flex ddG预测ΔΔG值。c,一个复合物的ΔΔG数据示例。PDB:1XGP;抗体以紫色表示,抗原以蓝色表示;亲和力数值来自SKEMPI 2.0。Mut表示突变体。
2 结果
2.1 Graphinity模型架构
Graphinity以野生型(WT)和突变型抗体–抗原复合物的结构作为输入,将相应的图表示送入一个孪生等变图神经网络(EGNN),并预测ΔΔG(图1a)。在原子级图中,所有非氢原子被表示为节点,节点之间距离小于4 Å的相互作用被表示为边。图的范围限制在突变位点周围的局部邻域内。该架构具有模块化特性,可以方便地扩展到回归或分类任务,也支持单图或多图输入。完整细节见方法部分中的“Graphinity: EGNN architecture”。
2.2 Graphinity在实验ΔΔG预测中的表现
研究者将Graphinity应用于AB-Bind提供的实验ΔΔG数据集,该数据集包含来自29个复合物的645个单点突变,下文称为Experimental_ΔΔG_645。研究者还考虑了假设性的反向突变,以及AB-Bind数据集中不结合的突变,其ΔΔG值被任意设定为−8 kcal mol−1。模型在10折交叉验证中的皮尔逊相关系数最高可达0.87,优于已有方法报道的最高约0.76的相关性。然而,当通过在不同折之间施加序列一致性阈值来检验模型鲁棒性时发现,这些较高的相关性主要来源于过度拟合而非真正的泛化学习。当施加互补决定区(CDR)长度匹配且序列一致性为100%的阈值,确保同一复合物中的突变不会同时出现在训练集和测试集中时,皮尔逊相关系数平均下降了63%。模型性能对是否包含非结合突变也高度敏感,并且在所有训练–测试划分条件下,不同折之间的相关性差异都很大。此前的多种方法在施加类似训练–测试划分约束后,也都观察到了实验ΔΔG预测泛化能力不足的问题。例如,对TopNetTree进行留一复合物验证时,平均皮尔逊相关系数下降至0.17。研究者还将两种通用蛋白–蛋白相互作用ΔΔG预测方法DGCddG和RDE-PPI Network重新训练并应用于Experimental_ΔΔG_645数据集,在90% CDR序列一致性阈值下,它们分别仅达到0.26和0.22的相关性。在基于SKEMPI 2.0数据库构建的基准数据集Experimental_ΔΔG_608上,包含更多抗体–抗原复合物,也观察到了相似趋势。
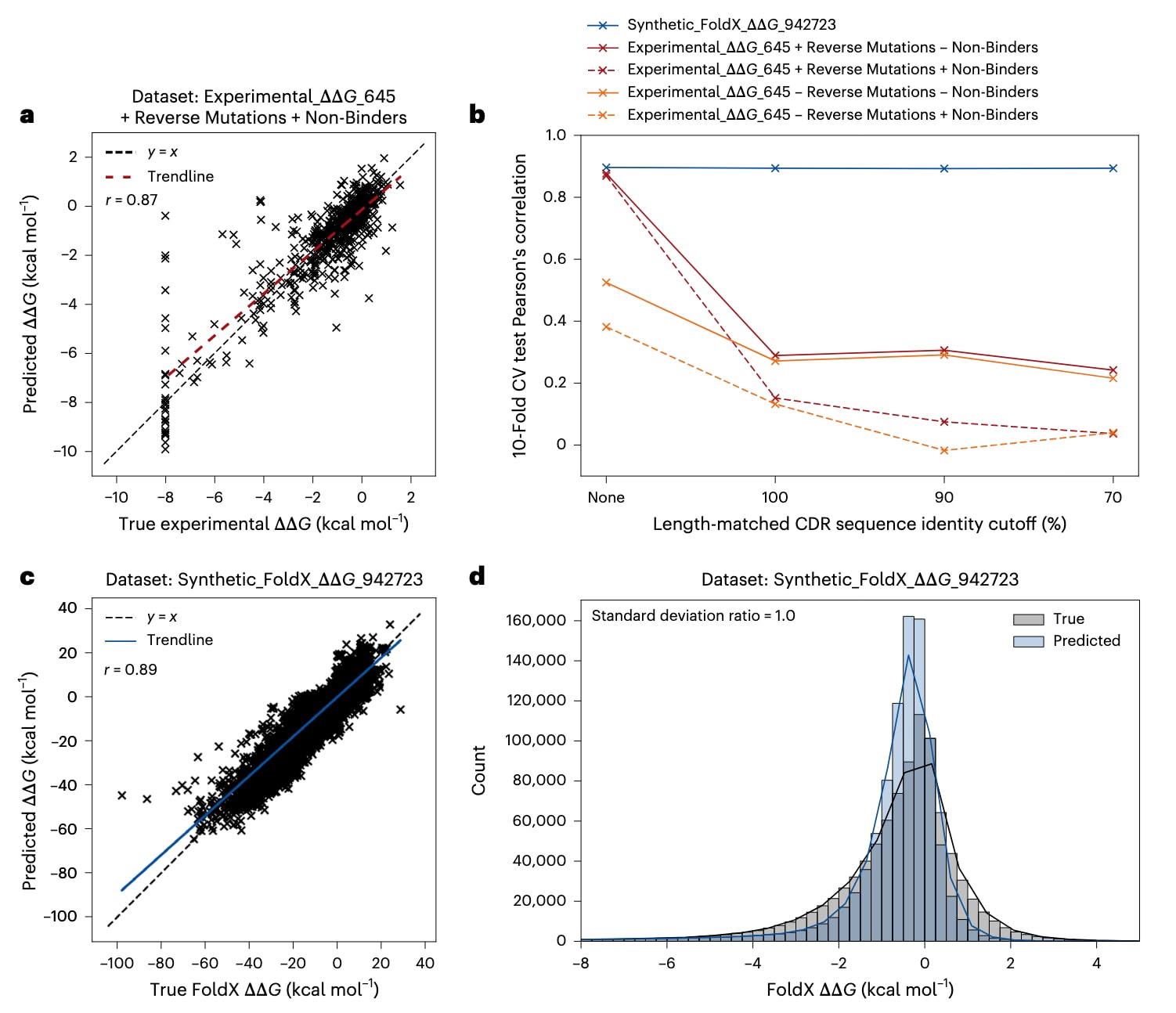
图2|Graphinity模型在ΔΔG预测中的性能。 a,在Experimental_ΔΔG_645+Reverse Mutations+Non-Binders数据集上,采用随机训练–验证–测试划分时,Graphinity预测值与真实实验值之间的相关性。反向突变仅用于训练和验证,不包含在测试集中。使用10折交叉验证,共训练了由10个模型组成的集成,每个模型训练500个epoch。红色趋势线为最小二乘多项式拟合结果。b,训练–验证–测试CDR序列一致性阈值对Graphinity性能的影响。该图的误差棒表示10个折之间的标准差,对应补充图2。c,在Synthetic_FoldX_ΔΔG_942723数据集上,施加90%长度匹配的CDR序列一致性阈值进行训练–验证–测试划分时,Graphinity预测值与真实合成值之间的相关性。采用10折交叉验证,训练10个epoch,由10个模型组成集成。蓝色趋势线为最小二乘多项式拟合结果。d,c中真实与预测FoldX ΔΔG值的直方图(为清晰起见,x轴范围限制在−8到+5 kcal mol−1)。实线表示核密度估计。a和c中给出了皮尔逊相关系数r。
2.3 使用约一百万突变的合成数据集
实验数据规模有限导致模型性能缺乏鲁棒性,促使研究者进一步研究在数据量显著增加的情况下ΔΔG预测的可行性。为此,研究者使用FoldX和Rosetta Flex ddG构建了更大规模的合成ΔΔG数据集。由于计算成本限制,Flex ddG可建模的突变数量较少,因此后续分析主要集中在FoldX数据集上。通过对结构已解析的抗体–抗原复合物界面进行穷举突变,研究者利用FoldX生成了近一百万个ΔΔG数据点。FoldX基于物理方程和经验测量来预测结合亲和力变化,尽管该合成数据集无法完全复现实验ΔΔG的复杂性,但仍捕捉了分子相互作用的关键特征。在AB-Bind数据集上,FoldX预测值与实验值的皮尔逊相关系数为0.34,对于对结合亲和力影响较大的突变,预测精度更高。当绝对ΔΔG值大于1 kcal mol−1时,用于判断突变是否稳定的ROC曲线下面积达到0.87,表明这些合成数据在一定程度上反映了实验特性。
在该合成数据集上,Graphinity在10折交叉验证并施加90%长度匹配CDR序列一致性阈值的条件下,测试集皮尔逊相关系数达到0.89。将训练轮数从10轮增加到100轮后,在单个折上的相关性略微提升至0.92,但由于计算成本较高,未进一步深入分析。与其他方法相比,Graphinity在该数据集上的ΔΔG预测性能最优。一个简单基线方法,即比较WT与突变结构之间接触数的变化,其相关性仅为0.42。研究者还评估了多种机器学习方法和不同输入形式,结果显示基于图的模型表现最强,其中EGNN和Equiformer的相关性分别达到0.87和0.89。由于EGNN在内存和训练时间方面更为高效,后续分析均基于该架构展开。
Graphinity的性能在多种训练–验证–测试序列一致性划分条件下都保持稳定。即使在最严格的划分条件下,即CDR和抗原序列一致性均限制在70%,皮尔逊相关系数仍维持在0.89。然而,当按亲和力进行数据划分时,模型相关性仅为0.52,反映了在域外预测方面的固有困难。进一步使用斯皮尔曼秩相关系数评估模型性能时,其数值约为0.64,低于皮尔逊相关系数。这主要是由于ΔΔG值在0附近高度密集,模型在该区间内的排序能力有限。当排除−1到+1 kcal mol−1之间的数值后,斯皮尔曼相关系数上升至约0.74。FoldX在预测对结合亲和力影响较小的突变时本身精度较低,因此该区域内的数据信号可能较弱。此外,合成数据集中还包含一些高度破坏性的突变,其ΔΔG值低于当前实验手段可准确测量的范围。将测试集限制为ΔΔG大于−12.2 kcal mol−1的突变后,测试集皮尔逊相关系数略降至0.78。
研究者还考察了不同图输入方式对模型性能的影响。将输入范围扩展到整个界面而非仅限于突变位点邻域时,模型性能仍然保持在较高水平。在一项初步分析中,研究者使用预测结构作为输入,将未经额外训练的Graphinity应用于100个随机选取突变的建模结构数据集,其预测性能几乎为零,这与当前抗体–抗原复合物结构建模仍面临的挑战相一致。总体而言,Graphinity在大规模合成数据集上的成功应用为一个概念验证,表明在数据量充足的条件下,ΔΔG是可以被准确预测的。
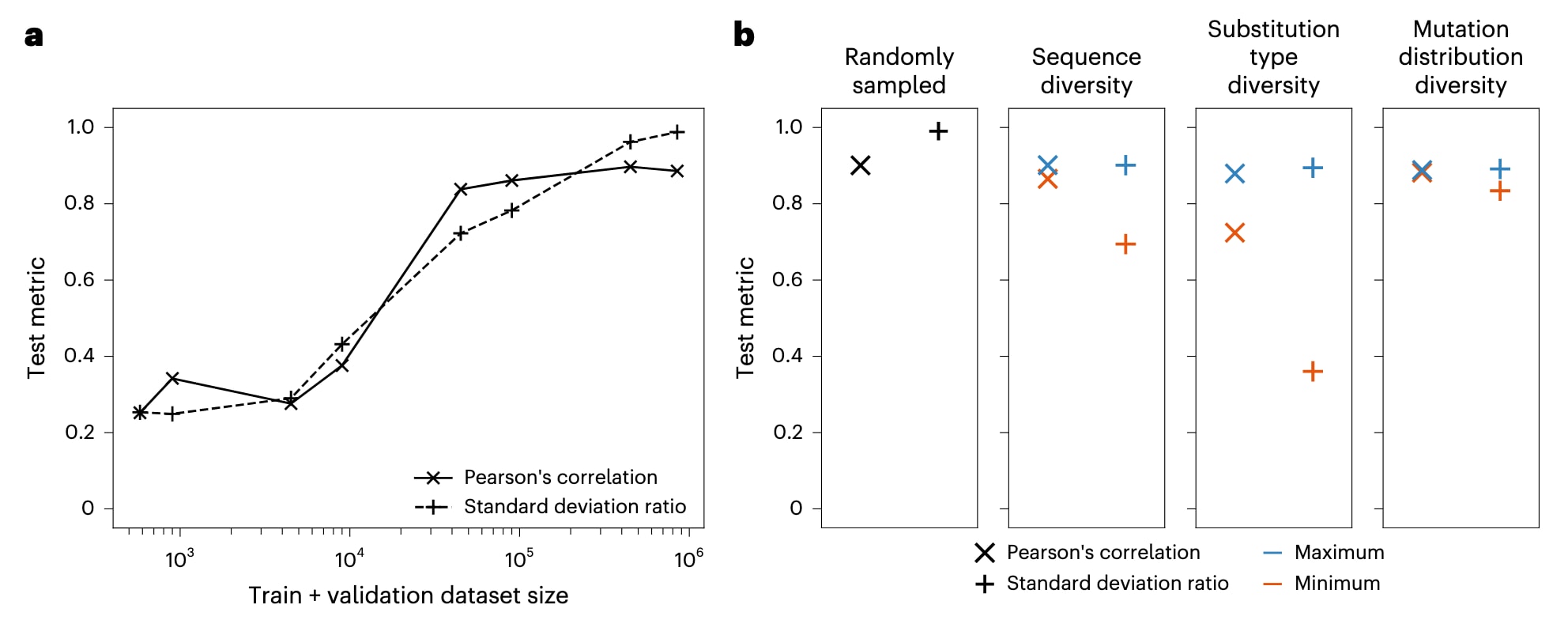
图3|从机器学习预测能力角度出发的实验ΔΔG数据集构建考量。 a,b,模型性能随训练集加验证集规模变化(a,数据集: Synthetic_FoldX_ΔΔG_{580-450000})以及随数据集多样性变化(b,数据集: Synthetic_FoldX_ΔΔG_100000_randomly_sampled, Synthetic_FoldX_ΔΔG_100000_{sequence/substitution_type/substitution_distribution}_{min/max})的情况。在b中,考虑了抗体CDR序列一致性、氨基酸替换类型频率以及突变位点在复合物界面中的分布这三种多样性因素。
2.4 实验ΔΔG数据集生成中的数据规模
在证明EGNN架构在训练数据充足时具有预测ΔΔG的潜力之后,研究者进一步尝试量化准确预测实验ΔΔG值所需的数据规模。研究者构建了不同训练集加验证集规模的模型,并在包含94,126个突变的测试集上进行评估,测试集中施加了90%长度匹配的CDR序列一致性阈值。结果显示,只有当训练数据量达到至少90,000个突变时,测试集皮尔逊相关系数才开始趋于平台,并达到约0.85(图3a)。在比较预测值与真实值的分布时可以观察到,由较小数据集训练得到的模型往往向均值回归,尽管相关系数较高,但预测值并未覆盖真实值的完整范围。为量化这一现象,研究者计算了标准差比值,即真实值与预测值标准差的相对比率(较小值除以较大值,标准差比<1)。只有当数据集规模达到450,000个突变时,该比值才超过0.8(图3a)。由于数据需求的估计会受到机器学习模型本身以及合成数据集性质的影响,研究者还采用其他方法进行了验证。将Equiformer架构应用于不同规模的训练数据子集后,观察到与EGNN相似的性能趋势。
此外,研究者还使用Rosetta Flex ddG生成了另一套合成数据集。Flex ddG在单个突变上的计算时间显著更长,从而限制了可建模的突变数量,但它提供了一种替代性的、基于物理参数化的方法来探索模型性能与数据需求。Flex ddG与实验值的相关性高于FoldX,尽管两者都能够反映蛋白–蛋白相互作用的物理特性,且精度均存在一定局限。在包含20,829个Flex ddG突变的数据集上,模型性能略低于FoldX数据集,但整体趋势一致。这些结果支持这样一个结论:要实现准确且具有泛化能力的ΔΔG预测,仍然需要比当前多出数个数量级的实验数据。
2.5 实验ΔΔG数据集生成中的数据多样性
多样性是任何用于模型训练的数据集的重要特征。研究者通过三种指标评估数据集多样性的作用:抗体序列多样性、氨基酸替换类型多样性以及界面突变的结构分布多样性。研究者在FoldX数据集中分别构建了在上述指标上最小化和最大化的训练与验证数据集。例如,在最小序列多样性的数据集中,突变仅来自75个抗体–抗原复合物,而在最大多样性的数据集中,突变来自1,177个复合物。所有模型均在同一测试集上进行评估,该测试集包含10,000个突变,并施加了90%长度匹配的CDR序列一致性阈值。结果表明,界面突变的结构分布对模型性能影响较小,这可能与输入图仅表示突变位点邻域有关。然而,序列多样性和替换类型多样性对模型性能具有显著影响,尤其体现在测试集标准差比值上。与对应的最大多样性数据集相比,最小序列多样性和最小替换类型多样性数据集的标准差比值分别降低了23%和60%。
2.6 大规模合成ΔΔG数据集中的噪声影响
实验ΔΔG数据通常存在噪声,尤其是在来自不同实验体系或实验室时。研究者通过两种方式评估Graphinity对噪声的鲁棒性:一是随机打乱突变对应的亲和力标签,二是在标签中加入高斯分布的随机噪声。在0%到60%标签被打乱的情况下,保留测试集上的皮尔逊相关系数仍保持在约0.85(图4b)。然而,进一步分析预测值与真实FoldX ΔΔG值的分布发现,随着打乱比例增加,模型的预测分布逐渐收缩,预测能力下降。当100%的标签被打乱时,模型性能降为0,这表明尽管FoldX生成的数值不如实验数据精确,但其中确实包含可从复合物结构中学习到的真实信号。在SKEMPI 2.0中存在82组重复的抗体–抗原单点突变,其ΔΔG标准差平均为0.19 kcal mol−1,最大为0.90 kcal mol−1。在这一噪声范围内,甚至在高斯噪声尺度达到5时,Graphinity仍能保持皮尔逊相关系数和标准差比值高于0.8(图4c)。
2.7 按氨基酸替换类型的模型性能
研究者进一步分析了模型在特定氨基酸替换类型上的表现,例如Arg到Lys。实验值与预测值在平均ΔΔG及其标准差上的整体模式高度一致,表明模型能够学习到结构上下文信息。特定替换类型的平均ΔΔG预测值与真实值之间的皮尔逊相关系数仅为0.35,明显低于完整模型的预测性能0.89。对于同一种替换类型,FoldX ΔΔG值的变化范围很大,标准差介于0.5到10.6 kcal mol−1之间。某些突变,例如从Gly出发的突变或突变为Phe、His、Trp或Tyr,表现出更低的平均ΔΔG值和更高的标准差,这与小氨基酸被较大残基替换时可能产生的破坏性效应一致。EGNN模型在这些突变上的预测性能更高。未来的实验数据生成应更多覆盖那些预测难度较大的突变类型,例如向小残基的突变。
2.8 在实验结合数据集上的Graphinity测试
为检验Graphinity是否能够学习实验数据的分布,研究者将该架构调整并应用于一个包含36,391个曲妥珠单抗CDRH3变体的数据集。该数据集将变体按是否与抗原HER2结合进行分类。由于仅涉及单一抗原,这一任务相对简单,也并非Graphinity架构的主要设计目标,但数据规模足够大,预期应能够实现有效预测。模型成功地区分了结合型与非结合型变体,ROC AUC达到0.90,平均精度为0.82。这一性能接近此前应用于该数据集的基于序列的CNN方法。进一步地,在施加V基因和J基因克隆型以及CDRH3序列一致性阈值后,模型性能依然稳健,ROC AUC始终保持在0.90以上。
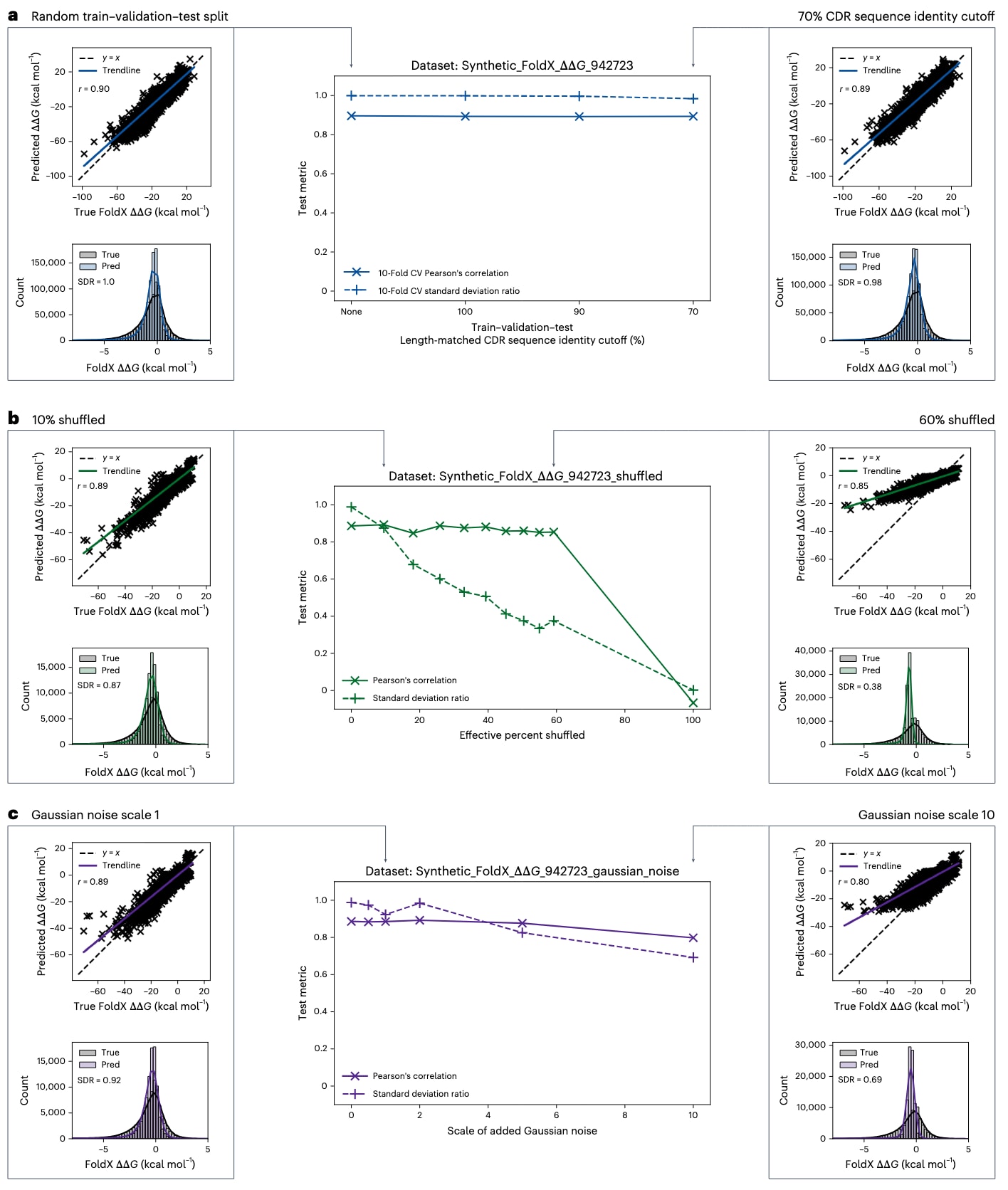
**图4|模型对训练–验证–测试划分与噪声的鲁棒性。**a–c,训练–验证–测试划分阈值(a)、标签打乱(Synthetic_FoldX_ΔΔG_942723_shuffled) (b)以及高斯噪声(Synthetic_FoldX_ΔΔG_942723_gaussian_noise) (c)对模型性能的影响。a中结果基于10折交叉验证,b和c中结果基于单一折的独立测试集。a–c中的散点图给出了皮尔逊相关系数r。所有直方图中,x轴均限制在−8到+5 kcal mol−1范围内,实线表示核密度估计。Pred表示预测值,SDR表示标准差比值。
3 讨论
抗原结合亲和力对抗体的功能和疗效至关重要,其本身具有高度复杂性,在计算上也极具挑战。机器学习模型在诸如AB-Bind这类规模较小的实验数据集上,采用随机训练–测试划分时往往能够取得较高的预测性能。然而,这些较高的相关性主要源于过拟合,当面对与训练数据差异较大的复合物时,模型性能并不能泛化。为避免信息泄漏,在训练集与测试集之间施加有效的序列一致性阈值至关重要。为检验在数据量显著增加的情况下亲和力是否能够被准确且稳健地预测,研究者将机器学习方法应用于一个由FoldX生成、包含近一百万个突变的合成数据集。通过评估多种基于序列和结构输入的模型架构,验证了基于图的深度学习方法在ΔΔG预测中的适用性。Graphinity的EGNN模型在严格限制抗体和抗原训练–测试序列一致性的条件下,以及在接近实验数据噪声水平的情况下,依然保持了较高的相关性。
需要注意的是,合成数据的结果必须结合其数据来源进行解读。这些数据均由同一软件生成,因此相较于实验数据,其内部一致性更高、噪声更低,且其分布形式也可能不同于真实实验值。然而,FoldX能够较为准确地区分对结合亲和力具有显著影响的突变是稳定的还是不稳定的,这表明该数据集中确实包含可学习的信号。研究者还通过将Graphinity应用于一个包含超过36,000个曲妥珠单抗变体的实验数据集,验证了该架构不仅能够学习合成数据的分布,也能够学习实验数据的分布。EGNN模型在该任务上的性能与此前的CNN方法相当,但在潜在的泛化能力方面具有进一步优势,尤其是在不同抗体–抗原复合物之间的应用潜力。
机器学习在大规模数据集上的成功应用支持这样一种观点:实验ΔΔG预测面临的主要瓶颈并非模型架构本身,而是数据的可获得性。研究者利用合成数据系统性地探索了实现准确且具有泛化能力的实验ΔΔG预测所需的数据规模。基于EGNN和Equiformer架构,以及FoldX和Flex ddG数据集的结果表明,当前可用的实验数据远远不足,很可能需要多出数个数量级的数据,即数万到数十万条数据点。在该研究所测试的条件下,估计至少需要90,000个ΔΔG数据点,才能使测试集皮尔逊相关系数超过0.85。随着实验方法通量的提升,如此规模的数据在未来将更易获得。然而,所需数据量的具体数值仍取决于数据集的多样性和所采用的机器学习方法,并可能随着研究进展而发生变化。随着数据的不断积累,也有可能通过机器学习层面的技术手段在一定程度上弥补数据不足,例如设计对数据需求更低的模型架构、采用分层采样策略,或利用迁移学习从相关的高数据量任务或合成数据中获益。未来的模型设计还可以纳入当前方法中通常忽略的生理因素,例如水分子以及蛋白构象柔性。
除数据规模之外,研究者还发现数据集多样性同样是一个关键因素,尤其体现在抗体序列多样性和氨基酸替换类型多样性方面。而这两类多样性在现有实验数据中都极为有限。例如,SKEMPI 2.0中的抗体–抗原单点突变数据来源于不足50个复合物,且在替换类型上高度偏倚,其中超过一半为向丙氨酸的突变。研究者的结果强调,需要向“机器学习级”数据转变,即在数据生成过程中引入模型开发的反馈。在此基础上,若要推动具有泛化能力的亲和力预测,还应重点考虑以下方向:提高亲和力测量实验方法的通量,设计具有高度多样性且结构良好的数据集,建立标准化且定期更新的ΔΔG数据与元数据仓库,以及实施稳健的盲测评估以检验模型性能。这些措施将有助于提升机器学习在抗体–抗原亲和力预测中的可靠性。