Science 2026 | DrugCLIP: 深度对比学习实现全基因组尺度的虚拟筛选
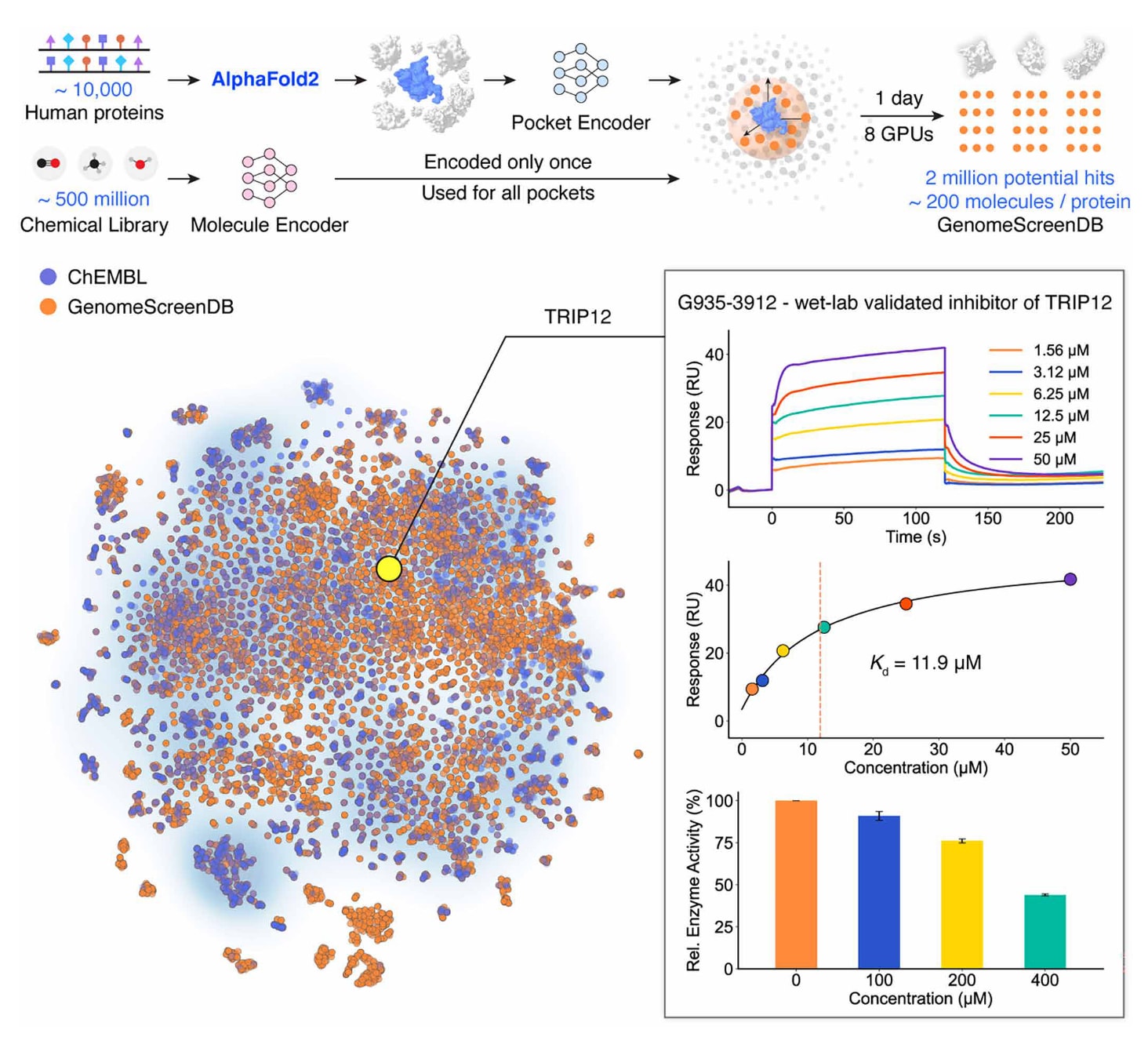
DrugCLIP实现了超高速的全基因组虚拟筛选。 该方法基于约1万个由AlphaFold预测的人类蛋白结构,对包含5亿个化合物的分子库进行了全基因组尺度的虚拟筛选,仅使用8块GPU便在1天内完成。由此构建的数据库GenomeScreenDB在靶点覆盖范围上超过了ChEMBL数据库。针对TRIP12的筛选结果通过实验得到了验证,成功鉴定出具有功能性的小分子结合物。Kd表示解离常数,RU表示响应单位。
Preface
A. Introduction
人类可成药基因组中仍有相当一部分尚未被小分子治疗药物成功靶向。随着以AlphaFold为代表的蛋白质结构预测技术不断发展,在全基因组尺度上开展药物发现已逐渐成为可实现的目标。然而,现有虚拟筛选工具远不能满足这一需求。无论是经典的分子对接方法,还是近年来兴起的深度学习方法,其计算开销都过于庞大,难以覆盖全基因组范围的靶点。基于此,该研究旨在开发一种高效的全基因组虚拟筛选方法,能够快速为人类基因组中所有可成药靶点识别潜在的小分子配体。
B. Rationale
研究提出了DrugCLIP,一种用于快速且高精度虚拟筛选的对比学习框架。DrugCLIP将蛋白质口袋与小分子共同编码到同一潜在空间中,并结合大规模合成数据以及实验解析的蛋白-配体复合物结构进行训练。在此基础上,可借助类似现代搜索引擎的稠密检索技术,将大规模化合物库高效查询于给定蛋白靶点。为提升其在AlphaFold预测结构上的适用性,研究还提出了GenPack,一种生成式口袋精修模块,用于提高结合口袋识别的准确性。DrugCLIP的性能通过标准基准数据集与湿实验验证进行了系统评估,并进一步开展了全基因组尺度的虚拟筛选研究,所有结果均对外公开。
C. Results
在DUD-E和LIT-PCBA这两个广泛使用的虚拟筛选基准数据集上,DrugCLIP在速度和准确性方面均优于传统分子对接方法及当前最先进的深度学习模型。该方法在不同化学骨架和蛋白家族之间表现出良好的泛化能力,同时对结构扰动具有较强鲁棒性。在实验验证中,DrugCLIP成功识别了血清素2A受体和去甲肾上腺素转运体的高效配体,这两个靶点均与精神类疾病密切相关。其中,两个血清素2A受体激动剂的半数有效浓度低于100 nM,两种去甲肾上腺素转运体抑制剂则通过冷冻电镜结构得到验证。在结合GenPack后,DrugCLIP在具有挑战性的apo结构及AlphaFold预测结构上显著优于常规对接和诱导契合对接方法。进一步地,该框架还成功用于一个研究较少的靶点甲状腺激素受体相互作用蛋白12,该靶点此前既无已知全长结构,也无已报道配体。模型在表面等离子共振实验中获得了17.5%的命中率,并有两种抑制剂在酶学实验中得到进一步确认。最后,研究将DrugCLIP应用于全基因组虚拟筛选,针对约1万种人类蛋白与5亿个化合物进行匹配,在仅使用8块GPU的情况下,于24小时内完成了超过10万亿个蛋白-配体对的评分。该筛选共获得200多万个候选分子,覆盖约2万个结合口袋,约占人类基因组的一半,所有筛选数据均已公开以支持更广泛的药物研发应用。
D. Conclusion
DrugCLIP是一种经过系统计算评测与湿实验严格验证的超高速虚拟筛选方法。其计算效率使得在万亿规模上覆盖整个人类可成药蛋白组成为可能,并以开放数据资源的形式为下一代药物发现奠定基础,尤其适用于当前认知有限的潜在靶点。

获取详情及资源:
- 📄 论文: https://doi.org/10.1126/science.ads9530
- 💻 代码: https://github.com/THU-ATOM/DrugCLIP_screen_pipeline
- 🛜 网站: https://drugclip.com
0 摘要
近期蛋白质结构预测领域的突破为全基因组尺度的药物发现开辟了新的路径,但现有虚拟筛选方法在计算开销上仍然难以承受。研究提出了DrugCLIP,一种基于对比学习的框架,能够实现超高速且高精度的虚拟筛选,在速度上相较分子对接最高可提升一千万倍,并在多种计算基准测试中持续优于不同对照方法。在湿实验验证中,DrugCLIP在去甲肾上腺素转运体靶点上取得了15%的命中率,并解析了两种已识别抑制剂与靶蛋白形成复合物的结构。对于缺乏全长结构和已知小分子配体的甲状腺激素受体相互作用蛋白12,DrugCLIP仅依赖AlphaFold2预测结构便实现了17.5%的命中率。最后,研究发布了GenomeScreenDB这一开放数据库,提供约1万种人类蛋白与5亿个化合物的预计算筛选结果,为后AlphaFold时代的药物发现范式奠定了基础。
1 引言
人类基因组由约2万个蛋白编码基因组成,其中许多与多种疾病密切相关。尽管如此,目前只有大约10%的基因被美国食品药品监督管理局批准的药物成功靶向,或在文献中已有小分子结合物报道。这意味着相当大一部分潜在可成药基因组仍基本未被探索,为治疗手段的创新提供了广阔空间。科研界普遍希望将具有生物学意义的靶点转化为真正的药物突破。然而,多数研究者既难以获得先进的高通量筛选设备,也缺乏足够的计算资源来开展全面的虚拟筛选。此外,蛋白质通常以家族或通路的形式发挥功能,单一靶点并不总是最有效的干预策略。这些限制显著降低了药物发现的成功率,尤其是在新靶点研究中。因此,构建一个包含全基因组虚拟筛选结果的综合化学数据库,将成为生物医学研究领域的重要资源,并有望显著加速新药发现进程。
鉴于对所有人类蛋白开展实验筛选在时间和经济成本上的巨大负担,虚拟筛选逐渐成为应对海量潜在靶点的更现实选择。在传统的计算机辅助药物设计中,分子对接是基于靶点虚拟筛选的核心技术。尽管在简化评分函数、算法优化以及硬件加速方面取得了进展,分子对接仍然十分耗时,通常需要数秒到数分钟才能评估一个蛋白-配体对。例如,近期一项大规模对接研究即便使用了1万核CPU,也耗时两周才完成针对单一靶点的10亿分子筛选。因此,在现有技术条件下,全基因组尺度的虚拟筛选在计算上几乎不可行。
人工智能为药物发现带来了新的机遇。近年来,多种深度学习方法被用于虚拟筛选,主要聚焦于预测配体与受体之间的亲和力。然而,将这些方法直接应用于大规模虚拟筛选仍面临显著挑战。首先,由于实验条件的异质性,亲和力数值本身存在不一致性,这可能削弱模型训练效果。其次,训练数据集与真实应用场景之间存在明显的分布偏移,现实中的虚拟筛选往往包含比例更高的非活性分子,从而限制了模型的泛化能力。此外,深度学习模型通常包含数百万参数,其推理阶段的计算开销成为速度瓶颈,尤其是在化合物库和靶点数量不断扩大的情况下。因此,亟需开发更加高效且稳健的人工智能方法来系统性地应对这些问题。
在该研究中,提出了DrugCLIP,一种基于对比学习的虚拟筛选方法,通过对齐蛋白口袋与配体的表示,实现对强结合物与非结合物的有效区分。对比学习已在多个领域展现出显著优势,DrugCLIP将这一思想成功引入药物发现任务中。该方法不依赖于结合口袋的精确局部几何信息,即使仅使用apo结构或计算预测结构,也能够进行结合预测。在DUD-E和LIT-PCBA这两个广泛使用的虚拟筛选基准数据集上,DrugCLIP表现出具有竞争力的性能,优于多种传统对接方法和神经网络模型。更重要的是,该方法在真实应用中成功识别并实验验证了血清素2A受体、去甲肾上腺素转运体以及甲状腺激素受体相互作用蛋白12的结合物,其中后者此前既无已报道抑制剂,也缺乏实验解析的全长复合物结构。
最后,研究利用DrugCLIP对AlphaFold2预测的人类蛋白结构开展了全基因组虚拟筛选,结合口袋检测算法与生成式人工智能模型定义结合位点,并对超过5亿个公开化合物进行了筛选。整个筛选过程在8块GPU上于24小时内完成,累计执行了超过10万亿次蛋白-配体评分计算。由此构建的GenomeScreenDB数据库包含来自约1万种蛋白、2万多个结合口袋的200多万个潜在命中分子,覆盖了近一半的人类基因组。所有分子、评分结果及构象信息均已在https://drugclip.com公开,以支持后续研究与应用。
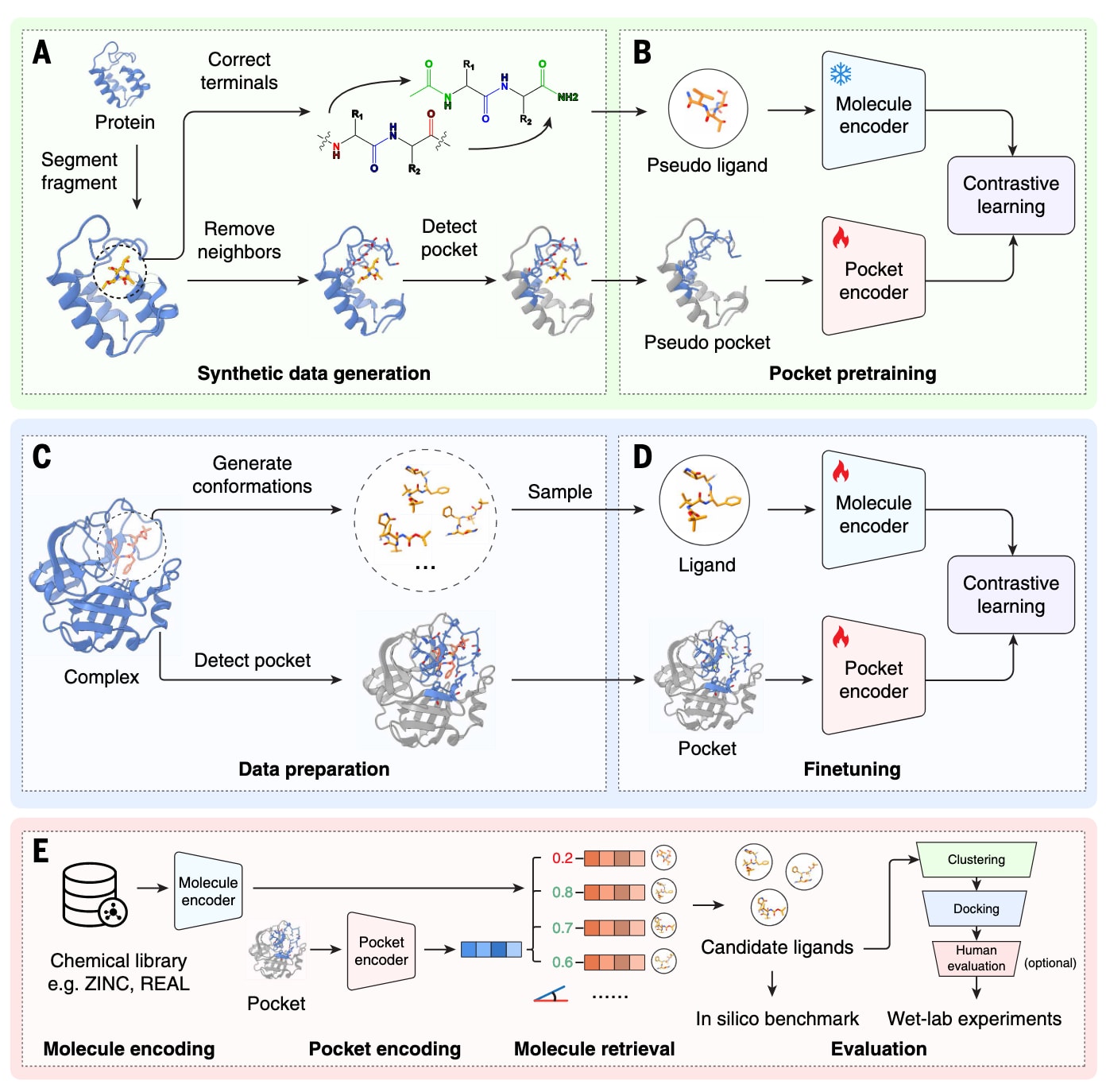
图1|DrugCLIP的整体框架。 (A) 预训练阶段采用基于ProFSA策略构建的大规模合成数据集。具体而言,通过对蛋白结构数据进行片段切分、末端修正、邻域去除以及口袋检测等一系列操作,构建伪口袋-配体对。(B) 口袋编码器在对比蒸馏范式下,利用伪口袋-配体对进行预训练,将成熟分子编码器中的知识迁移至口袋编码器。(C) 在微调过程中,采用实验解析的蛋白-配体对作为训练数据,并使用RDKit生成多种配体构象。(D) 微调阶段中,口袋编码器与分子编码器同时通过对比损失进行更新,该损失函数最大化正样本对之间的相似度,并最小化负样本对之间的相似度。(E) 基于DrugCLIP的虚拟筛选流程。首先使用训练好的分子编码器对化合物库中的候选分子进行预编码。对于给定的蛋白口袋,利用训练好的口袋编码器将其表示为向量,随后通过计算余弦相似度筛选得分最高的配体。在进入湿实验之前,还需对筛选结果依次进行聚类、分子对接以及人工评估。
2 DrugCLIP模型的设计
不同于以往依赖回归模型直接预测蛋白-配体亲和力数值的机器学习方法,DrugCLIP重新将虚拟筛选定义为一个稠密检索任务。如图1所示,其核心创新在于训练目标的设定,即通过两个相互独立的神经网络,分别对蛋白结合口袋和小分子进行编码,并学习一个对齐的嵌入空间,使得向量相似度能够反映二者的结合概率。在训练过程中,模型采用对比损失函数,最大化蛋白口袋与其真实结合物之间的相似度,同时最小化蛋白口袋与结合于其他靶点分子的相似度。
DrugCLIP的训练流程分为预训练和微调两个阶段。分子编码器和口袋编码器首先在大规模合成数据上进行预训练,随后再利用实验解析的蛋白-配体复合物结构进行微调。在预训练阶段,分子编码器采用成熟的小分子表示模型Uni-Mol进行初始化,并在训练中保持参数冻结。口袋编码器则随机初始化,通过对比学习与分子编码器对齐。如图1A所示,研究提出了蛋白片段-环境对齐框架ProFSA,用于构建专门服务于对比预训练的大规模合成数据。在该框架中,从仅包含蛋白的结构中截取短肽片段作为伪配体,并将其周围区域定义为伪结合口袋。蛋白内部相互作用在多方面与蛋白-配体相互作用相似,包括氢键、离子相互作用、π-π堆积以及其他非共价作用。以往在配体结合蛋白设计研究中,也曾利用蛋白内部堆积信息来确定化学基团相对于主链的统计偏好取向,从而辅助蛋白-配体界面建模,这一思想构成了ProFSA的理论基础。
为进一步提升模型性能,研究对伪配体和伪结合口袋的化学性质分布进行了精细校准,使其尽可能接近真实蛋白-配体复合物中的分布特征,从而缩小合成数据与真实数据之间的差距。相关技术细节在材料与方法部分的口袋编码器预训练章节中给出。将ProFSA框架应用于蛋白质结构数据库后,共生成约550万个伪口袋-配体对用于预训练。预训练得到的口袋编码器在多种下游任务中进行了评估,包括口袋性质预测、口袋匹配以及蛋白-配体亲和力预测。实验结果表明,即使在零样本条件下,该口袋编码器依然展现出优异性能,超过了多种基于监督学习的方法以及基于物理或知识的模型,充分说明预训练阶段成功学习到了具有生物学意义的口袋表示。
在完成预训练后,模型进一步使用BioLip2数据库中收集的4万个实验解析的蛋白-配体复合物结构进行微调。如图1D所示,考虑到虚拟筛选中通常无法获得分子的真实结合构象,仅已知其拓扑结构,研究采用RDKit生成随机构象进行数据增强,以更真实地模拟实际筛选场景中的构象多样性。这一策略显著提升了DrugCLIP在性能和泛化能力上的表现。
在实际筛选阶段,模型首先将分子和蛋白口袋编码为向量表示,随后计算二者之间的余弦相似度,并据此对候选分子进行排序。该排序结果可直接用于计算基准评测,同时在进入湿实验之前,还会结合聚类、分子对接以及人工评估等步骤进行进一步筛选。由于分子表示可以提前离线计算,DrugCLIP在筛选过程中仅需进行简单的余弦相似度计算和排序,因此具有极高的计算效率。在合理的预编码和并行化条件下,DrugCLIP仅凭单块GPU即可完成万亿级别的靶点-分子配对评估,其速度相比传统分子对接等计算方法提升超过一千万倍。
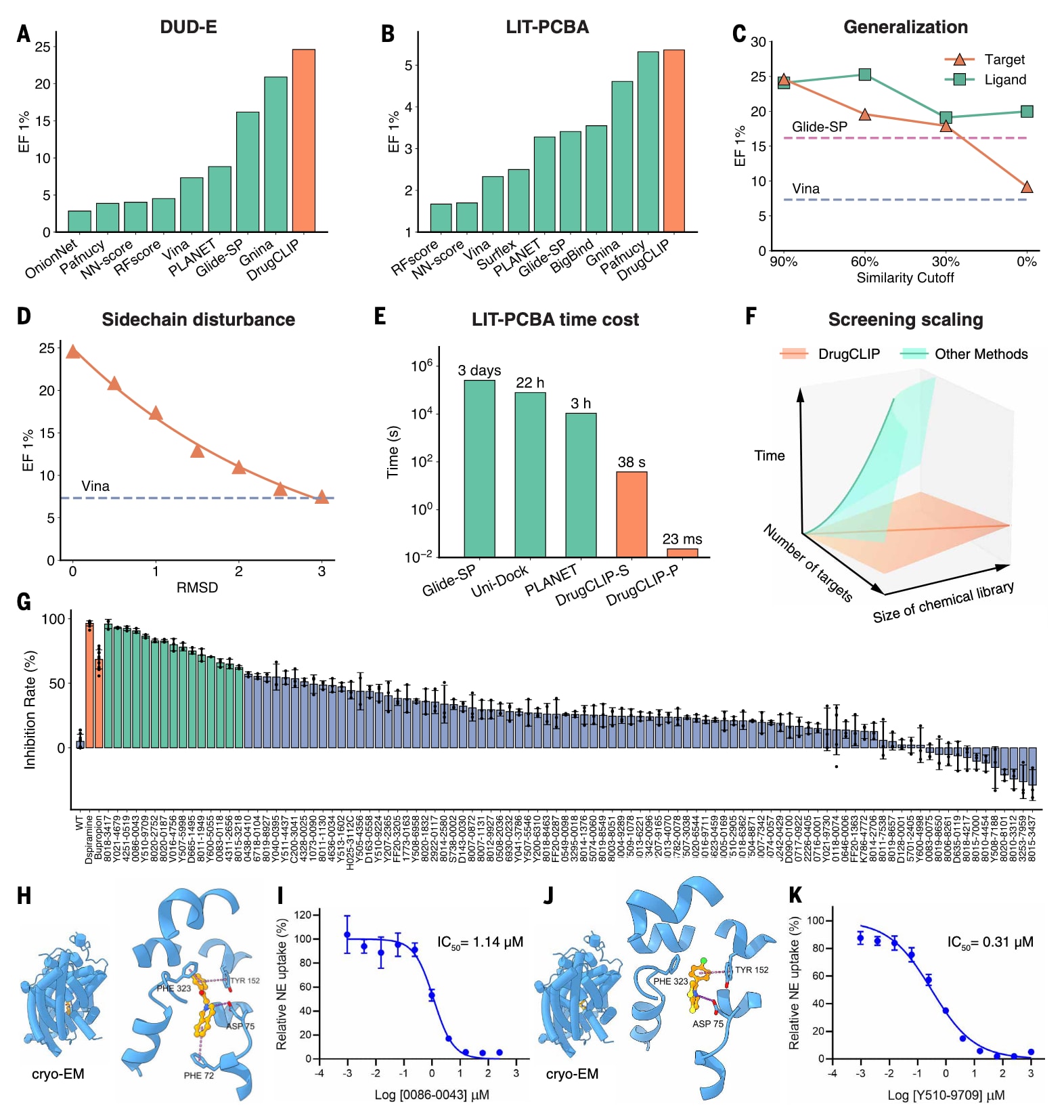
图2|DrugCLIP的计算基准评测结果及其在NET靶点上的湿实验验证。 (A) 在DUD-E数据集上对DrugCLIP进行评估,采用EF1%指标衡量模型性能,对照模型的结果引自既有研究。(B) 在LIT-PCBA数据集上的评估结果,同样使用EF1%作为性能指标,基线模型结果来源于既有工作。(C) 通过改变DUD-E数据集中测试靶点或分子与训练数据之间的相似性阈值,评估DrugCLIP的泛化能力,其中Glide-SP和Vina以虚线表示。蛋白相似性阈值30%、60%和90%由MMSeqs2计算,0%表示利用HMMER和PFAM完全移除对应蛋白家族。分子相似性阈值30%、60%和90%基于Morgan2指纹计算,0%表示基于通用Murcko骨架移除整个分子系列。(D) 通过在蛋白口袋侧链构象中引入0至3 Å的RMSD误差,评估DrugCLIP对结构误差的鲁棒性,Vina结果以虚线作为参考。(E) 在LIT-PCBA数据集上的筛选速度比较,包括Glide-SP、Uni-Dock等对接方法以及机器学习模型PLANET。基线方法的速度数据引自既有研究。Glide-SP的时间消耗基于128核CPU换算得到,以弥补原研究中16核CPU设置与现代GPU之间的不可比性。Uni-Dock的时间消耗估计为在8块GPU上每个配体0.04 s。对于DrugCLIP,在A100 GPU上以顺序计算方式对全部LIT-PCBA靶点进行筛选共耗时38 s,这是由于该数据集中的分子和口袋数量较少,无法充分利用GPU并行能力。因此,同时报告了并行计算情形下的速度,即在A100 GPU上对100,000个口袋筛选1,000万个分子,耗时约25 min。在该设置下,完成与LIT-PCBA等量计算仅需0.023 s。(F) 随着筛选规模增加的时间消耗示意图,x轴表示化合物库规模,y轴表示靶点数量,z轴表示虚拟筛选的时间成本。DrugCLIP仅需对网络前向传播M+N次,分别对应M个靶点和N个分子,而多数现有方法需要对每一对靶点-分子组合运行一次网络或对接流程,总计M×N次。(G) 对DrugCLIP筛选得到的100个候选化合物在10 μM浓度下进行NET放射性配体转运抑制实验评估,其中15个化合物的抑制率超过60%。每个化合物的实验均包含3个生物学重复,结果以均值±SD表示。(H) 化合物0086-0043与NET的复合物结构通过冷冻电镜解析。(I) 化合物0086-0043在放射性配体转运实验中的剂量-反应曲线,每个浓度点均进行3个生物学重复,结果以均值±SEM表示,NE表示去甲肾上腺素。(J) 化合物Y510-9709与NET的复合物结构通过冷冻电镜解析。(K) 化合物Y510-9709在放射性配体转运实验中的剂量-反应曲线,每个浓度点均进行3个生物学重复,结果以均值±SEM表示。
3 基准评测与湿实验验证DrugCLIP的性能
研究首先在DUD-E和LIT-PCBA两个基准数据集上系统评估了DrugCLIP的性能。DUD-E数据集包含102个蛋白靶点对应的22,886个活性化合物,并为每个活性分子构建了50个在物理性质上相似但结构不同的诱饵分子。相比之下,LIT-PCBA来源于PubChem BioAssay数据库的实验结果,覆盖15个靶点,包含约8000个活性分子和264万个非活性分子。研究将DrugCLIP与多种成熟的虚拟筛选方法进行了对比,包括基于物理信息的分子对接软件以及以回归为导向的机器学习模型。在所有评测结果中,DrugCLIP在EF1%这一衡量虚拟筛选召回能力的关键指标上均优于所有对照方法。
进一步分析显示,在移除训练集中与测试集具有相似分子子结构或骨架的样本后,DrugCLIP的性能仅出现轻微下降,并且仍然稳定优于广泛使用的商业对接软件。这种稳健性不仅体现在对未知分子结构的适应能力上,在面对全新蛋白家族时同样表现突出。当测试蛋白家族在训练阶段被完全排除后,DrugCLIP依然优于常用的分子对接方法,体现出其对新靶点的强泛化能力。此外,即便在蛋白口袋侧链构象存在3 Å均方根偏差的情况下,DrugCLIP仍能保持优异表现,表明其对结构误差具有很强的鲁棒性。
在计算效率方面,DrugCLIP仅依赖向量点积完成评分,具有显著优势。例如,在顺序计算模式下,DrugCLIP仅需38 s即可完成LIT-PCBA数据集的筛选,而传统对接和其他机器学习方法往往需要数小时甚至数天。借助GPU并行计算,在同时评估大量分子和口袋时,相同规模的计算可进一步压缩至0.023 s。同时,DrugCLIP的计算时间随靶点数量和分子数量的增加呈线性扩展,非常适合多靶点虚拟筛选任务。这些计算评测结果表明,DrugCLIP在性能、泛化性、鲁棒性和效率方面实现了有力结合。
除计算评测外,研究还通过湿实验对DrugCLIP在真实靶点上的表现进行了验证。实验选取了两个与精神类疾病密切相关的经典靶点,血清素2A受体和去甲肾上腺素转运体。血清素2A受体被认为是新型抗抑郁药的重要靶点,其激动剂在动物模型和人体中均表现出持久而显著的抗抑郁效果。已有研究表明,受体激活后β-arrestin2的招募是产生这些疗效的重要分子机制。在初步虚拟筛选实验中,研究从商业化合物库中选取了78个排名靠前的候选分子进行测试,其中8个在钙流实验中表现为阳性激动剂。进一步的放射性竞争结合实验显示,其中6个化合物的抑制常数低于10 μM。随后采用NanoBit体系评估其诱导β-arrestin2招募的细胞功能,这6个化合物的半数有效浓度均低于1 μM。其中性能最优的化合物亲和力达到21.0 nM,在NanoBit实验中的EC50为60.3 nM,最大效应达到35.8%。
在血清素2A受体取得成功后,研究将DrugCLIP应用于去甲肾上腺素转运体这一成熟药物靶点。尽管已有多种获批抑制剂,但该靶点与抑制剂形成复合物的结构直到2024年才被解析,且现有结构与数据集中最接近的蛋白相似度不足60%,因此该任务对模型的泛化能力提出了更高要求。研究最终选取了100个在化学新颖性和多样性上具有代表性的化合物,通过检测放射性标记去甲肾上腺素在含NET脂质体中的转运来评估抑制效果。结果显示,其中15%的化合物对NET的抑制率超过60%,且有12个化合物的效力优于常用抗抑郁药安非他酮。与以往通常依赖脂肪族氮原子形成盐桥相互作用的NET抑制剂不同,DrugCLIP筛选得到的多个命中分子含有带正电的芳香氮原子。其中两个代表性化合物在抑制效力上明显优于安非他酮。冷冻电镜解析的复合物结构表明,这些芳香环能够与NET中的芳香残基形成更有利的相互作用,为理解NET抑制机制提供了新的化学视角。这些结果进一步凸显了DrugCLIP在药物发现中挖掘新型化学空间和机制的潜力。
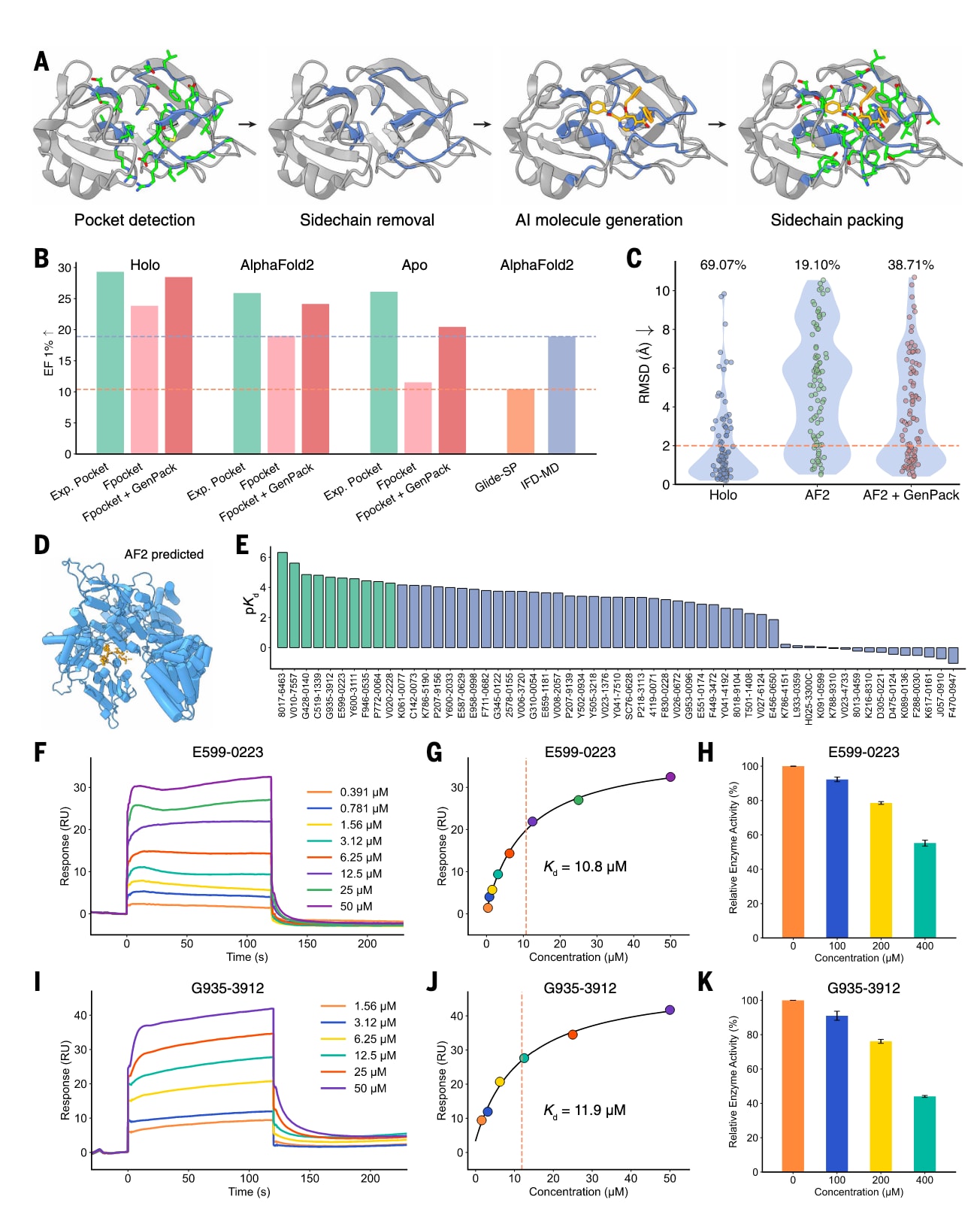
图3|在GenPack辅助下将DrugCLIP应用于AlphaFold预测结构。 (A) GenPack从AlphaFold2预测结构中提取结合口袋的流程,包括使用Fpocket检测初始口袋、去除侧链、基于主链结构应用生成式AI模型生成分子,随后在生成分子的条件下进行侧链重构。(B) 在DUD-E子集上对全配体结构、AlphaFold2预测结构以及apo结构进行虚拟筛选的EF1%比较,采用不同的口袋定义方式,包括与全配体结构对齐得到的口袋(Exp.Pocket)、Fpocket检测的口袋(Fpocket)以及经GenPack生成的口袋(Fpocket+GenPack)。Glide-SP和IFD-MD的性能作为参考。(C) 不同口袋定义方式下的重对接RMSD比较,包括全配体口袋、AlphaFold2预测结构上的口袋以及经GenPack精修后的AlphaFold2预测口袋。橙色虚线表示2 Å的RMSD阈值,对应的对接成功率标注于各柱状图上方,蓝色阴影区域表示RMSD分布的核密度估计。(D) TRIP12的AlphaFold2预测结构及用于DrugCLIP虚拟筛选的结合口袋位置(橙色点)。(E) 对57个筛选候选化合物进行单循环SPR初筛得到的pKd值,绿色表示Kd小于50 μM且在后续多循环SPR实验中得到验证的命中化合物。(F) 化合物E599-0223的多循环SPR实验传感曲线,RU表示响应单位。(G) 化合物E599-0223多循环SPR实验的稳态结合曲线。(H) 在不同浓度E599-0223条件下TRIP12的酶活性,每个浓度包含4个生物学重复,结果以均值±SEM表示。(I) 化合物G935-3912的多循环SPR实验传感曲线。(J) 化合物G935-3912多循环SPR实验的稳态结合曲线。(K) 在不同浓度G935-3912条件下TRIP12的酶活性,每个浓度包含4个生物学重复,结果以均值±SEM表示。
4 将DrugCLIP应用于AlphaFold预测结构
在通过计算评测和湿实验系统验证DrugCLIP模型之后,研究进一步将其应用于计算预测得到的蛋白质结构。近年来,蛋白质结构预测领域取得了突破性进展,尤其是AlphaFold2几乎覆盖了整个人类蛋白质组,以及AlphaFold3和RoseTTAFold All-Atom等全原子结构预测模型的出现,为大量缺乏实验结构数据的重要药物靶点提供了结构信息。这些进展为突破仅依赖实验解析结构的结构基础药物发现开辟了新的空间。
然而,基于AlphaFold预测结构进行虚拟筛选仍存在争议。主要顾虑在于预测结构可能无法充分复现实验构象,从而影响对非活性分子的有效过滤。尽管如此,已有研究表明,在部分靶点上使用AlphaFold预测结构进行虚拟筛选仍可获得合理结果。鉴于DrugCLIP对蛋白侧链构象误差具有较强鲁棒性,研究进一步评估了预测结构对模型性能的影响,采用了一个专门用于AlphaFold预测结构和apo结构虚拟筛选的DUD-E子集。
研究首先发现,只要能够通过与全配体结构的构象比对准确定义结合口袋,DrugCLIP即可较好地适应AlphaFold2预测结构或apo结构中固有的构象差异。对于缺乏同源结构的蛋白靶点,通常需要借助Fpocket等工具预测潜在结合口袋。在实验中,直接使用Fpocket结果会显著降低DrugCLIP的性能,EF1%由29.3%下降至19.0%,这一现象与分子对接和构象预测研究中观察到的挑战一致。
为提升AlphaFold预测结构在虚拟筛选中的实用性,研究提出了一种称为GenPack的生成-重构策略。该方法通过在蛋白口袋主链结构条件下训练分子生成模型,尽管生成的分子并不一定具备可合成性,但能够帮助更精确地定位结合口袋,并诱导口袋构象向更有利于结合的状态转变。随后,研究将侧链重新引入并结合物理力场对整体构象进行优化。借助GenPack,AlphaFold2预测结构在DUD-E子集上的EF1%由19.0%提升至24.1%,而对于apo结构,提升幅度更为显著,EF1%由11.5%提高至20.4%。与此前针对apo或AlphaFold预测结构的先进虚拟筛选方法相比,该策略在活性分子富集能力上表现更优。此外,GenPack还显著提升了基于AlphaFold2受体结构的分子对接成功率。
为深入理解GenPack提升DrugCLIP和分子对接性能的机制,研究进一步分析了其对口袋构象精修的作用。结果显示,GenPack并未在整体上降低预测结构相对于全配体结构的侧链RMSD,且侧链RMSD与对接或筛选性能之间并不存在显著相关性。这表明,GenPack并非通过改善口袋构象精度发挥作用,侧链准确性在当前设置下对虚拟筛选和对接性能的影响有限,这一结论与既有研究结果一致。进一步分析发现,虚拟筛选性能下降程度与口袋检测精度高度相关,而GenPack能够显著提升预测口袋与真实口袋之间的一致性,从而改善筛选效果。相关分析表明,GenPack主要通过提升口袋定位与定义精度来增强虚拟筛选能力,而非直接优化对接构象。
综合上述结果可以看出,在GenPack的辅助下,DrugCLIP在apo结构和AlphaFold2预测结构上的虚拟筛选性能显著优于传统基于物理信息的方法。除计算评测外,研究还选择了一个此前尚未被系统探索的重要靶点TRIP12,进一步验证DrugCLIP与GenPack的实际应用潜力。TRIP12是一种E3泛素连接酶,与肿瘤和神经退行性疾病密切相关。该蛋白通过介导p14ARF的泛素化降解抑制癌细胞中的p53活性,同时在神经系统中调控葡糖脑苷脂酶的降解,进而影响α-突触核蛋白的积累,这是帕金森病的重要病理特征。尽管具有重要生物学意义,TRIP12在药物发现中仍极具挑战性,目前尚无包含其催化HECT结构域和小分子抑制剂的复合物结构报道,仅有apo结构被解析。
研究基于AlphaFold预测结构中靠近催化位点的结合口袋,对TRIP12开展了虚拟筛选,并选取排名前1%的化合物得到57个候选分子用于实验验证。其中10个化合物在表面等离子共振实验中表现出低于50 μM的解离常数,命中率达到17.5%。两种表现最优的化合物不仅显示出较好的结合亲和力,其对TRIP12泛素化活性的剂量依赖性抑制也在荧光泛素化实验中得到确认,且在最高测试浓度下未观察到对其他泛素通路关键酶的非特异性抑制。尽管其功能抑制效力相对结合亲和力略弱,这可能源于多组分酶反应体系与SPR直接结合测定之间的本质差异,但这些结果仍表明其具备作为先导化合物进一步优化的潜力。
总体而言,计算与实验结果共同表明,DrugCLIP是一种适用于AlphaFold预测蛋白结构的高效虚拟筛选工具,为缺乏实验结构信息的蛋白靶点开展结构基础药物发现提供了一条切实可行的新路径。
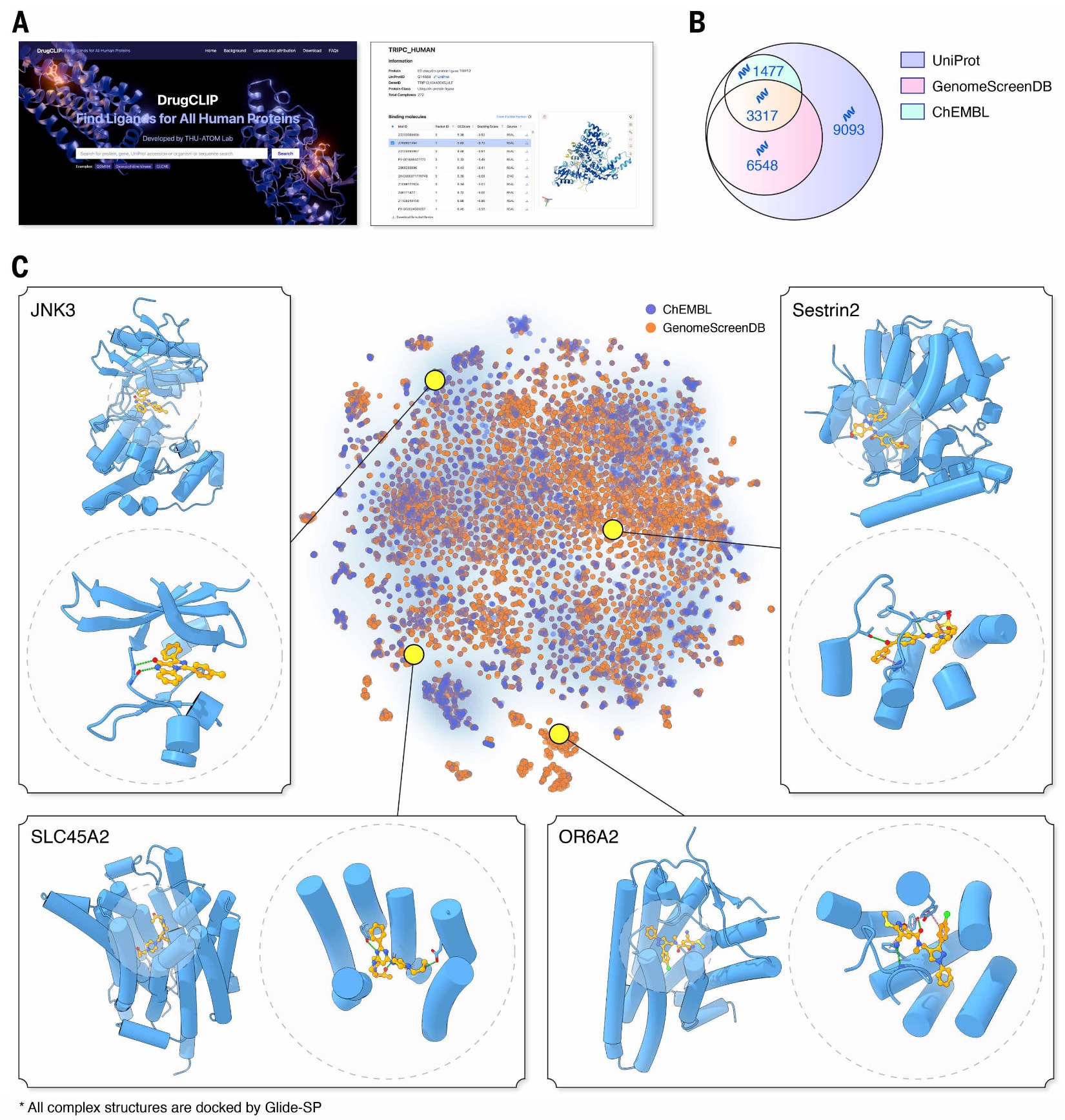
图4|DrugCLIP实现全基因组尺度的虚拟筛选。 (A) 用于访问全基因组虚拟筛选结果的网页界面(https://drugclip.com)。(B) 不同数据库中靶点数量的维恩图,分别以UniProt、DrugCLIP和ChEMBL表示。(C) 全基因组虚拟筛选结果的t-SNE可视化及示例,黄色点及插图表示筛选结果示例,橙色点表示本数据库中的靶点,蓝色点及蓝白渐变区域表示ChEMBL数据库中的靶点,其中颜色由蓝到白表示密度由高到低。
5 基于DrugCLIP的全基因组虚拟筛选
最后,研究构建了一条全基因组虚拟筛选流程,以支持未来的药物发现工作。首先,根据预测的局部距离差检验(plDDT)和预测比对误差(PAE)评分,将所有人类蛋白的AlphaFold预测结构划分为高置信度区域。随后,针对每个区域,结合同源比对方法、Fpocket工具以及GenPack策略来识别潜在的结合口袋。在此基础上,利用DrugCLIP模型对来自ZINC和Enamine REAL数据库的超过5亿个类药小分子进行了筛选。整个筛选过程涉及超过10万亿次蛋白-配体评分计算,在配备8块A100 GPU的单个计算节点上约24小时内完成。
对DrugCLIP筛选得到的高排名分子,研究进一步进行聚类分析,并通过分子对接加以评估,过滤掉Glide评分高于−6 kcal/mol的不良构象。最终构建的GenomeScreenDB数据库包含来自约1万个人类靶点、2万多个结合口袋的200多万个潜在命中分子。所有分子信息、对接评分及构象结果均已在https://drugclip.com公开,以便于后续研究和药物发现流程的开展。同样的自动化后处理流程也被应用于TRIP12靶点。在该靶点的111个候选分子中,有43个进入湿实验测试,最终验证了6个结合物,对应的命中率约为10%。值得注意的是,对TRIP12亲和力最优的结合物E599-0223完全通过自动化流程获得,进一步证明了该筛选管线在生成高质量虚拟筛选数据库方面的有效性。
从覆盖范围上看,该全基因组筛选结果涵盖的靶点数量明显超过了目前最为全面的生物活性分子数据库之一ChEMBL。UniProt中收录了20,436个人类经审校的蛋白编码基因,而最新版本的ChEMBL仅覆盖其中的4,810个靶点,且并非所有靶点都具有高亲和力的小分子配体,部分仅存在肽类或抗体配体,甚至只有质量较低的实验结果。相比之下,GenomeScreenDB覆盖了9,908个靶点,数量超过ChEMBL的两倍,涉及近一半的人类基因组。
为直观比较两者在蛋白空间上的差异,研究使用ESM1b模型对所有蛋白序列进行编码,并通过t-SNE降维可视化。结果显示,GenomeScreenDB覆盖了更广泛的蛋白类型,包括许多与ChEMBL中靶点亲缘关系较远的蛋白,既包含研究充分的经典靶点,也涵盖了同一家族中研究较少的成员以及药理学认知有限的蛋白。例如,JNK3是一个经典的激酶靶点,已具有大量配体结合的晶体结构,DrugCLIP识别的分子被预测可结合其ATP结合口袋,并与铰链区的Met149主链形成氢键。SLC45A2属于溶质转运蛋白超家族,该家族中许多成员是重要药物靶点,但SLC45A2本身鲜有药理学研究。该基因在色素形成中发挥关键作用,并在皮肤黑色素瘤中广泛表达,已有证据提示其具有致癌潜能。数据库中预测的分子均结合于Leu374附近,该位点对蛋白稳定性至关重要,因此可能对SLC45A2功能产生调控作用。
另一个值得关注的例子是OR6A2,其属于嗅觉受体家族。尽管该家族成员主要在嗅觉神经元中表达,但越来越多的证据表明它们也在多种其他组织中表达,具有尚未充分挖掘的药物开发潜力。OR6A2在巨噬细胞中表达,能够感知血液中的辛醛并促进动脉粥样硬化斑块形成。数据库中预测的分子能够很好地匹配其正构结合口袋,有望作为治疗动脉粥样硬化的潜在抑制剂。最后一个例子是Sestrin-2,其属于一个高度保守且可诱导应激反应的蛋白家族,在人类基因组中仅包含三个成员。该蛋白能够感知亮氨酸并促进癌细胞的耐药性。数据库中预测的分子与亮氨酸结合口袋相同,可作为抗癌药物研发的良好起点。
这些实例表明,GenomeScreenDB为探索尚未充分开发的人类基因组提供了重要资源,并为未来的药物发现研究奠定了坚实基础。
6 结论与讨论
快速且高精度的虚拟筛选能够显著加速结构生物学成果向治疗应用的转化,同时也为系统性探索此前缺乏表征的人类靶点提供了可能,从而与结构预测技术的发展形成互补。在该研究中,研究开展了一次全基因组尺度的虚拟筛选工作,在大量尚未充分研究的靶点上预测了数百万个潜在的小分子结合物,并以其中一个代表性案例为例,仅依赖AlphaFold2预测结构完成了命中结果的实验验证。
随着计算结构预测方法的持续进步,这一研究范式有望得到进一步强化。全原子结构预测模型,如AlphaFold3和RoseTTAFold All-Atom,能够对蛋白、核酸及配体复合物进行建模,从而揭示在单体结构中难以识别的结合口袋。此外,这类模型还能够为预测得到的命中分子提供高质量的结合构象。在AlphaFold3架构的基础上,结构-亲和力联合预测模型,如Boltz-2、AuroBind和AlphaRank,可以给出较为准确的亲和力估计,从而辅助候选分子的优先级筛选,并降低对高成本物理模拟方法的依赖。
综合来看,这些技术进展有望扩大可识别的结合口袋数量,提升结合构象预测的准确性,并整体提高虚拟筛选的命中率。将DrugCLIP这类超高速虚拟筛选框架与新一代结构建模和亲和力预测技术相结合,将推动在全人类基因组范围内开展更深入、更系统的药物发现研究。这种技术融合有助于绘制更加精确的可成药基因组图谱,并为未来药物研发的加速奠定坚实基础。