Nat. Mach. Intell. | 负训练数据在增强抗体结合预测稳健性中的重要性
Ta, Wesley, and Jonathan M. Stokes. “The Importance of Negative Training Data for Robust Antibody Binding Prediction.” Nature Machine Intelligence 7, no. 8 (2025): 1192–94. https://doi.org/10.1038/s42256-025-01080-0.
在机器学习模型广泛应用于药物发现和抗体设计的今天,如何构建有效的训练数据集成为模型能否泛化至真实世界的关键之一。Ursu 等人于 Nature Machine Intelligence 发表的工作,聚焦于一个经常被忽视但极其关键的点:负样本的设计方式对模型性能有深远影响。他们提出,精心设计的“困难负样本”不仅能提升模型的泛化能力,还可能帮助模型学会更具生物学意义的规律。
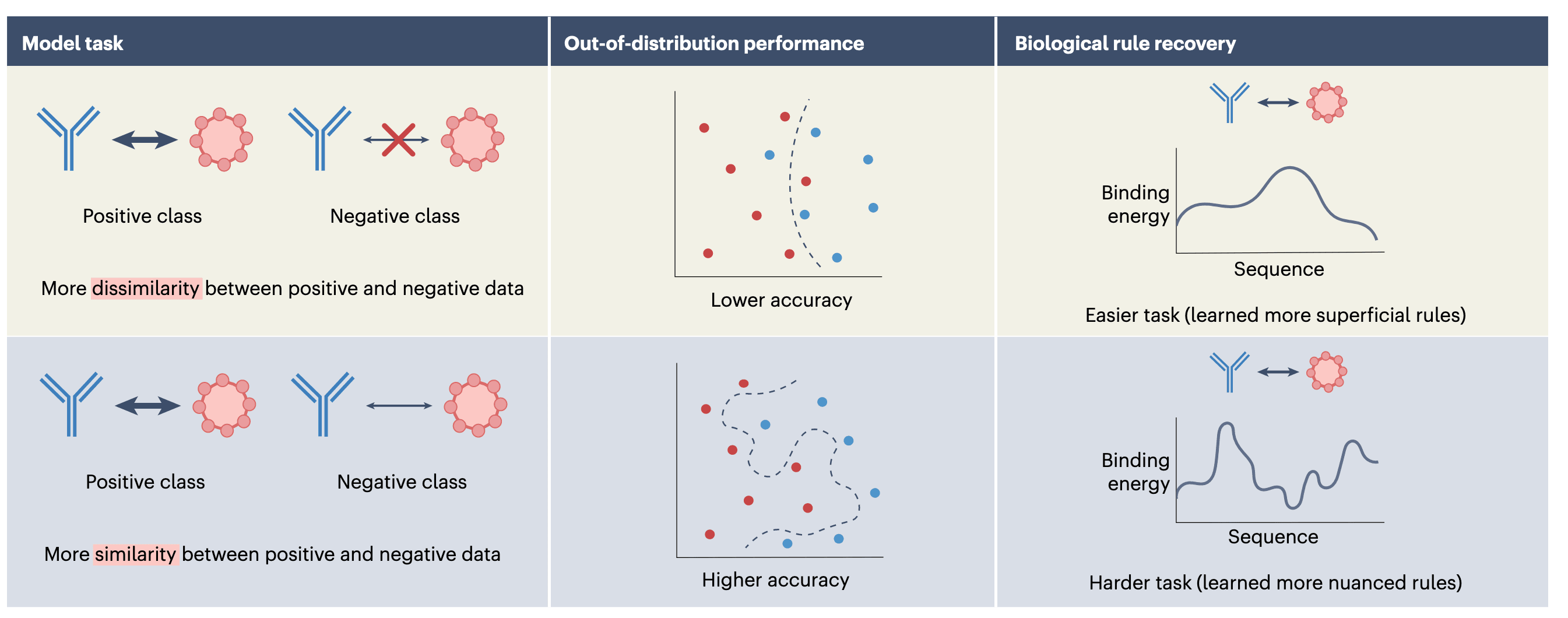
抗体预测任务中,受体与抗原之间的结合能力依赖于复杂的序列特征,尤其是 CDR 区域。本文作者选取抗体中的 CDRH3 结构域 作为研究核心,并利用物理驱动的分子对接模拟器 Absolut! 生成了约 70 万条带有真实结合自由能的抗体–抗原对。在这些数据中,作者挑选出 15,000 条高亲和力的抗体作为正样本,并设计了四类负样本用于建模训练,这四类负样本难度逐级增加,分别是:
- 与目标抗原结合力极低的远离类;
- 仅略低于高亲和力阈值的弱结合类;
- 对另一个抗原具有高亲和力的类;
- 对 9 个不同抗原都具有高亲和力的泛结合类。
每类中有 10,000 条序列用于测试,确保对泛化能力进行充分评估。
为了避免模型复杂性掩盖数据集设计的效果,作者故意选用了结构简单的神经网络架构 SN10,仅由一个 ReLU 激活的隐藏层(10 个神经元)组成。对照模型则是没有隐藏层的逻辑回归模型。结果显示:
- 当模型在同一分布内进行测试时,分类“强结合–远离类”时几乎达到完美(准确率约为 0.99),但在处理“强结合–弱结合”时准确率下降至约 0.92。
- 然而,在分布外测试中,训练于“弱结合负样本”的模型展现出更好的迁移能力,而“远离类”训练的模型则几乎崩溃。
这一发现挑战了监督学习中的常识假设:即在测试集上获得高准确率就意味着模型有良好泛化能力。作者进一步通过 Jensen–Shannon 距离 衡量正负样本间氨基酸分布的相似度,发现:当正负样本越相似(距离越小),模型泛化能力越强。这揭示出一个关键点:困难负样本会促使模型学习更复杂、更接近真实机制的特征。
这不仅体现在性能上,还体现在模型的解释性上。作者采用 DeepLIFT 工具,对模型的每个输入残基分配归因得分,结果发现:
- 在困难任务中训练的模型,其归因图与真实的结合能量更高度相关;
- 而在简单任务上训练的模型,其归因几乎与逻辑回归模型一致,反映出模型仅捕捉到线性、表层特征。
这些结果强烈表明,困难的负样本设计不仅提高泛化能力,更让模型掌握了一定程度的“机制性理解”。这种能力,是目前很多机器学习模型在生物问题中欠缺的重要特性。
作者进一步指出了三点启示:
- 负样本越“困难”,训练越有效,即使牺牲部分训练集上的准确率,也可能带来更强的现实适应性;
- 传统的 held-out test set 并不总能代表真实世界中的模型表现;
- 归因图与物理知识的一致性可以作为快速的“生物直觉检查”。
此外,该研究也促发了对“分布外数据”的反思:模型的表示空间远超人类直觉定义的结构、生化规则所能触及,因此在设计训练目标与任务定义时,也应重新思考模型如何理解并组织信息。
尽管该研究聚焦于抗体预测任务,但其方法与洞见对更广泛的领域同样适用,例如语言模型、蛋白质设计模型和分子属性预测等都面临类似的问题,即:当负样本太简单,模型容易走捷径,难以学到真正有意义的规则。
Ursu 等人清晰地传达了一个深刻的观点:如果希望构建能应对复杂现实世界的模型,就必须在训练阶段给予它们真正有挑战的问题。