Acc. Chem. Res. 2025 | 人工智能驱动的抗菌肽发现:挖掘与生成
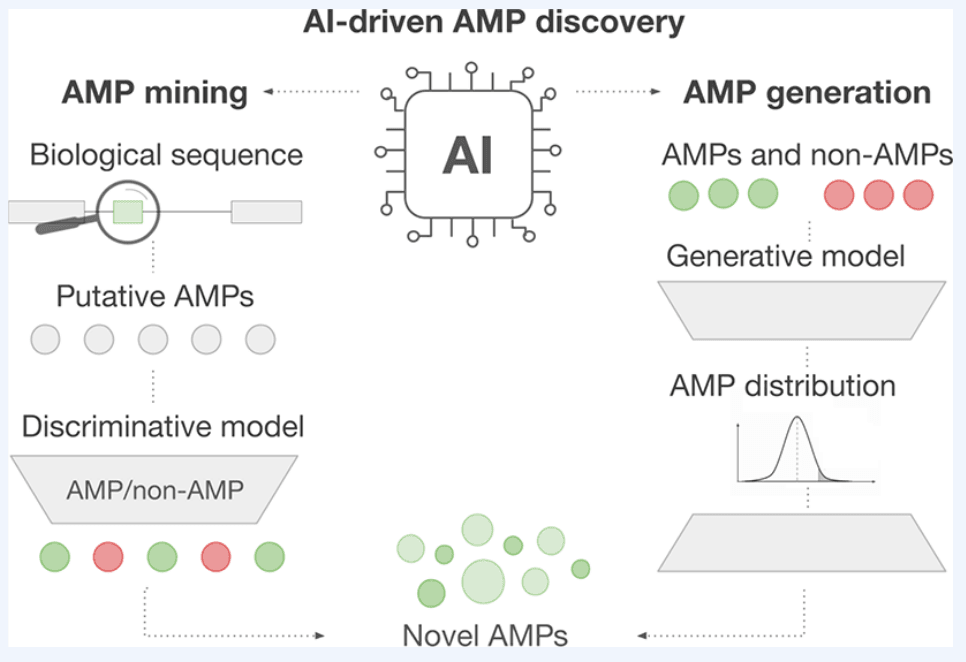
今天介绍一篇发表在Accounts of Chemical Research上的综述文章,主题聚焦于人工智能驱动的抗菌肽(AMPs)发现。抗菌耐药性(AMR)正迅速演变为全球性的健康危机,预计到2050年可能超过癌症,成为主要死因。传统抗生素研发步伐缓慢,与耐药机制的快速扩散之间形成严重脱节。AMPs因具有选择性强、诱导耐药性慢等优势,被认为是新一代抗菌药物的重要候选,但其设计受制于庞大的序列空间以及活性与毒性之间的平衡。
该综述强调了人工智能在AMP研究中的突破性作用:一方面,通过AMP挖掘,AI能够在基因组、蛋白质组及宏基因组数据中高效筛选候选分子;另一方面,借助AMP生成,AI能够利用生成式模型设计出可能优于天然肽的新型分子。文章指出,这些方法不仅显著加速了AMP的发现,还为定制化应对耐药病原体提供了新思路。AI与生物技术的结合,正为抗击AMR危机开辟新的研究前沿。
Szymczak, P.; Zarzecki, W.; Wang, J.; Duan, Y.; Wang, J.; Coelho, L. P.; de la Fuente-Nunez, C.; Szczurek, E. AI-Driven Antimicrobial Peptide Discovery: Mining and Generation. Acc. Chem. Res. 2025, 58 (12), 1831–1846. https://doi.org/10.1021/acs.accounts.0c00594.

0 摘要
抗菌耐药性(AMR)正成为全球严重的健康危机,预计到2050年或将超过癌症成为主要死因。传统抗生素研发难以跟上耐药性的演化,因此亟需新型治疗策略。抗菌肽(AMPs)因其对细菌的选择性及较低的耐药风险,被视为有前景的候选分子,但其设计受限于庞大的序列空间以及活性与毒性之间的平衡。
人工智能(AI)为AMP发现提供了突破。AMP挖掘通过扫描生物序列并结合判别模型预测活性与毒性,已发现多种经实验验证的候选肽;AMP生成利用生成模型从数据中学习,创造具备更高活性与更低毒性的新序列,有望获得优于天然肽的分子。尽管存在生成不现实序列的风险,这些方法显著加速了发现进程。
综上,AI与AMP研究的结合正在开辟下一代抗菌疗法的新前沿,不仅能提升效率,还可能带来具有前所未有性质的分子,为应对AMR危机提供希望。
1 引言
20世纪抗生素发现的繁荣之后,过去三十年来却几乎没有新的抗生素类别问世,而耐药性却在不断加剧。抗菌耐药性已成为严重的全球公共卫生与经济问题,预计到2050年甚至可能超过癌症成为主要死因。抗生素的研发过程极为缓慢,往往需要多年才能筛选出临床前候选分子,这也是抗生素创新不足的根本原因。
一种有前景的策略是设计新型抗菌肽(AMPs)。AMPs通常由10−100个氨基酸组成,具有正电荷(一般为+2到+9)以及较高比例的疏水氨基酸(≥30%)。带正电荷的AMPs能选择性作用于带负电荷的微生物膜,而不易攻击中性的真核细胞膜。除了膜靶向作用外,AMPs还可通过抑制蛋白质或核酸合成、抑制蛋白酶活性或阻断细胞分裂等方式发挥作用。其作用机制由氨基酸组成和结构决定,主要类别包括富含脯氨酸、色氨酸/精氨酸、组氨酸或甘氨酸的肽,多数呈α螺旋构象,但也可能呈β折叠、线性或混合结构。AMPs在哺乳动物、两栖类、昆虫及微生物中普遍存在,参与机体对病原体的防御。重要的是,微生物对AMPs的耐药形成速度通常比对传统抗生素慢。然而,AMPs常表现出对哺乳动物细胞的毒性,通常通过细胞毒性与溶血实验评估。虽然溶解性、稳定性等性质也很关键,但如何设计兼具高活性与低毒性的肽仍是核心挑战。
AMP设计本质上是一个优化问题:在庞大的序列空间中寻找最活跃且毒性最低的肽。以长度25个氨基酸的肽为例,暴力搜索需评估约
人工智能驱动的AMP设计主要包括两种策略:序列挖掘与生成模型。AMP挖掘通过分析基因组与蛋白质组,结合判别模型预测活性和毒性,从而发现可能由自然产生的候选肽,强调现实性。而生成模型则从已有数据学习分布,产生新序列,并通过预测模型优化其性质,从而获得可能优于天然肽的理想化合成肽,尽管可能生成不够现实的序列。这两种策略均能显著加速抗生素发现,识别数十万潜在候选分子。已有AI算法已成功设计并发现部分抗菌肽,其中一些在小鼠模型中表现出疗效。
该综述将介绍最新进展及判别式方法在活性与毒性评估中的关键作用,并系统综述AMP挖掘与生成式AI策略在抗菌肽发现中的应用,最后讨论AMP设计中的挑战与未来研究方向。
1 判别式方法
判别式方法在AMP挖掘与生成中均发挥着核心作用,是筛选具备高活性且低毒性候选分子的关键工具。大多数模型用于区分AMP与非AMP,例如sAMP-pred-GAT、AMPlify与AMPpredMFA等。更为精细的方法则结合最小抑菌浓度(MIC)数据,通过分类或回归来识别高效肽。部分研究还开发了针对特定菌株或物种的判别模型,如AMPMETA或MBC-attention,以筛选对特定微生物具有靶向活性的肽。
尽管由于数据稀缺而研究较少,仍有一些方法尝试预测AMP的毒性,例如EnDL-HemoLyt、AMP-META、Macrel等。然而,目前只有少数判别模型经过微生物学实验验证,而通过溶血实验与细胞毒性实验进行验证的情况则更为罕见。
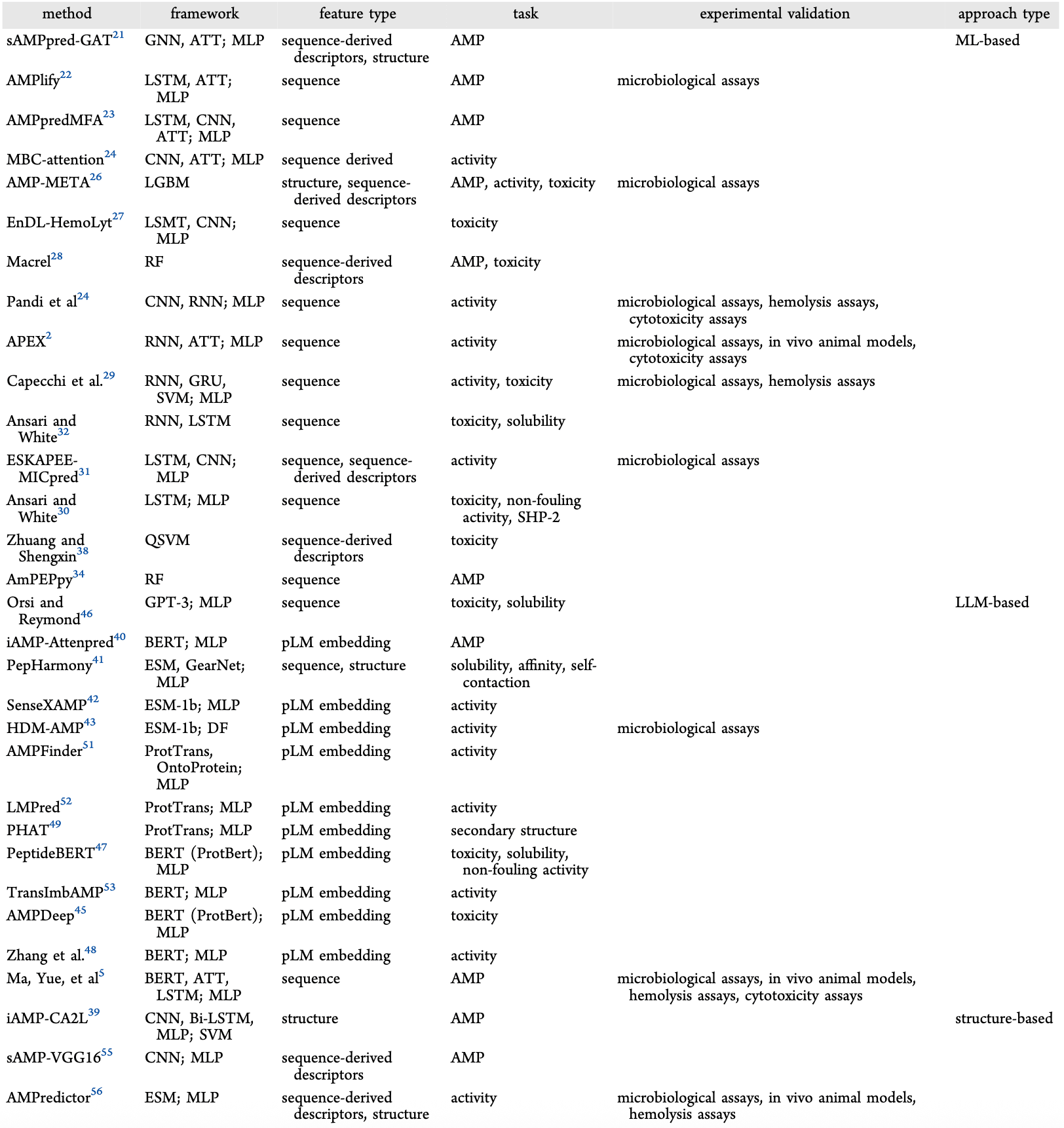
近期判别式方法的整体进展,包括其框架、特征类型、任务设置及实验验证情况,已在表1中进行了总结。
表1 | 判别式方法在抗菌肽发现中的应用
2.1 判别式方法中的模型与架构
传统的机器学习方法,如决策树、支持向量机(SVM)和随机森林(RF),主要依赖序列衍生的描述符来预测AMP。这类方法因其简洁性,不仅能用于分类,还可用于生物学机制解析,例如借助Shapley Additive exPlanations揭示革兰氏阴性菌与阳性菌之间AMP作用机制的差异。一个代表性案例是随机森林模型Macrel,它在训练时使用了AMP与非AMP约1:50的不平衡数据集,更贴近基因组挖掘中的真实分布。Macrel已被成功应用于大规模AMP挖掘研究AMPSphere。尽管相对简单,这些传统方法在某些任务上的表现可与更复杂的深度学习(DL)方法相媲美,甚至更优,因此仍是AMP识别的推荐选择。
相比之下,深度学习模型在应对更复杂问题与提升判别精度方面展现出更大潜力。常见的DL方法借鉴了自然语言处理中的模型,例如循环神经网络(RNN)和长短期记忆网络(LSTM),并在AMP预测中广泛应用。近年来,注意力机制成为关键组件,通过分析序列组成特征(如氨基酸出现频率、前后依赖关系),这些模型能够更深入地理解生物序列的“语义”。例如AMPlify模型结合了自回归BiLSTM与注意力层,从而提升预测效果。
卷积神经网络(CNN)最初用于计算机视觉,但也被引入AMP预测,利用序列衍生特征进行建模。MBC-Attention就是基于CNN的典型模型,它结合多分支CNN与注意力机制,用于回归预测AMP对大肠杆菌的最小抑菌浓度。另一种方法AMPpred-MFA则融合BiLSTM与CNN,并引入多头注意力机制,以捕捉肽序列的上下文依赖关系。
最后,还有探索前沿方法的尝试,例如Zhuang与Shengxin提出的量子支持向量机(QSVM),基于序列特征用于AMP毒性预测,显示出新型计算范式在该领域的潜力。
2.2 判别式方法中的大语言模型应用
尽管RNN、LSTM或CNN等深度学习网络能够捕捉氨基酸间的上下文关系,但基于Transformer架构的大语言模型(LLMs)为分析大规模序列数据提供了全新机遇,尤其是在高效利用注意力机制方面。LLMs已被成功应用于蛋白质序列,发展出所谓的蛋白语言模型(PLMs)。这类模型的训练通常分两步:首先在大规模蛋白质语料上进行生成式预训练,其后针对特定下游任务(如功能、性质或结构预测)进行微调。类似于传统ML方法,PLMs已用于预测抗菌活性与低毒性,也有研究扩展至溶解性或二级结构等性质。
与蛋白相比,肽链更短、三级结构更简单,且已知的生物活性肽数量远少于蛋白,实验验证的AMP数量更是有限。因此,若直接将PLMs应用于AMP而不加额外微调,模型往往会偏向蛋白性质。研究表明,基于蛋白、切分蛋白片段及肽序列分别训练的模型会产生不同的序列嵌入,其中短序列训练的模型往往更具泛化性,并在下游任务中表现更佳。相较之下,直接利用仅在自然语言文本上预训练的LLMs往往效果不佳,其表现甚至不如RNN,因为自然语言嵌入并不适用于肽序列。
最常用的LLM架构是基于Transformer的BERT模型,其优势在于能处理长距离依赖,捕捉序列的全局语境信息。除BERT外,ESM编码器等仅编码架构也被用于AMP分类,结合了序列与进化信息。另一个例子是OntoProtein,它在蛋白序列与基因本体(GO)的基础上开发,被整合进AMPFinder用于预测AMP功能类型。然而,近期评估显示,完整的编码器-解码器架构在性能上优于仅编码模型,这与先前在蛋白质研究中的基准测试结果一致。
除了架构差异,预训练语料的选择对模型表现也有显著影响。多数方法使用UniRef50,部分使用UniRef100,个别使用Pfam、BFD或UniProt,也有融合多语料的案例。事实证明,更加多样化、序列间相似性更低的语料(如UniRef50)能在不改变模型架构的前提下显著提升性能。多数模型在预训练基础上直接添加预测层并进行微调,但也有方法增加了额外的微调阶段,例如利用分泌相关数据提升毒性预测,或用短于50个氨基酸的序列数据引导模型更贴近肽分布。
综上,由于肽的长度与结构显著区别于蛋白,若仅依赖蛋白语料预训练,LLMs可能无法充分代表肽的分布。因此,针对肽类特性的额外预训练与微调是提升AMP判别模型表现的关键。
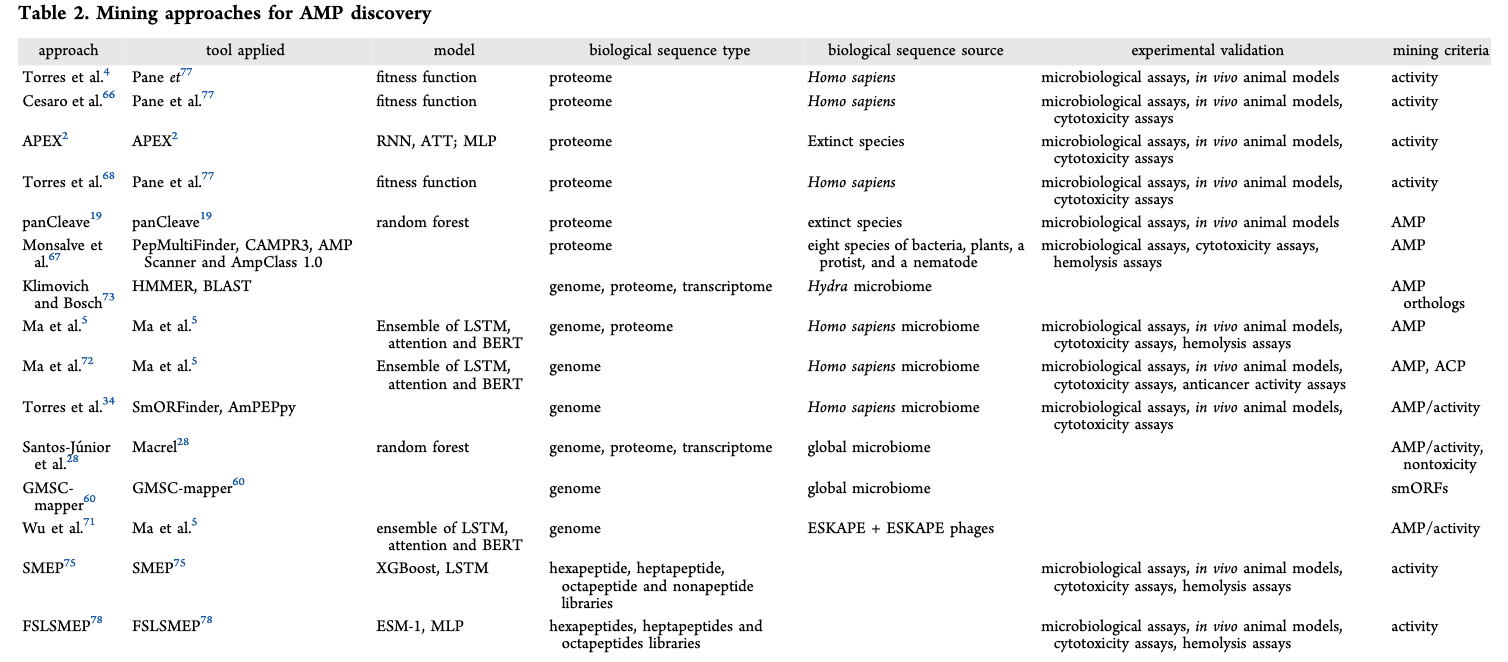
表2 | 抗菌肽发现中的挖掘方法
2.3 判别式方法中的肽表征方式
不同判别模型在输入特征的表征方式上存在显著差异。最常见的表征是氨基酸序列,既可直接作为模型输入,也可用于生成序列衍生的描述符或从预训练模型中提取嵌入。研究表明,基于蛋白语言模型(PLMs)的特征编码优于人工设计的特征。不过,也有研究发现融合方法效果更佳。例如Zhang等人在SenseXAMP模型中将预训练蛋白模型的嵌入与传统蛋白描述符(PD)结合,在AMP预测中表现优于单纯微调预训练模型,这说明传统PD在AMP筛选中依然具有重要价值。
除基于序列的表征外,部分方法将序列转化为图像,例如通过元胞自动机或原子连接信息,再利用CNN进行判别建模。此外,也有方法尝试引入结构信息作为补充视角,尤其是基于图的编码方法。例如,sAMP-pred-GAT整合了结构、序列与进化信息,利用图注意力网络(GAT)识别AMP;AMPredictor则基于图卷积网络(GCN),结合Morgan指纹、肽接触图与ESM嵌入预测MIC值。更进一步,PepHarmony将ESM的序列级嵌入与GearNet的结构级嵌入融合,通过多视角对比学习实现序列与结构信息的协同编码。
这一系列研究表明,在判别式方法中,**多模态表征(序列+结构)**正成为趋势,能更全面捕捉肽的功能特性。
3 AMP 挖掘
近年来生物序列数据的可获得性空前增长,推动了基于挖掘策略的新型抗菌肽(AMPs)发现。AMP挖掘的核心是将前述判别式方法应用于基因组、蛋白质组及宏基因组等生物序列数据。历史上,AMP常被发现于两栖类动物的皮肤分泌物中。虽然AMP挖掘需要谨慎处理以减少假阳性,尤其在面对海量输入数据时,但其预测结果已多次在体外和体内实验中得到验证。目前多数AMP挖掘方法主要关注抗菌活性,而较少涉及毒性预测,可能是由于相关预测模型可靠性不足。
3.1 适合AMP挖掘的生物序列资源
如今已公开数百万个基因组,同时还包括宏基因组(来自微生物群落的多种基因组)和蛋白质组,并广泛存储在公共数据库中。例如,全球微生物基因目录(GMGCv1)源自数千个不同生境的宏基因组,包含数十亿开放阅读框(ORFs),经高度核苷酸一致性聚类后形成数亿个物种水平的单基因。GMGCv1还包括数万个耐药基因,这些基因通过同源性比对综合抗生素耐药数据库(CARD)以及已知耐药基因序列获得。另一项研究中,Duan等人建立了全球微生物小ORF基因目录(GMSC),整合数千个宏基因组和高质量分离株基因组,收录近百万条非冗余smORFs,并开发了GMSC-mapper工具,用于识别和注释微生物(宏)基因组中的小蛋白。
3.2 基因组与蛋白质组的AMP挖掘
近期,人类蛋白质组被探索为新的抗生素来源。Torres等人的研究提出一种基于理化性质的算法(如序列长度、净电荷、平均疏水性),建模AMP活性与理化性质指数函数之间的关系。该算法扫描了42,361条人类蛋白序列,发现2,603个潜在AMP候选,其中许多此前未被视为抗菌因子或宿主免疫相关分子。通过避开已知AMP基序,而侧重理化特征,该方法发现了一批新型抗菌肽,其中部分已被合成、验证,并在动物模型中表现出疗效。
人工智能也推动了更广泛的生物挖掘。例如,对尼安德特人、丹尼索瓦人等灭绝物种的蛋白进行探索,开辟了分子反向复活(molecular de-extinction)的新领域。在相关研究中,作者提出了panCleave随机森林模型用于预测蛋白质组范围的切割位点,并结合包括Macrel在内的六个公开AMP模型进行共识筛选。另一项研究利用深度学习模型APEX对所有可获得的灭绝物种(如猛犸象)的蛋白质组进行挖掘,发现了新型抗菌肽,如neanderthalin-1、mammuthusin-2和elephasin-2,这些分子已成为临床前候选。得益于这些计算方法,新型抗生素的发现速度从过去的数年缩短至数小时。
另一种挖掘思路是针对噬菌体肽聚糖水解酶(PGHs)衍生的抗菌肽。Wu等人提出了一个计算流程,从临床危险病原体ESKAPE(屎肠球菌、金黄色葡萄球菌、肺炎克雷伯菌、鲍曼不动杆菌、铜绿假单胞菌和肠杆菌属)及其噬菌体中挖掘AMP。为评估提取肽的抗菌活性,研究者基于Ma等人的方法,采用CNN与LSTM层构建模型。最终建立了一个名为ESKtides的数据库,收录超过1200万条预测具有高抗菌活性的肽序列。
3.3 微生物组中的AMP挖掘
人类肠道微生物组已成为抗菌肽发现的重要资源。Ma等人利用深度学习技术(LSTM、注意力机制和BERT)在肠道微生物组中挖掘AMP,识别出181条具有抗菌活性的肽,其中多数与已知AMP的序列同源性低于40%。这些肽在体外对耐药革兰氏阴性菌表现出显著抑菌效果,并在小鼠肺部感染模型中有效降低细菌负荷。另一项研究则基于判别式深度学习方法,开展了**抗癌肽(ACP)**预测,利用ACP与AMP间的交集,从肠道宏基因组中高通量筛选出40条候选肽,其中39条在多种癌细胞系中显示出抗癌活性,两条在小鼠肿瘤模型中能显著缩小肿瘤体积且无毒性。
更大规模的计算挖掘分析了63,410个宏基因组与87,920个微生物基因组,结合蛋白质组与转录组数据作为过滤步骤,预测出近百万条潜在AMP,并收录于AMPSphere数据库。另一项研究整合来自人体四个不同部位的宏基因组,聚焦小ORF(smORFs)编码肽,利用随机森林模型预测出323条候选抗菌肽,并在体内外实验中验证了其对临床相关病原体的活性。
近年来,研究者也开始探索非人类微生物组。Klimovich等人通过淡水水螅(Hydra)的转录组与基因组测序结合机器学习分析,发现AMP基因在该物种中经历了快速进化,并在不同细胞类型中选择性表达,且其活性具有空间分布特征:根据微环境的不同,会分泌不同组合的AMPs,以塑造局部的化学景观并调控微生物群落组成。另一项研究则针对蟑螂肠道微生物组,利用包含DenseNet与自注意力模块的深度学习模型,揭示了其独特的抗菌潜力。
3.4 短肽组合序列空间的穷尽式挖掘
除了自然序列资源,近期研究还尝试系统性评估所有短肽的组合空间。Huang等人开发了基于机器学习的流程,从虚拟肽库(由6−9个氨基酸组成)中筛选AMP。该流程通过多步模块化建模进行过滤、分类、排序与效能预测,并基于GRAMPA数据集(MIC测量集合)训练判别器。为避免实验室特异性偏差,研究者采用两步验证策略,在初步实验后对判别模型进行再优化。最终确定了三条六肽,它们在体外对多重耐药菌表现出强效抗菌活性,并在小鼠感染模型中展现出与青霉素相当的疗效且毒性低。另一项研究聚焦鲍曼不动杆菌(临床高危耐药菌),系统性扫描了六肽、七肽和八肽的全组合库,涉及数百亿个候选。研究者基于仅148条序列的极少量数据,采用小样本学习策略(预训练+多次微调),构建了针对鲍曼不动杆菌特异性的AMP分类器,实现了在极度稀缺数据条件下的有效筛选。
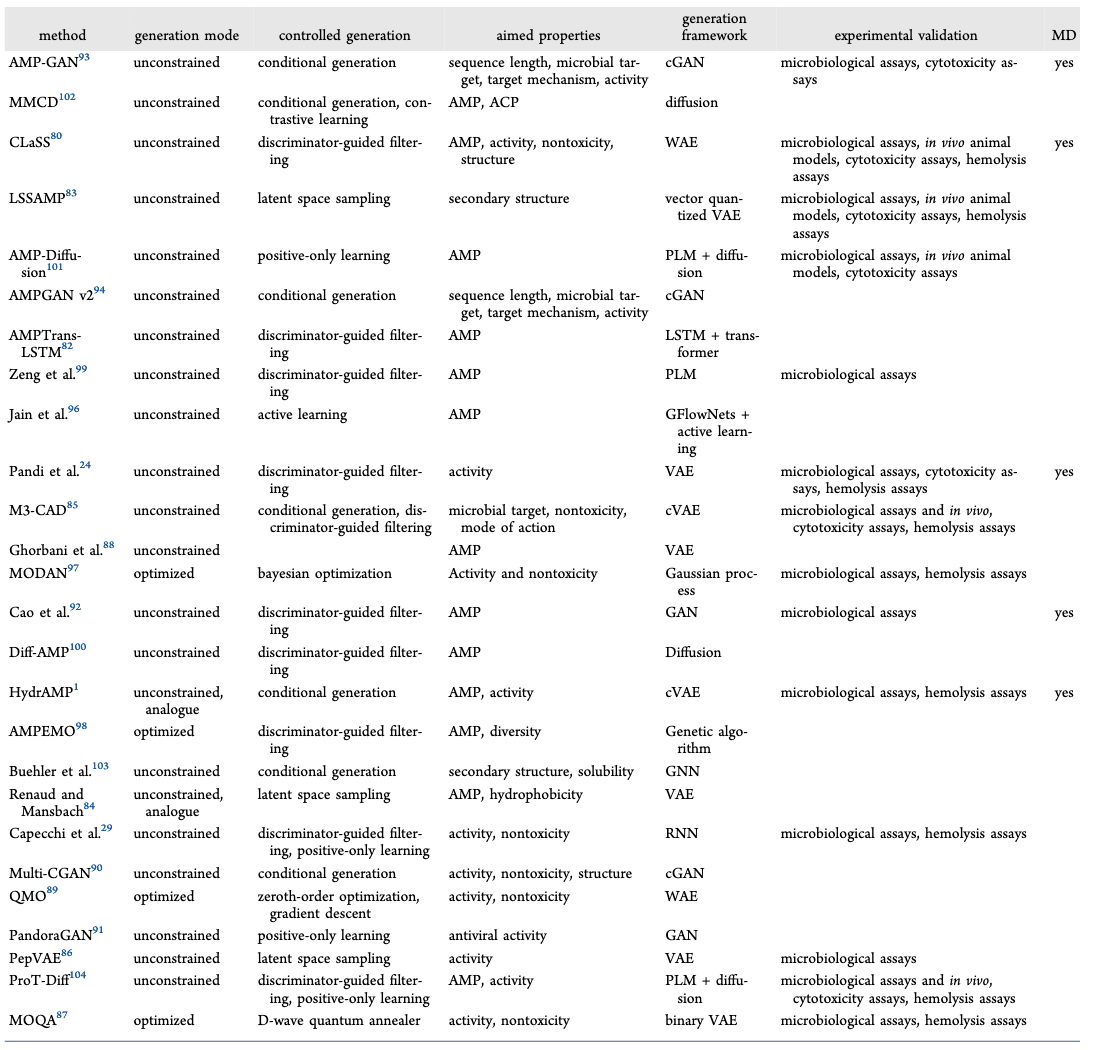
表3 | 抗菌肽发现中的生成方法
4 AMP生成
生成式人工智能为新型药物分子的发现带来了巨大潜力。通过学习和建模底层数据分布,生成模型有望成为未来肽生成的重要工具。目前已有多种生成式AI方法应用于AMP设计,并取得了初步成果,为基于肽的抗菌药物研发奠定了基础。
4.1 MP生成的建模框架
现有方法主要包括自回归模型(如LSTM、RNN)、变分自编码器(VAE)、Wasserstein自编码器(WAE)以及生成对抗网络(GAN)。其中,VAE及其变体应用最为广泛,而GAN在生成新型肽序列方面也展现出强大能力。多数AMP生成方法以发现具备抗菌活性的候选分子为目标,少部分同时兼顾溶血性或细胞毒性特征。尽管已有研究在微生物学层面验证了部分生成的肽,但进入动物模型验证的案例仍较少。
4.2 受控的AMP生成
生成式AI能高效产出成千上万条候选肽,因此需通过受控生成策略提高命中率。一种方法是利用判别模型对潜在空间进行约束与筛选,例如CLaSS模型利用判别器引导WAE生成目标活性与毒性的肽。另一种策略是正样本学习,如PandoraGAN仅使用高活性肽进行训练。除此之外,条件生成模型(cGAN、cVAE)成为重要方向,可在生成阶段控制特定性质。例如Multi-CGAN同时优化多个属性,M3-CAD则能面向八类特征(包括结构、物种特异性抗菌活性、作用机制和毒性)进行多任务生成。
一些方法利用潜在空间采样来选择更可能具备理想性质的区域,例如LSSAMP通过离散化潜在表示结合序列与结构信息,生成具有特定二级结构的肽。HydrAMP则在cVAE框架中引入多项改进:通过条件生成聚焦低MIC值肽,结合预训练分类器确保性质符合预期,同时在损失函数中增加约束,提升稳定性。它还能基于现有肽进行“改造”,由创造力参数控制新变体的多样性。借助分子动力学模拟与分类器集成,研究者筛选出最优候选并进行实验验证,最终发现15条新型高效AMP,其中多条对耐药菌株有效。
在更理想化的肽设计方向上,研究者提出了直接优化生成,通过定制代价函数或优化算法在潜在空间中导航,如QMO采用零阶梯度优化,其他方法包括GFlowNets主动学习、量子退火、贝叶斯优化和进化算法等。
4.3 大语言模型在AMP生成中的应用
随着ChatGPT等工具的成功,生成式语言建模在AMP领域的应用逐渐兴起。与判别任务中广泛应用的预训练语言模型不同,其在AMP生成中的应用仍有限。常见做法包括使用GPT类解码器架构,或基于预训练语言模型的连续嵌入开展扩散生成。然而,这些方法的受控设计能力仍较薄弱,多依赖正样本学习或判别器过滤。一个前景方向是对比学习,例如MMCD方法在扩散模型训练中利用正负样本对比嵌入,从而增强生成效果与控制能力。
5 挑战与未来展望
人工智能的快速发展为新型抗菌肽(AMPs)设计带来了巨大潜力,正在为抗击耐药性提供新的路径。AMP挖掘与AMP生成,在判别式方法的辅助下,成为AI驱动AMP发现的两大重要策略。然而,现有工具仍存在诸多局限,亟待改进,同时也为未来研究开辟了新方向。
5.1 判别模型的挑战
判别模型发展迅速,但在AMP应用中仍面临困难。首先,数据规模有限严重制约了预测方法的开发。迁移学习及基于预训练大语言模型的微调在一定程度上缓解了数据稀缺问题,但仍需更多适应低数据场景的计算方法,以及推动数据共享和实验验证的努力。尤其是针对多重耐药菌株的特异性活性预测,数据不足的问题在可预见的未来仍将存在。其次,AMP毒性预测方法不足,现有的溶血性预测精度有限,而细胞毒性预测几乎缺乏有效模型。更大的挑战在于缺乏经过实验验证的阴性样本,导致监督学习中模型性能受限。此外,不同实验条件下生成的正负样本进一步增加了标签混乱。为解决这些问题,AMP数据库不仅应纳入阳性数据,还需标准化实验条件并补充阴性数据。与此同时,结构信息的利用也亟待加强。目前数据库中的结构信息有限,且多未考虑膜环境或自聚集效应。未来若能结合更多二级、三级结构及翻译后修饰信息,将有助于提升预测性能。最后,现有模型缺乏在外部独立数据集上的客观评估,且对非线性、环肽或化学修饰肽的预测能力不足,而这类分子在临床中尤为重要。因此,需增加相关数据并开展稳定性、半衰期、ADMET等方面的系统实验,为AI驱动的AMP设计提供更坚实的基础。
5.2 AMP挖掘的挑战
生物序列挖掘的优势在于能发现接近天然的候选肽,这些分子易于合成,且常具备宿主选择性。然而其效果依赖于两个前提:其一,目标序列本身需包含高效低毒的AMP;其二,判别方法必须能够准确识别。目前挖掘方法虽已取得成功,但丰富的生物数据集与鲁棒的判别模型仍是必要条件。进一步地,将自然语言处理技术应用于基因功能预测的做法,提示AMP挖掘也可借助类似方法优化对有限标注数据的利用。同时,单一基因组或蛋白质组信息已不足,应尝试整合转录组、核糖体测序等多层数据。此外,多序列比对分析比单序列分析能捕捉更多变异信息,也值得推广。需要指出的是,现有挖掘方法受制于判别模型的局限,难以发现复杂修饰肽,目前仅限于线性肽。尽管部分候选在动物模型中获得验证,但至今尚无挖掘发现的AMP进入临床阶段。
5.3 AMP生成的挑战
与AMP挖掘类似,生成式人工智能在加速AMP发现方面潜力巨大,但仍面临诸多障碍。首先,生成模型的评估与基准测试困难。目前生成肽的评价多集中在多样性、新颖性及与训练数据的相似度,而其真实活性与毒性仅在极少数经实验验证的子集中得到确认。常用的判别器虽能估计这些性质,但选择往往带有任意性,导致模型间难以比较。
其次,生成模型能在极短时间内产生成千上万的候选肽,但缺乏高效排序机制。当前筛选主要依赖大量过滤与专家经验。此外,数据稀缺限制了生成方法的表现,而寻找高效肽本质上是一种分布外生成问题,这是生成建模中的普遍难题。
目前大多数方法仅基于20种标准氨基酸,忽略翻译后修饰与非标准氨基酸,因而低估了肽分子的真正复杂性。若能扩展到环化、磷酸化、脂修饰等典型化学修饰,或纳入非标准氨基酸,未来有望生成更高效的AMP。然而,受限于训练数据不足,生成模型尚无法直接设计临床已用的复杂肽。相对而言,现阶段更现实的路径是通过理性设计优化生成的线性肽,从而提升其稳定性、效力与安全性。
与挖掘方法相比,生成AI获得的AMP在临床前验证中比例更低,这部分归因于多数AI实验室缺乏大规模实验能力。因此,AI发现的AMP尚未进入临床试验,这也意味着未来必须依赖AI、化学与生物实验室及工业界的协作,才能贯通从发现到临床的全流程。
最后,现有生成模型多源于文本与图像生成框架,并不完全适用于肽生成。尤其是受控生成仍需特定建模扩展,这一方向尚未得到深入探索,但对AMP设计至关重要。
6 总结
随着人工智能技术的深入融合,抗菌肽发现正处于一场变革的临界点。自早期研究首次证明机器能够设计出在临床前小鼠模型中有效的肽类抗生素以来,这一领域已取得显著发展与成熟。AI驱动的方法已大幅加速了新型AMP的识别进程。通过利用大规模基因组与蛋白质组数据,并结合先进的生成式与判别式模型,AI将推动高效AMP的设计,尤其是针对新兴耐药病原体的定制化开发。人工智能与生物技术的协同不仅有望加快药物发现流程,还能突破传统方法的局限,为抗菌疗法的未来提供新的希望。